Data.table: fread غير قادر على التعامل مع الحقل المقتبس بشكل خاطئ إذا كان خارج العينة
هذا المثال:
require(data.table)
DT = data.table(A=rep("abc", 10000), B="def")
DT[110, A:='"a"b']
fwrite(DT, f<-tempfile(), quote=F)
fread(f)
ينتج عن رسالة خطأ مضللة:
Expecting 2 cols but row 0 contains only 1 cols (sep=','). Consider fill=true. <<"a"b,def>>
على الأقل لا ينهار (والذي اعتقدت أنه سيعطي أن type[0] يصطدم من CT_STRING إلى نوع غير موجود) ...
ال 3 كومينتر
النهج الممكن:
- أدخل قاعدة اقتباس جديدة QR0 (تصبح جميع القواعد الأخرى QR1..QR4). سيكون هذا هو QR الافتراضي. بموجب هذه القاعدة ، قد يتم اقتباس الحقول أو لا ، ولكن لا يُسمح بعلامات اقتباس داخلية . وبالتالي ، فإن الحقول التالية مقبولة ضمن QR0:
1,foo,"","bar",,"baz,baz"، بينما هذه الحقول غير مقبولة:"foo""bar","foo\"bar",foo"bar,f"oo,bar". - عند قراءة ملف ، إذا كان بعض الحقول من نوع STRING ولا يمكن قراءتها ضمن QR الحالي ، فعندئذٍ:
- إذا كنا حاليًا في QR0 - قم بضرب QR حتى يمكن قراءة الحقل ، ثم استمر في مسح الملف ؛
- بخلاف ذلك ، قم برفع QR ولكن بعد ذلك ارجع وأعد فحص جميع حقول السلسلة (نظرًا لأن معنى علامات الاقتباس قد تغير في البيانات التي تمت قراءتها بالفعل).
- تحتوي مطبات QR على التسلسل الهرمي التالي:
QR0 -> {QR1|QR2|QR3} -> QR4.
أنا أواجه هذا أيضًا ، لكني أتلقى خطأ.
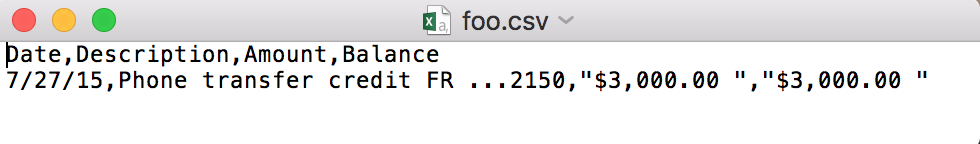
fread("foo.csv", select=c("Date", "Description", "Amount"), header = T) # error
fread("foo.csv", header = T, verbose = F) # works

@ ben519 تحتوي مجموعة البيانات الخاصة بك على صف واحد فقط ، لذا فهي بالتأكيد ليست بسبب مخالفات خارج العينة. لقد قمت بإنشاء مشكلة جديدة لخطأك (انظر الرابط أعلاه)
هل كانت هذه الصفحة مفيدة؟
0 / 5 - 0 التقييمات
التعليق الأكثر فائدة
@ ben519 تحتوي مجموعة البيانات الخاصة بك على صف واحد فقط ، لذا فهي بالتأكيد ليست بسبب مخالفات خارج العينة. لقد قمت بإنشاء مشكلة جديدة لخطأك (انظر الرابط أعلاه)