Scikit-learn: Presisi numerik jarak_ euclidean dengan float32
Deskripsi
Saya perhatikan bahwa fungsi sklearn.metrics.pairwise.pairwise_distances sesuai dengan np.linalg.norm saat menggunakan array np.float64, tetapi tidak setuju saat menggunakan array np.float32. Lihat cuplikan kode di bawah ini.
Langkah / Kode untuk Direproduksi
import numpy as np
import scipy
import sklearn.metrics.pairwise
# create 64-bit vectors a and b that are very similar to each other
a_64 = np.array([61.221637725830078125, 71.60662841796875, -65.7512664794921875], dtype=np.float64)
b_64 = np.array([61.221637725830078125, 71.60894012451171875, -65.72847747802734375], dtype=np.float64)
# create 32-bit versions of a and b
a_32 = a_64.astype(np.float32)
b_32 = b_64.astype(np.float32)
# compute the distance from a to b using numpy, for both 64-bit and 32-bit
dist_64_np = np.array([np.linalg.norm(a_64 - b_64)], dtype=np.float64)
dist_32_np = np.array([np.linalg.norm(a_32 - b_32)], dtype=np.float32)
# compute the distance from a to b using sklearn, for both 64-bit and 32-bit
dist_64_sklearn = sklearn.metrics.pairwise.pairwise_distances([a_64], [b_64])
dist_32_sklearn = sklearn.metrics.pairwise.pairwise_distances([a_32], [b_32])
# note that the 64-bit sklearn results agree exactly with numpy, but the 32-bit results disagree
np.set_printoptions(precision=200)
print(dist_64_np)
print(dist_32_np)
print(dist_64_sklearn)
print(dist_32_sklearn)
hasil yang diharapkan
Saya berharap bahwa hasil dari sklearn.metrics.pairwise.pairwise_distances akan sesuai dengan np.linalg.norm untuk 64-bit dan 32-bit. Dengan kata lain, saya mengharapkan hasil sebagai berikut:
[ 0.0229059506440019884643266578905240749008953571319580078125]
[ 0.02290595136582851409912109375]
[[ 0.0229059506440019884643266578905240749008953571319580078125]]
[[ 0.02290595136582851409912109375]]
Hasil nyata
Cuplikan kode di atas menghasilkan keluaran berikut untuk saya:
[ 0.0229059506440019884643266578905240749008953571319580078125]
[ 0.02290595136582851409912109375]
[[ 0.0229059506440019884643266578905240749008953571319580078125]]
[[ 0.03125]]
Versi
Darwin-16.6.0-x86_64-i386-64bit
('Python', '2.7.11 | 64-bit | (default, Jun 11 2016, 03:41:56) \n[GCC 4.2.1 Compatible Apple LLVM 6.0 (clang-600.0.57)]')
('NumPy', '1.11.3')
('SciPy', '0.19.0')
('Scikit-Learn', '0.18.1')
Semua 102 komentar
Hasil yang sama dengan python 3.5:
Darwin-15.6.0-x86_64-i386-64bit
Python 3.5.1 (v3.5.1:37a07cee5969, Dec 5 2015, 21:12:44)
[GCC 4.2.1 (Apple Inc. build 5666) (dot 3)]
NumPy 1.11.0
SciPy 0.18.1
Scikit-Learn 0.17.1
Itu terjadi hanya dengan jarak euclidean dan dapat direproduksi langsung menggunakan sklearn.metrics.pairwise.euclidean_distances :
import scipy
import sklearn.metrics.pairwise
# create 64-bit vectors a and b that are very similar to each other
a_64 = np.array([61.221637725830078125, 71.60662841796875, -65.7512664794921875], dtype=np.float64)
b_64 = np.array([61.221637725830078125, 71.60894012451171875, -65.72847747802734375], dtype=np.float64)
# create 32-bit versions of a and b
a_32 = a_64.astype(np.float32)
b_32 = b_64.astype(np.float32)
# compute the distance from a to b using sklearn, for both 64-bit and 32-bit
dist_64_sklearn = sklearn.metrics.pairwise.euclidean_distances([a_64], [b_64])
dist_32_sklearn = sklearn.metrics.pairwise.euclidean_distances([a_32], [b_32])
np.set_printoptions(precision=200)
print(dist_64_sklearn)
print(dist_32_sklearn)
Saya tidak dapat melacak lebih lanjut kesalahan tersebut.
Saya harap ini bisa membantu.
numpy mungkin menggunakan akumulator presisi lebih tinggi. ya, tampilannya seperti ini
layak diperbaiki.
Pada 19 Juli 2017 12:05, "nvauquie" [email protected] menulis:
Hasil yang sama dengan python 3.5:
Darwin-15.6.0-x86_64-i386-64bit
Python 3.5.1 (v3.5.1: 37a07cee5969, 5 Desember 2015, 21:12:44)
[GCC 4.2.1 (Apple Inc. build 5666) (titik 3)]
Jumlah 1.11.0
SciPy 0.18.1
Scikit-Learn 0.17.1Itu terjadi hanya dengan jarak euclidean dan dapat direproduksi menggunakan
langsung sklearn.metrics.pairwise.euclidean_distances:impor scipy
impor sklearn.metrics.pairwisemembuat vektor 64-bit a dan b yang sangat mirip satu sama lain
a_64 = np.array ([61.221637725830078125, 71.60662841796875, -65.7512664794921875], dtype = np.float64)
b_64 = np.array ([61.221637725830078125, 71.60894012451171875, -65.72847747802734375], dtype = np.float64)membuat versi 32-bit dari a dan b
a_32 = a_64.astype (np.float32)
b_32 = b_64.astype (np.float32)hitung jarak dari a ke b menggunakan sklearn, untuk 64-bit dan 32-bit
dist_64_sklearn = sklearn.metrics.pairwise.euclidean_distances ([a_64], [b_64])
dist_32_sklearn = sklearn.metrics.pairwise.euclidean_distances ([a_32], [b_32])np.set_printoptions (presisi = 200)
cetak (dist_64_sklearn)
cetak (dist_32_sklearn)Saya tidak dapat melacak lebih lanjut kesalahan tersebut.
Saya harap ini bisa membantu.-
Anda menerima ini karena Anda berlangganan utas ini.
Balas email ini secara langsung, lihat di GitHub
https://github.com/scikit-learn/scikit-learn/issues/9354#issuecomment-316074315 ,
atau nonaktifkan utasnya
https://github.com/notifications/unsubscribe-auth/AAEz65yy8Aq2FcsDAcWHT8qkkdXF_MfPks5sPLu_gaJpZM4OXbpZ
.
Saya ingin mengerjakan ini jika memungkinkan
Lakukan!
Jadi saya pikir masalahnya terletak pada fakta bahwa kami menggunakan sqrt(dot(x, x) - 2 * dot(x, y) + dot(y, y)) untuk menghitung jarak euclidean
Karena jika saya mencoba - (-2 * np.dot(X, Y.T) + (X * X).sum(axis=1) + (Y * Y).sum(axis=1) saya mendapatkan jawaban 0 untuk np.float32, sedangkan saya mendapatkan jawaban yang benar untuk np.float 64.
@jnothman Menurut Anda apa yang harus saya lakukan? Seperti disebutkan dalam komentar saya di atas, masalahnya mungkin menghitung jarak euclidean menggunakan sqrt(dot(x, x) - 2 * dot(x, y) + dot(y, y))
Jadi Anda mengatakan bahwa titik mengembalikan hasil yang kurang tepat daripada produk-lalu-jumlah?
Tidak, yang ingin saya katakan adalah titik mengembalikan hasil yang lebih tepat daripada produk-kemudian-jumlah
-2 * np.dot(X, Y.T) + (X * X).sum(axis=1) + (Y * Y).sum(axis=1) memberikan keluaran [[0.]]
sementara np.sqrt(((X-Y) * (X-Y)).sum(axis=1)) memberikan keluaran [ 0.02290595]
Tidak jelas apa yang Anda lakukan, sebagian karena Anda tidak memposting cuplikan yang berdiri sendiri sepenuhnya.
Dengan cepat melihat posting terakhir Anda, dua hal yang Anda coba bandingkan [[0.]] dan [0.022...] tidak memiliki dimensi yang sama (mungkin masalah salin dan tempel tetapi sekali lagi sulit untuk diketahui karena kami tidak memiliki cuplikan lengkap).
Ok maaf saya buruk
import numpy as np
import scipy
from sklearn.metrics.pairwise import check_pairwise_arrays, row_norms
from sklearn.utils.extmath import safe_sparse_dot
# create 64-bit vectors a and b that are very similar to each other
a_64 = np.array([61.221637725830078125, 71.60662841796875, -65.7512664794921875], dtype=np.float64)
b_64 = np.array([61.221637725830078125, 71.60894012451171875, -65.72847747802734375], dtype=np.float64)
# create 32-bit versions of a and b
X = a_64.astype(np.float32)
Y = b_64.astype(np.float32)
X, Y = check_pairwise_arrays(X, Y)
XX = row_norms(X, squared=True)[:, np.newaxis]
YY = row_norms(Y, squared=True)[np.newaxis, :]
#Euclidean distance computed using product-then-sum
distances = safe_sparse_dot(X, Y.T, dense_output=True)
distances *= -2
distances += XX
distances += YY
print(np.sqrt(distances))
#Euclidean distance computed using (X-Y)^2
print(np.sqrt(row_norms(X-Y, squared=True)[:, np.newaxis]))
KELUARAN
[[ 0.03125]]
[[ 0.02290595136582851409912109375]]
Metode pertama adalah bagaimana cara menghitungnya dengan fungsi jarak euclidean.
Juga untuk memperjelas apa yang saya maksud di atas adalah fakta bahwa sum-then-product memiliki presisi yang lebih rendah bahkan ketika kita menggunakan fungsi numpy untuk melakukannya.
Ya, saya bisa meniru ini. Saya melihat bahwa melakukan pengurangan pada awalnya
memungkinkan ketepatan perbedaan dipertahankan. Melakukan titik
produk dan kemudian mengurangi (atau meniadakan dan menambahkan), seperti yang saat ini kami lakukan,
kehilangan presisi ini karena angka paling signifikan jauh lebih besar dari
perbedaan.
Implementasi saat ini lebih hemat memori untuk sejumlah besar
fitur. Tapi saya kira jarak euclidean menjadi semakin tidak relevan
dalam dimensi tinggi, sehingga memori didominasi oleh jumlah keluaran
nilai-nilai.
Jadi saya memilih untuk mengadopsi implementasi yang lebih stabil secara numerik daripada
d-implementasi efisien asimtotik yang kami miliki saat ini. Sebuah pendapat,
@ogrisel? @agramfort?
Dan ini tentu saja lebih menjadi perhatian karena kami baru-baru ini mengizinkan float32s
menjadi lebih umum di seluruh estimator.
Jadi untuk contoh ini product-then-sum berfungsi dengan baik untuk np.float64, jadi solusi yang mungkin dapat dilakukan adalah dengan mengubah input menjadi float64 kemudian menghitung hasilnya dan mengembalikan hasil yang dikonversi kembali ke float32. Saya kira ini akan lebih efisien, tetapi tidak yakin apakah ini akan berfungsi dengan baik untuk beberapa contoh lain.
mengonversi ke float64 tidak akan lebih efisien dalam penggunaan memori daripada
pengurangan.
Oh ya Anda benar, maaf tentang itu, tetapi saya pikir menggunakan float64 dan kemudian melakukan product-then-sum akan lebih efisien secara komputasi jika tidak dalam hal memori.
Dan alasan menggunakan product-then-sum adalah untuk memiliki efisiensi komputasi yang lebih tinggi dan bukan efisiensi memori.
tentu, tapi saya tidak percaya ada alasan untuk berasumsi bahwa itu sebenarnya
lebih efisien secara komputasi kecuali dengan cara tidak harus mewujudkan
array menengah. Dengan asumsi kita membatasi memori kerja absolut (misalnya oleh
chunking), mengapa mengambil produk titik, norma menggandakan dan mengurangi
jauh lebih efisien daripada mengurangi dan mengkuadratkan?
Berikan tolok ukur?
Ok jadi saya buat script python untuk membandingkan waktu yang dibutuhkan oleh subtraction-then-squaring dan convert ke float64 lalu product-then-sum dan ternyata jika kita memilih X dan Y sebagai vektor yang sangat besar maka 2 hasilnya sangat berbeda . Juga @jnothman Anda benar pengurangan-maka-kuadrat lebih cepat.
Berikut script yang saya tulis, jika ada masalah tolong beri tahu saya
import numpy as np
import scipy
from sklearn.metrics.pairwise import check_pairwise_arrays, row_norms
from sklearn.utils.extmath import safe_sparse_dot
from timeit import default_timer as timer
for i in range(9):
X = np.random.rand(1,3 * (10**i)).astype(np.float32)
Y = np.random.rand(1,3 * (10**i)).astype(np.float32)
X, Y = check_pairwise_arrays(X, Y)
XX = row_norms(X, squared=True)[:, np.newaxis]
YY = row_norms(Y, squared=True)[np.newaxis, :]
#Euclidean distance computed using product-then-sum
distances = safe_sparse_dot(X, Y.T, dense_output=True)
distances *= -2
distances += XX
distances += YY
ans1 = np.sqrt(distances)
start = timer()
ans2 = np.sqrt(row_norms(X-Y, squared=True)[:, np.newaxis])
end = timer()
if ans1 != ans2:
print(end-start)
start = timer()
X = X.astype(np.float64)
Y = Y.astype(np.float64)
X, Y = check_pairwise_arrays(X, Y)
XX = row_norms(X, squared=True)[:, np.newaxis]
YY = row_norms(Y, squared=True)[np.newaxis, :]
distances = safe_sparse_dot(X, Y.T, dense_output=True)
distances *= -2
distances += XX
distances += YY
distances = np.sqrt(distances)
end = timer()
print(end-start)
print('')
if abs(ans2 - distances) > 1e-3:
# np.set_printoptions(precision=200)
print(ans2)
print(np.sqrt(distances))
print(X, Y)
break
ada baiknya menguji bagaimana skala dengan jumlah sampel, bukan hanya
sejumlah fitur ... mengambil norma mungkin mendapat keuntungan dari menghitung beberapa
hal sekali per sampel, tidak sekali per pasang sampel
Pada 20 Oktober 2017 02:39, "Osaid Rehman Nasir" [email protected]
menulis:
Ok jadi saya membuat skrip python untuk membandingkan waktu yang dibutuhkan
pengurangan-kemudian-kuadratkan dan konversi ke float64 lalu perkalian-kemudian-jumlah
dan ternyata jika kita memilih X dan Y sebagai vektor yang sangat besar maka 2
hasilnya sangat berbeda. Juga @jnothman https://github.com/jnothman
Anda benar pengurangan-kemudian-kuadrat lebih cepat.
Berikut script yang saya tulis, jika ada masalah tolong beri tahu sayaimpor numpy sebagai np
impor scipy
dari sklearn.metrics.pairwise import check_pairwise_arrays, row_norms
dari sklearn.utils.extmath impor safe_sparse_dot
dari timeit impor default_timer sebagai timeruntuk saya dalam rentang (9):
X = np.random.rand (1,3 * (10 saya)). Astype (np.float32)Y = np.random.rand (1,3 * (10 saya)). Astype (np.float32)X, Y = check_pairwise_arrays (X, Y)
XX = row_norms (X, squared = True) [:, np.newaxis]
YY = row_norms (Y, squared = True) [np.newaxis,:]#Euclidean distance dihitung menggunakan product-then-sum
jarak = safe_sparse_dot (X, YT, dense_output = True)
jarak * = -2
jarak + = XX
jarak + = YYans1 = np.sqrt (jarak)
mulai = timer ()
ans2 = np.sqrt (row_norms (XY, square = True) [:, np.newaxis])
akhir = timer ()
jika ans1! = ans2:
cetak (akhir-mulai)start = timer() X = X.astype(np.float64) Y = Y.astype(np.float64) X, Y = check_pairwise_arrays(X, Y) XX = row_norms(X, squared=True)[:, np.newaxis] YY = row_norms(Y, squared=True)[np.newaxis, :] distances = safe_sparse_dot(X, Y.T, dense_output=True) distances *= -2 distances += XX distances += YY distances = np.sqrt(distances) end = timer() print(end-start) print('') if abs(ans2 - distances) > 1e-3: # np.set_printoptions(precision=200) print(ans2) print(np.sqrt(distances)) print(X, Y) break-
Anda menerima ini karena Anda disebutkan.
Balas email ini secara langsung, lihat di GitHub
https://github.com/scikit-learn/scikit-learn/issues/9354#issuecomment-337948154 ,
atau nonaktifkan utasnya
https://github.com/notifications/unsubscribe-auth/AAEz6z5o2Ao_7V5-Lflb4HosMrHCeOrVks5st209gaJpZM4OXbpZ
.
ngomong-ngomong, maukah kamu mengirimkan PR, @ragnerok?
ya tentu, apa yang kamu ingin aku lakukan?
menyediakan implementasi yang lebih stabil, juga pengujian yang akan gagal di bawah
implementasi saat ini, dan idealnya menjadi patokan yang menunjukkan kita tidak rugi
banyak dari perubahan, dalam kasus yang wajar.
Saya ingin bertanya apakah mungkin menemukan jarak antara setiap pasangan baris dengan vektorisasi. Saya tidak dapat memikirkan bagaimana melakukannya vektorisasi.
Maksud Anda perbedaan (bukan jarak) antara pasangan baris? Tentu Anda bisa melakukannya jika Anda bekerja dengan array numpy. Jika Anda memiliki array dengan bentuk (n_samples1, n_features) dan (n_samples2, n_features), Anda hanya perlu membentuknya kembali menjadi (n_samples1, 1, n_features) dan (1, n_samples2, n_features) dan lakukan pengurangan:
>>> X = np.random.randint(10, size=(10, 5))
>>> Y = np.random.randint(10, size=(11, 5))
X.reshape(-1, 1, X.shape[1]) - Y.reshape(1, -1, X.shape[1])
Ya terima kasih itu sangat membantu 😄
Saya juga ingin bertanya apakah saya memberikan implementasi yang lebih stabil, saya tidak akan menggunakan X_norm_squared dan Y_norm_squared. Jadi apakah saya juga menghapusnya dari argumen atau haruskah saya memperingatkan tentang hal itu tidak ada gunanya?
Saya pikir mereka tidak akan digunakan lagi, tapi kita mungkin perlu diyakinkan terlebih dahulu
tidak ada kasus di mana kami harus menyimpan versi itu.
kami akan sangat berhati-hati dalam mengubah ini. itu banyak digunakan dan
implementasi lama. kita harus yakin untuk tidak memperlambat apapun yang penting
kasus. kita mungkin perlu melakukan operasi dalam beberapa bagian untuk menghindari memori tinggi
penggunaan (yang mungkin dibuat lebih rumit oleh fakta bahwa ini disebut
dalam fungsi yang dipotong untuk meminimalkan penghentian memori keluaran
jarak berpasangan).
Saya sangat ingin mendengar dari pengembang inti lainnya yang tahu tentang komputasi
biaya dan ketepatan numerik ... @ogrisel , @lesteve , @rth ...
Pada 5 Nov 2017 05:27, "Osaid Rehman Nasir" [email protected]
menulis:
Saya juga ingin bertanya apakah saya memberikan implementasi yang lebih stabil, saya tidak akan melakukannya
menggunakan X_norm_squared dan Y_norm_squared. Jadi, apakah saya menghapusnya dari
argumen juga atau haruskah saya memperingatkan tentang hal itu tidak ada gunanya?-
Anda menerima ini karena Anda disebutkan.
Balas email ini secara langsung, lihat di GitHub
https://github.com/scikit-learn/scikit-learn/issues/9354#issuecomment-341919282 ,
atau nonaktifkan utasnya
https://github.com/notifications/unsubscribe-auth/AAEz63izdpQGDEuW32m8Aob6rrsvV6q-ks5szKyHgaJpZM4OXbpZ
.
tapi akan lebih mudah untuk membahasnya justru jika membuka PR
Ok saya akan membuka PR, dengan implementasi yang sangat dasar dari fungsi ini
Pertanyaannya adalah apa yang harus dilakukan tentang ini untuk rilis 0.20. Mungkinkah ada beberapa perbaikan sederhana / sementara (kejadian dengan biaya misalnya penggunaan memori) yang dapat dipertimbangkan?
Solusi dan analisis yang diusulkan dalam # 11271 pasti sangat berharga, tetapi mungkin memerlukan beberapa diskusi lagi untuk memastikan ini adalah solusi yang optimal. Secara khusus, saya prihatin tentang fakta bahwa sekarang kita memiliki beberapa diskusi tertunda tentang memori kerja global yang optimal di https://github.com/scikit-learn/scikit-learn/issues/11506 tergantung pada jenis CPU dll sementara ini akan menambah tingkat chunking lain dan kompleksitas keseluruhan akan mendapatkan sedikit kontrol IMO. Tapi mungkin itu hanya saya, mencari opini kedua.
Menurut Anda apa yang harus dilakukan tentang masalah ini untuk rilis @jnothman @amueller @ogrisel ?
Stabilitas mengalahkan efisiensi. Masalah stabilitas harus diperbaiki bahkan ketika
efisiensi masih membutuhkan penyesuaian.
Fokus working_memory adalah membuat siluet dengan sampel besar
ukuran bekerja. Ini juga meningkatkan efisiensi, tetapi itu dapat diubah
garis.
Saya sangat yakin kita harus mencoba memperbaiki jarak dengan euclidean
float32 in. Kami memecahkannya di 0.19 dengan asumsi bahwa kami bisa membuatnya
euclidean_distances bekerja pada 32 bit dengan cara yang naif.
Saya setuju bahwa kami membutuhkan perbaikan. Perhatian saya di sini bukanlah efisiensi tetapi kompleksitas tambahan dalam basis kode.
Mengambil langkah mundur, implementasi euclidean scipy tampaknya 10 baris kode C dan untuk 32 bit, cukup lemparkan ke 64bit. Saya mengerti bahwa ini bukan yang tercepat tetapi secara konseptual mudah diikuti dan dipahami. Di scikit-learn, kami menggunakan trik untuk membuat komputasi lebih cepat di BLAS, kemudian ada kemungkinan peningkatan karena di https://github.com/scikit-learn/scikit-learn/pull/10212 dan sekarang kemungkinan solusi potongan untuk euclidean jarak dalam 32 bit.
Saya hanya mencari masukan tentang apa arah umum tentang topik ini seharusnya (misalnya, coba hulu sebagian ke scipy dll).
scipy tampaknya tidak peduli dengan menyalin data ...
Pindah ke 0,21 setelah PR.
Hapus pemblokir?
sqrt(dot(x, x) - 2 * dot(x, y) + dot(y, y))
secara numerik tidak stabil, jika titik (x, x) dan titik (y, y) sama besarnya dengan titik (x, y) karena apa yang dikenal sebagai pembatalan katastropik .
Ini tidak hanya memengaruhi presisi FP32, tetapi tentu saja lebih menonjol, dan akan gagal jauh lebih awal.
Berikut adalah kasus uji sederhana yang menunjukkan seberapa buruk hal ini bahkan dengan presisi ganda:
import numpy
from sklearn.metrics.pairwise import euclidean_distances
a = numpy.array([[100000001, 100000000]])
b = numpy.array([[100000000, 100000001]])
print "skelarn:", euclidean_distances(a, b)[0,0]
print "correct:", numpy.sqrt(numpy.sum((a-b)**2))
a = numpy.array([[10001, 10000]], numpy.float32)
b = numpy.array([[10000, 10001]], numpy.float32)
print "skelarn:", euclidean_distances(a, b)[0,0]
print "correct:", numpy.sqrt(numpy.sum((a-b)**2))
sklearn menghitung jarak 0 di sini kedua kali, bukan akar persegi (2).
Sebuah diskusi tentang masalah numerik untuk varians dan kovarian - dan ini secara sepele dibawa ke pendekatan percepatan jarak euclidean - dapat ditemukan di sini:
Erich Schubert, dan Michael Gertz.
Perhitungan Paralel Stabil Secara Numerik untuk Varians (Co-).
Dalam: Prosiding Konferensi Internasional ke-30 tentang Manajemen Basis Data Ilmiah dan Statistik (SSDBM), Bolzano-Bozen, Italia. 2018, 10: 1–10: 12
Sebenarnya koordinat y bisa dihilangkan dari test case itu, jarak yang benar lalu mudahnya menjadi 1. Saya membuat pull request yang memicu masalah numerik ini:
XA = np.array([[10000]], np.float32)
XB = np.array([[10001]], np.float32)
assert_equal(euclidean_distances(XA, XB)[0,0], 1)
Saya tidak berpikir makalah saya yang disebutkan di atas memberikan solusi untuk masalah ini - cukup hitung jarak Euclidean sebagai sqrt (sum (power ())) dan ini adalah single-pass dan memiliki presisi yang wajar. Kerugiannya adalah menggunakan kuadratnya, yaitu titik (x, x) itu sendiri sudah kehilangan presisi.
@amueller karena masalahnya mungkin lebih parah dari yang diharapkan, saya sarankan untuk menambahkan kembali label pemblokir ...
Terima kasih untuk contoh yang sangat sederhana ini.
Alasan penerapannya seperti ini adalah karena lebih cepat. Lihat di bawah:
x = np.random.random_sample((1000, 1000))
%timeit euclidean_distances(x,x)
20.6 ms ± 452 µs per loop (mean ± std. dev. of 7 runs, 10 loops each)
%timeit cdist(x,x)
695 ms ± 4.06 ms per loop (mean ± std. dev. of 7 runs, 1 loop each)
Meskipun jumlah operasi memiliki urutan yang sama di kedua metode (1,5x lebih banyak pada metode kedua), percepatan berasal dari kemungkinan untuk menggunakan pustaka BLAS yang dioptimalkan dengan baik untuk perkalian matriks matriks.
Ini akan menjadi perlambatan besar bagi beberapa estimator di scikit-learn.
Ya, tapi presisi 3-4 digit dengan FP32, dan 7-8 digit dengan FP64 memang menyebabkan ketidaktepatan yang substansial, bukan? Secara khusus, karena kesalahan seperti itu cenderung memperkuat ...
Saya tidak mengatakan bahwa tidak apa-apa sekarang. :)
Saya mengatakan bahwa kita perlu menemukan solusi di antaranya.
Ada PR (# 11271) yang mengusulkan untuk dilemparkan pada float64 untuk melakukan perhitungan. In tidak memperbaiki masalah float64 tetapi memberikan presisi yang lebih baik untuk float32.
Apakah Anda memiliki contoh di mana menggunakan estimator yang menggunakan jarak_ euclidean memberikan hasil yang salah karena hilangnya presisi?
Saya pasti masih menganggap ini masalah besar dan harus menjadi pemblokir untuk 0,21. Itu adalah masalah yang diperkenalkan untuk 32 bit di 0.19, dan itu bukan urusan yang bagus untuk pergi. Saya berharap kami telah menyelesaikannya lebih awal pada 0,20, dan saya akan baik-baik saja, atau bahkan ingin, untuk melihat # 11271 digabungkan untuk sementara. Satu-satunya masalah dalam PR yang saya ketahui tentang pengoptimalan surround dari efisiensi memori, yang merupakan lubang kelinci yang dalam.
Kami sudah memiliki versi "cepat" ini sejak lama, tetapi selalu dalam float64. Saya tahu, @ kno10 , ada masalah dengan presisi. Apakah Anda memiliki heuristik yang baik dan cepat untuk kami kerjakan saat itu mungkin menjadi masalah dan menggunakan solusi yang lebih lambat tapi pasti?
Ya, tapi presisi 3-4 digit dengan FP32, dan 7-8 digit dengan FP64 memang menyebabkan ketidaktepatan yang substansial, bukan?
Terima kasih telah mengilustrasikan masalah ini dengan contoh yang sangat sederhana!
Namun, menurut saya masalahnya tidak seluas yang Anda sarankan - ini sebagian besar memengaruhi sampel yang jarak mutualnya kecil sehubungan dengan norma mereka.
Gambar di bawah mengilustrasikan hal ini, untuk 2e6 pasangan sampel acak, di mana setiap sampel 1D berada dalam interval [-100, 100]. Kesalahan relatif antara implementasi scikit-learn dan scipy diplot sebagai fungsi jarak antar sampel, dinormalisasi oleh norma L2 mereka, yaitu,
d_norm(A, B) = d(A, B) / sqrt(‖A‖₂*‖B‖₂)
(tidak yakin ini parametrisisasi yang tepat, tetapi hanya untuk mendapatkan hasil yang agak berbeda dengan skala data),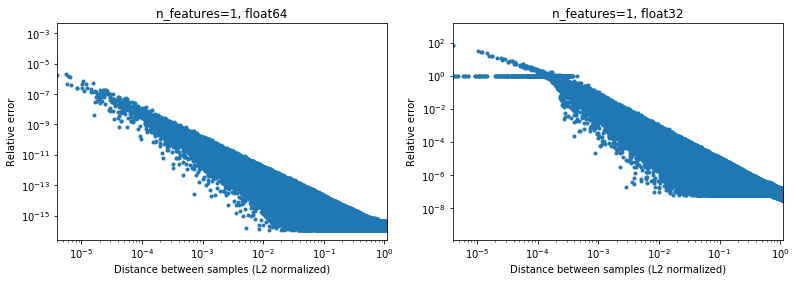
Misalnya,
- jika seseorang mengambil
[10000]dan[10001]jarak normal L2 adalah 1e-4 dan kesalahan relatif pada penghitungan jarak adalah 1e-8 dalam 64 bit, dan> 1 dalam 32 bit (Atau 1e -8 dan> 1 masing-masing dalam nilai absolut). Dalam 32 bit kasus ini memang cukup mengerikan. - di sisi lain untuk
[1]dan[10001], kesalahan relatifnya akan menjadi ~ 1e-7 dalam 32 bit, atau presisi semaksimal mungkin.
Pertanyaannya adalah seberapa sering kasus 1. akan terjadi dalam praktik di aplikasi ML.
Menariknya, jika kita masuk ke 2D, lagi-lagi dengan distribusi acak yang seragam, akan sulit menemukan titik yang sangat dekat,
Tentu saja, pada kenyataannya, data kita tidak akan diambil sampelnya secara seragam, tetapi untuk distribusi apa pun karena kutukan dimensionalitas, jarak antara dua titik mana pun perlahan-lahan akan menyatu ke nilai yang sangat mirip (berbeda dari 0) seiring dengan meningkatnya dimensi. Meskipun ini adalah masalah ML umum, ini dapat mengurangi masalah akurasi ini, bahkan untuk dimensi yang relatif rendah. Di bawah hasil untuk n_features=5 ,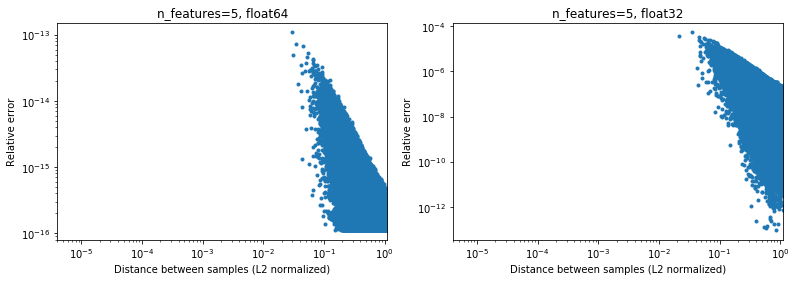 .
.
Jadi untuk data terpusat, setidaknya dalam 64 bit, ini mungkin tidak terlalu menjadi masalah dalam praktiknya (dengan asumsi ada lebih dari 2 fitur). Percepatan komputasi 50x (seperti yang digambarkan di atas) mungkin sepadan (dalam 64 bit). Tentu saja seseorang selalu dapat menambahkan 1e6 ke beberapa data yang dinormalisasi dalam [-1, 1] dan mengatakan bahwa hasilnya tidak akurat, tetapi saya berpendapat bahwa hal yang sama berlaku untuk sejumlah algoritma numerik, dan bekerja dengan data yang diekspresikan dalam ke-6 digit signifikan hanya mencari masalah.
(Kode untuk gambar di atas dapat ditemukan di sini ).
Pendekatan cepat apa pun yang menggunakan versi dot (x, x) + dot (y, y) -2 dot (x, y) kemungkinan akan memiliki masalah yang sama untuk semua yang saya tahu, tetapi Anda sebaiknya bertanya kepada ahli numerik yang sebenarnya untuk ini. ketepatan data
Saya tidak tahu berapa banyak overhead yang Anda dapatkan dari mengubah float32 ke float64 dengan cepat karena menggunakan pendekatan ini. Setidaknya untuk float32, menurut pemahaman saya, melakukan semua perhitungan dan menyimpan produk titik sebagai float64 seharusnya baik-baik saja.
IMHO, peningkatan kinerja (yang tidak eksponensial, hanya faktor konstan) tidak sebanding dengan kerugian dalam presisi (yang dapat menggigit Anda secara tidak terduga) dan cara yang tepat adalah dengan tidak menggunakan trik bermasalah ini. Namun, mungkin juga untuk mengoptimalkan kode lebih lanjut dengan melakukan komputasi "tradisional", misalnya menggunakan AVX. Karena sum ((xy) * 2) sulit untuk diterapkan di AVX.Minimal, saya akan menyarankan untuk mengganti nama metode menjadi approximate_euclidean_distances , karena kadang-kadang presisi rendah (yang semakin buruk semakin dekat dua nilai, yang * mungkin baik-baik saja pada awalnya kemudian mulai menjadi masalah ketika berkumpul ke beberapa optimal) , agar pengguna mengetahui masalah ini.
@rth terima kasih atas ilustrasinya. Tetapi bagaimana jika Anda mencoba untuk mengoptimalkan, misalnya, x menuju beberapa optimal. Kemungkinan besar optimal tidak akan nol (jika itu akan selalu menjadi pusat data Anda, hidup akan menyenangkan), dan akhirnya delta yang Anda hitung untuk gradien dll. Mungkin memiliki beberapa perbedaan yang sangat kecil.
Demikian pula, dalam clustering, cluster tidak akan semuanya memiliki pusatnya mendekati nol, tetapi khususnya dengan banyak cluster, pusat x ≈ dengan beberapa digit sangat mungkin dilakukan.
Namun secara keseluruhan, saya setuju masalah ini perlu diperbaiki. Bagaimanapun kita perlu mendokumentasikan masalah presisi dari implementasi saat ini secepat mungkin.
Secara umum, saya rasa diskusi ini tidak harus terjadi di scikit-learn. Jarak euclidean digunakan dalam berbagai bidang komputasi ilmiah dan milis IMO scipy atau masalah adalah tempat yang lebih baik untuk membahasnya: komunitas itu juga memiliki lebih banyak pengalaman dengan masalah presisi numerik seperti itu. Sebenarnya apa yang kita miliki di sini adalah algoritma yang cepat tapi agak mendekati. Kami mungkin harus menerapkan beberapa solusi perbaikan dalam jangka pendek, tetapi dalam jangka panjang akan lebih baik untuk mengetahui bahwa ini akan berkontribusi di sana.
Untuk 32 bit, https://github.com/scikit-learn/scikit-learn/pull/11271 mungkin memang menjadi solusi, saya tidak begitu tertarik dengan berbagai tingkat penggabungan semua melalui perpustakaan karena itu meningkatkan kompleksitas kode , dan ingin memastikan tidak ada cara yang lebih baik untuk mengatasinya.
Terima kasih atas tanggapan Anda @ kno10! (Komentar saya di atas belum memperhitungkannya) Saya akan membalasnya nanti.
Ya, konvergensi ke beberapa titik di luar asalnya mungkin menjadi masalah.
IMHO, peningkatan kinerja (yang tidak eksponensial, hanya faktor konstan) tidak sebanding dengan kerugian dalam presisi (yang dapat menggigit Anda secara tidak terduga) dan cara yang tepat adalah dengan tidak menggunakan trik bermasalah ini.
Nah, perlambatan> 10x untuk perhitungan mereka dalam 64 bit akan memiliki efek yang sangat nyata pada pengguna.
Namun, mungkin juga untuk mengoptimalkan kode lebih lanjut dengan melakukan komputasi "tradisional", misalnya menggunakan AVX. Karena sum ((xy) ** 2) sulit untuk diterapkan di AVX.
Mencoba implementasi naif cepat dengan numba (yang seharusnya menggunakan SSE),
@numba.jit(nopython=True, fastmath=True)
def pairwise_distance_naive(A, B):
n_samples_a, n_features_a = A.shape
n_samples_b, n_features_b = B.shape
assert n_features_a == n_features_b
distance = np.empty((n_samples_a, n_samples_b), dtype=A.dtype)
for i in range(n_samples_a):
for j in range(n_samples_b):
psum = 0.0
for k in range(n_features_a):
psum += (A[i, k] - B[j, k])**2
distance[i, j] = math.sqrt(psum)
return distance
mendapatkan kecepatan yang mirip dengan scipy cdist sejauh ini (tapi saya bukan ahli numba), dan juga tidak yakin tentang efek fastmath .
menggunakan versi dot (x, x) + dot (y, y) -2 * dot (x, y)
Hanya untuk referensi di masa mendatang, apa yang saat ini kami lakukan kira-kira sebagai berikut (karena ada dimensi yang tidak muncul pada notasi di atas),
def quadratic_pairwise_distance(A, B):
A2 = np.einsum('ij,ij->i', A, A)
B2 = np.einsum('ij,ij->i', B, B)
return np.sqrt(A2[:, None] + B2[None, :] - 2*np.dot(A, B.T))
di mana einsum dan dot sekarang menggunakan BLAS. Saya heran, jika selain menggunakan BLAS, ini juga sebenarnya melakukan jumlah operasi matematika yang sama dengan versi pertama di atas.
Saya heran, jika selain menggunakan BLAS, ini juga sebenarnya melakukan jumlah operasi matematika yang sama dengan versi pertama di atas.
Tidak. ((X - y) * 2.sum ()) tampil* n_samples_x * n_samples_y * n_features * (1 substraksi + 1 penambahan + 1 perkalian)
sedangkan xx + yy -2x.y bekerja
n_samples_x * n_samples_y * n_features * (1 penjumlahan + 1 perkalian) .
Ada rasio 2/3 untuk jumlah operasi antara 2 versi.
Mengikuti pembahasan di atas,
- Membuat PR untuk secara opsional mengizinkan komputasi jarak euclidean persis https://github.com/scikit-learn/scikit-learn/pull/12136
- Beberapa WIP untuk melihat apakah kita dapat mendeteksi dan mengurangi titik bermasalah di https://github.com/scikit-learn/scikit-learn/pull/12142
Untuk 32 bit, kita masih perlu menggabungkan https://github.com/scikit-learn/scikit-learn/pull/11271 dalam beberapa bentuk meskipun IMO, PR di atas agak ortogonal untuk itu.
FYI: ketika memperbaiki beberapa masalah di OPTICS, dan menyegarkan tes untuk menggunakan hasil referensi dari ELKI, ini gagal dengan metric="euclidean" tetapi berhasil dengan metric="minkowski" . Perbedaan numerik cukup besar untuk menyebabkan urutan pemrosesan yang berbeda (hanya dengan menurunkan ambang saja tidak cukup).
Saya benar-benar tidak memahami ini, tetapi saya terkejut tidak ada solusi yang ada. Ini tampaknya merupakan perhitungan yang sangat umum dan sepertinya kami menemukan kembali roda. Adakah yang pernah mencoba menjangkau komunitas komputasi ilmiah yang lebih luas?
Belum, tapi saya setuju kita harus melakukannya. Satu-satunya hal yang saya temukan tentang ini di scipy adalah https://github.com/scipy/scipy/pull/2815 dan masalah terkait.
Saya merasa @jeremiedbb mungkin punya ide?
Sayangnya belum memuaskan :(
Kami ingin mengandalkan pustaka yang sangat dioptimalkan untuk jenis komputasi ini, seperti yang kami lakukan untuk aljabar linier dengan pustaka BLAS seperti OpenBLAS atau MKL. Tapi jarak euclidean bukanlah bagian darinya. Trik titik merupakan upaya melakukan hal tersebut dengan mengandalkan subrutin perkalian matriks-matriks BLAS level 3. Tetapi ini tidak tepat dan tidak ada cara untuk membuatnya lebih tepat menggunakan metode yang sama. Kami harus menurunkan ekspektasi kami baik dalam hal kecepatan atau presisi.
Saya pikir dalam beberapa situasi, presisi penuh tidak wajib dan tetap menggunakan metode cepat tidak masalah. Ini adalah saat jarak digunakan untuk tugas "temukan yang terdekat". Masalah presisi dalam metode cepat muncul ketika jarak antar titik kecil dibandingkan dengan normanya (dalam rasio ~ <1e-4 untuk float 32 dan ~ <1e-8 untuk float64). Pertama agar situasi ini terjadi, kumpulan data harus cukup padat. Kemudian untuk mendapatkan kesalahan pemesanan, Anda harus memiliki dua titik terdekat dengan jarak yang hampir sama. Selain itu, dalam kasus tersebut, dalam sudut pandang ML, keduanya akan menghasilkan kecocokan yang hampir sama.
Dalam situasi di atas, ada sesuatu yang dapat kita lakukan untuk menurunkan frekuensi urutan yang salah ini (turun ke 0?). Dalam situasi argmin jarak berpasangan. Kita bisa memindahkan misordering ke titik-titik yang tidak terdekat. Pada dasarnya menggunakan fakta bahwa salah satu norma tidak diperlukan untuk menemukan argmin, lihat komentar . Ini memiliki 2 keunggulan. Ini lebih kuat (sampai sekarang saya belum menemukan urutan yang salah) dan bahkan lebih cepat karena menghindari beberapa perhitungan.
Satu kekurangannya, masih dalam situasi yang sama, jika pada akhirnya kita menginginkan jarak sebenarnya ke titik terdekat, jarak yang dihitung dengan metode di atas tidak dapat digunakan. Mereka hanya dihitung sebagian dan bagaimanapun juga mereka tidak tepat. Kita perlu menghitung ulang jarak dari setiap titik ke titik terdekatnya. Tapi ini cepat karena untuk setiap titik hanya ada satu jarak untuk dihitung.
Saya ingin tahu apa yang saya jelaskan di atas mencakup semua kasus penggunaan euclidean_distances di sklearn. Tetapi saya menyarankan untuk melakukan itu di mana pun itu dapat diterapkan. Untuk melakukan itu kita dapat menambahkan parameter baru ke euclidean_distances untuk hanya menghitung bagian yang diperlukan untuk menghubungkannya dengan argmin. Kemudian gunakan dalam pairwise_distances_argmin dan pairwise_distances_argmin_min (menghitung ulang jarak min yang sebenarnya di akhir di akhir).
Jika kami tidak dapat melakukannya, kembali ke yang lambat namun tepat, atau tambahkan sakelar seperti di # 12136.
Kami dapat mencoba mengoptimalkannya sedikit untuk menurunkan penurunan kinerja karena saya setuju bahwa ini sepertinya tidak optimal. Saya punya beberapa ide untuk itu.
Kemungkinan lain untuk tetap menggunakan BLAS adalah menggabungkan axpy dengan nrm2 tetapi ini jauh dari optimal. Keduanya adalah fungsi BLAS level 1, dan ini melibatkan salinan. Ini hanya akan lebih cepat dalam dimensi> 100.
Idealnya kami ingin jarak euclidean dimasukkan dalam BLAS ...
Terakhir, ada solusi lain, yaitu upcasting. Ini dilakukan di # 11271 untuk float32. Keuntungannya adalah kecepatannya hanya setengah dari yang sekarang dan presisi tetap terjaga. Namun itu tidak menyelesaikan masalah untuk float64. Mungkin kita bisa menemukan cara untuk melakukan hal serupa di cython untuk float64. Saya tidak tahu persis bagaimana tetapi menggunakan 2 angka float64 untuk mensimulasikan float128. Saya bisa mencobanya untuk melihat apakah itu bisa dilakukan.
Idealnya kami ingin jarak euclidean dimasukkan dalam BLAS ...
Apakah itu sesuatu yang akan dipertimbangkan oleh perpustakaan? Jika OpenBLAS melakukannya, kita akan berada dalam situasi yang cukup baik ...
Juga, apa perbedaan persisnya antara kami melakukannya dan BLAS yang melakukannya? Mendeteksi kapabilitas CPU dan memutuskan implementasi mana yang akan digunakan, atau sesuatu seperti itu? Atau hanya memiliki versi yang dikompilasi untuk arsitektur yang lebih beragam?
Atau hanya lebih banyak waktu / energi yang dihabiskan untuk menulis implementasi yang efisien?
Ini menarik: implementasi alternatif dari metode cepat tidak stabil tetapi mengklaim jauh lebih cepat daripada sklearn:
https://github.com/droyed/eucl_dist
(tidak menyelesaikan masalah ini sama sekali lol)
Diskusi ini tampaknya terkait https://github.com/scipy/scipy/issues/5657
Inilah yang dilakukan julia: https://github.com/JuliaStats/Distances.jl/blob/master/README.md#precision -for-euclidean-and-sqeuclidean
Ini memungkinkan pengaturan ambang presisi untuk memaksa penghitungan ulang.
Menjawab pertanyaan saya sendiri: OpenBLAS memiliki apa yang tampak seperti rakitan tulisan tangan untuk setiap prosesor (bukan arsitektur!) Dan heutistik untuk memilih kernel untuk ukuran masalah yang berbeda. Jadi menurut saya ini bukan masalah memasukkannya ke openblas sebanyak menemukan seseorang untuk menulis / mengoptimalkan semua kernel itu ...
Terima kasih atas pemikiran tambahannya!
Sebagai tanggapan parsial,
Kami ingin mengandalkan pustaka yang sangat dioptimalkan untuk jenis komputasi ini, seperti yang kami lakukan untuk aljabar linier dengan pustaka BLAS seperti OpenBLAS atau MKL.
Ya, saya juga berharap kami bisa melakukan lebih banyak hal ini di BLAS. Terakhir kali saya tidak melihat apa-apa dalam BLAS API standar terlihat cukup dekat (tapi kemudian saya bukan ahli dalam hal itu). BLIS mungkin menawarkan lebih banyak fleksibilitas tetapi karena kami tidak menggunakannya secara default, penggunaannya agak terbatas (meskipun numpy mungkin suatu hari nanti https://github.com/numpy/numpy/issues/7372)
Inilah yang dilakukan julia: Ini memungkinkan pengaturan ambang presisi untuk memaksa penghitungan ulang.
Senang mengetahuinya!
Haruskah kita membuka masalah terpisah untuk perkiraan komputasi yang lebih cepat yang ditautkan di atas? Terlihat menarik
Percepatan mereka pada CPU x2-x4 mungkin disebabkan oleh https://github.com/scikit-learn/scikit-learn/pull/10212 .
Saya lebih suka membuka masalah tentang scipy setelah kami mempelajari pertanyaan ini cukup untuk menghasilkan solusi yang masuk akal di sana (dan kemudian mungkin mendukungnya) karena saya merasa jarak euclidean adalah sesuatu yang cukup mendasar yang seharusnya menarik bagi banyak orang di luar ML (dan pada saat yang sama memiliki pendapat orang-orang di sana misalnya tentang masalah akurasi akan sangat membantu).
Sampai 60x kan?
Ini menarik: implementasi alternatif dari metode cepat tidak stabil tetapi diklaim jauh lebih cepat daripada sklearn
bersenandung tidak yakin tentang itu. Mereka membandingkan %timeit pairwise_distances(a,b, 'sqeuclidean') , yang menggunakan scipy. Mereka harus melakukan %timeit pairwise_distances(a,b, 'euclidean', metric_params={'squared': True}) dan percepatan mereka tidak akan sebaik :)
Seperti yang ditunjukkan jauh sebelumnya dalam pembahasan, sklearn bisa 35x lebih cepat daripada scipy
Ya, tolok ukur mereka hanya ~ 30% lebih baik dengan metric="euclidean" (bukan squeclidean ),
In [1]: from eucl_dist.cpu_dist import dist
... import numpy as np
In [4]: rng = np.random.RandomState(1)
... a = rng.rand(1000, 300)
...b = rng.rand(1000, 300)
In [7]: from sklearn.metrics.pairwise import pairwise_distances
In [8]: %timeit pairwise_distances(a, b, 'sqeuclidean')
214 ms ± 2.06 ms per loop (mean ± std. dev. of 7 runs, 1 loop each)
In [9]: %timeit pairwise_distances(a, b)
27.4 ms ± 2.48 ms per loop (mean ± std. dev. of 7 runs, 10 loops each)
In [10]: from eucl_dist.cpu_dist import dist
In [11]: %timeit dist(a, b, matmul='gemm', method='ext', precision='float32')
20.8 ms ± 330 µs per loop (mean ± std. dev. of 7 runs, 10 loops each)
In [12]: %timeit dist(a, b, matmul='gemm', method='ext', precision='float64')
20.4 ms ± 222 µs per loop (mean ± std. dev. of 7 runs, 10 loops each)
Apakah itu sesuatu yang akan dipertimbangkan oleh perpustakaan? Jika OpenBLAS melakukannya, kita akan berada dalam situasi yang cukup baik ...
Kedengarannya tidak langsung. BLAS adalah sekumpulan spesifikasi untuk rutinitas aljabar linier dan ada beberapa penerapannya. Saya tidak tahu seberapa terbuka mereka untuk menambahkan fitur baru yang tidak ada dalam spesifikasi aslinya. Untuk itu mungkin blis akan lebih terbuka tapi seperti yang dikatakan sebelumnya, ini bukan default untuk saat ini.
Dibuka https://github.com/scikit-learn/scikit-learn/issues/12600 pada sqeuclidean vs euclidean penanganan pairwise_distances .
Saya butuh kejelasan tentang apa yang kami inginkan untuk ini. Apakah kita ingin pairwise_distances menjadi dekat - dalam arti all_close - untuk 'euclidean' dan 'sqeuclidean'?
Agak rumit. Karena x dekat dengan y tidak berarti x² dekat dengan y². Presisi hilang selama kuadrat.
Solusi julia yang ditautkan di atas sangat menarik dan mudah diterapkan. Namun saya menduga itu tidak bekerja seperti yang diharapkan untuk 'sqeuclidean'. Saya menduga Anda harus menyetel ambang batas di bawah ini untuk mendapatkan ketepatan yang diinginkan.
Masalah dengan menetapkan ambang batas yang sangat rendah adalah hal itu menyebabkan banyak penghitungan ulang dan penurunan kinerja yang sangat besar. Namun ini dimitigasi oleh dimensi dataset. Ambang yang sama akan memicu penghitungan ulang yang jauh lebih sedikit dalam dimensi tinggi (jarak lebih besar).
Mungkin kita bisa memiliki 2 implementasi dan beralih tergantung pada dimensi dataset. Yang lambat tapi aman untuk yang berdimensi rendah (bagaimanapun tidak banyak perbedaan antara scipy dan sklearn) dan ambang + cepat satu untuk yang berdimensi tinggi.
Ini akan membutuhkan beberapa tolok ukur untuk menemukan kapan harus beralih, dan mengatur ambang batas tetapi ini mungkin secercah harapan :)
Berikut adalah beberapa tolok ukur untuk perbandingan kecepatan antara scipy dan sklearn. Tolok ukur membandingkan sklearn.metrics.pairwise.euclidean_distances(X,X) dengan scipy.spatial.distance.cdist(X,X) untuk Xs dari semua ukuran. Jumlah sampel berubah dari 2⁴ (16) menjadi 2¹³ (8192), dan jumlah fitur berubah dari 2⁰ (1) menjadi 2¹³ (8192).
Nilai di setiap sel adalah speedup dari sklearn vs scipy, yaitu di bawah 1 sklearn lebih lambat dan di atas 1 sklearn lebih cepat.
Tolok ukur pertama adalah penerapan MKL BLAS dan single core.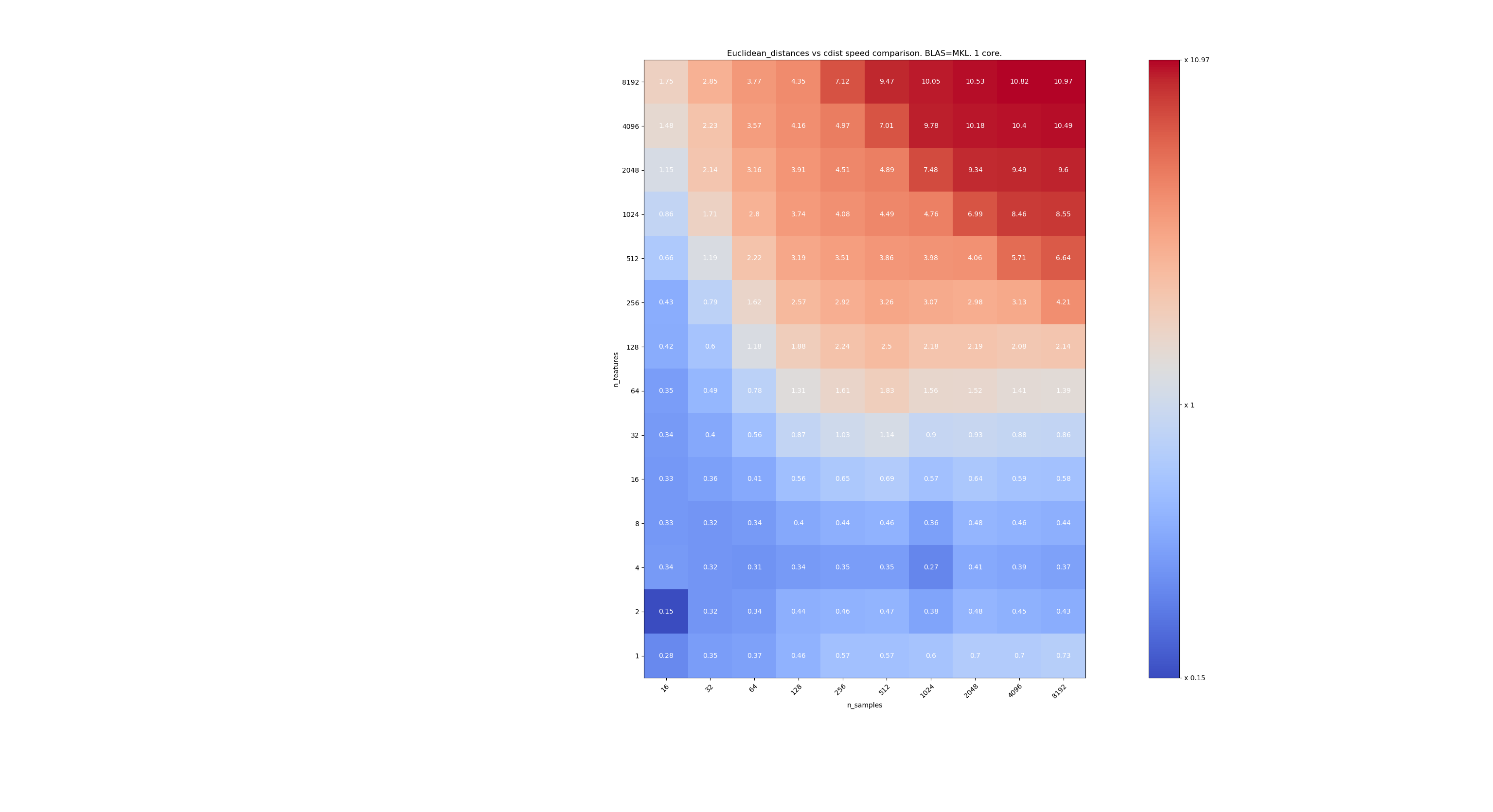
Yang kedua adalah menggunakan implementasi OpenBLAS dari BLAS dan inti tunggal. Ini hanya untuk memeriksa apakah MKL dan OpenBLAS memiliki perilaku yang sama.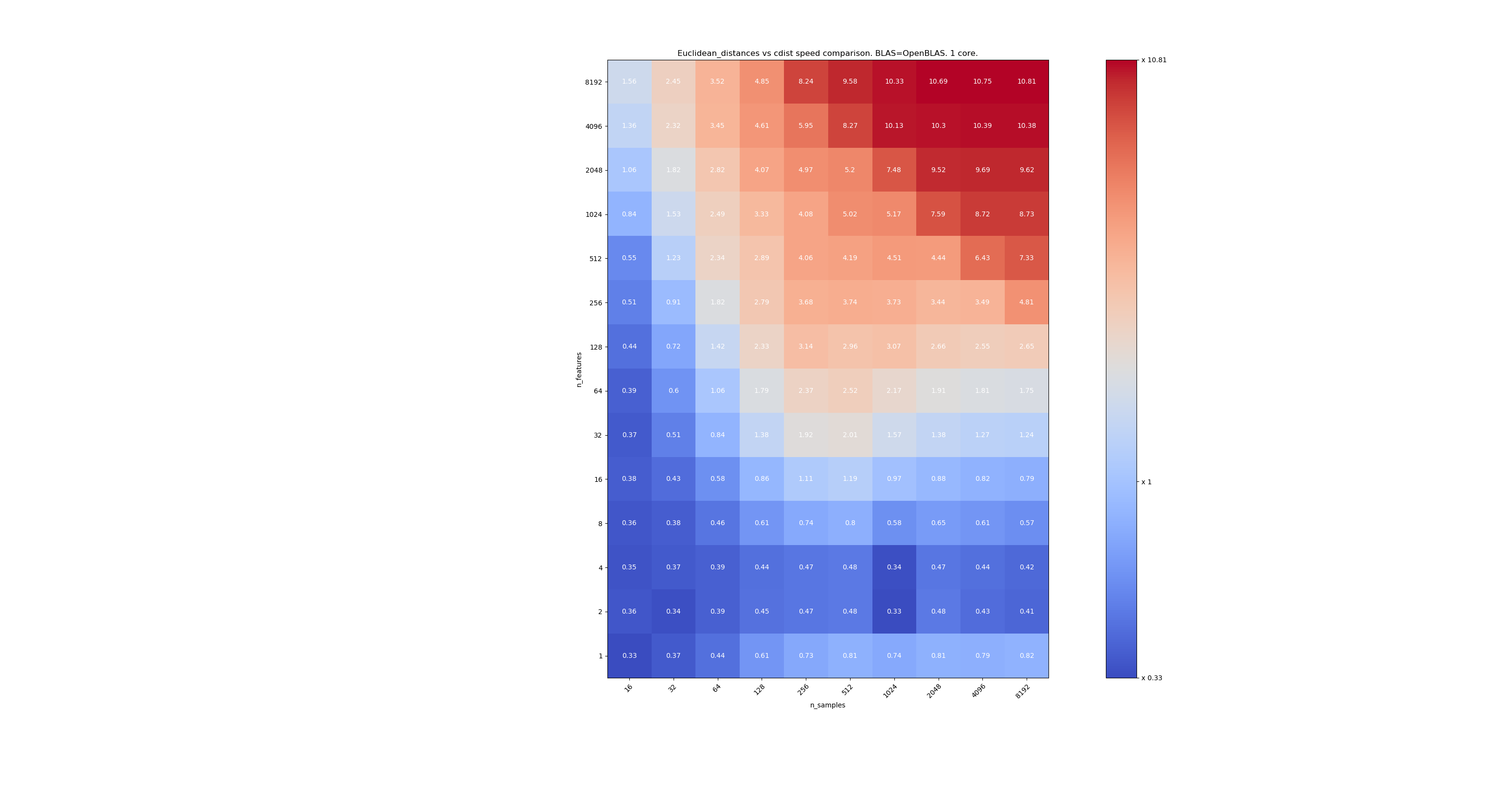
Yang ketiga adalah menggunakan implementasi MKL BLAS dan 4 core. Masalahnya adalah bahwa euclidean_distances diparalelkan melalui fungsi BLAS LEVEL 3 tetapi cdist hanya menggunakan fungsi BLAS LEVEL 1. Menariknya, itu hampir tidak mengubah perbatasan.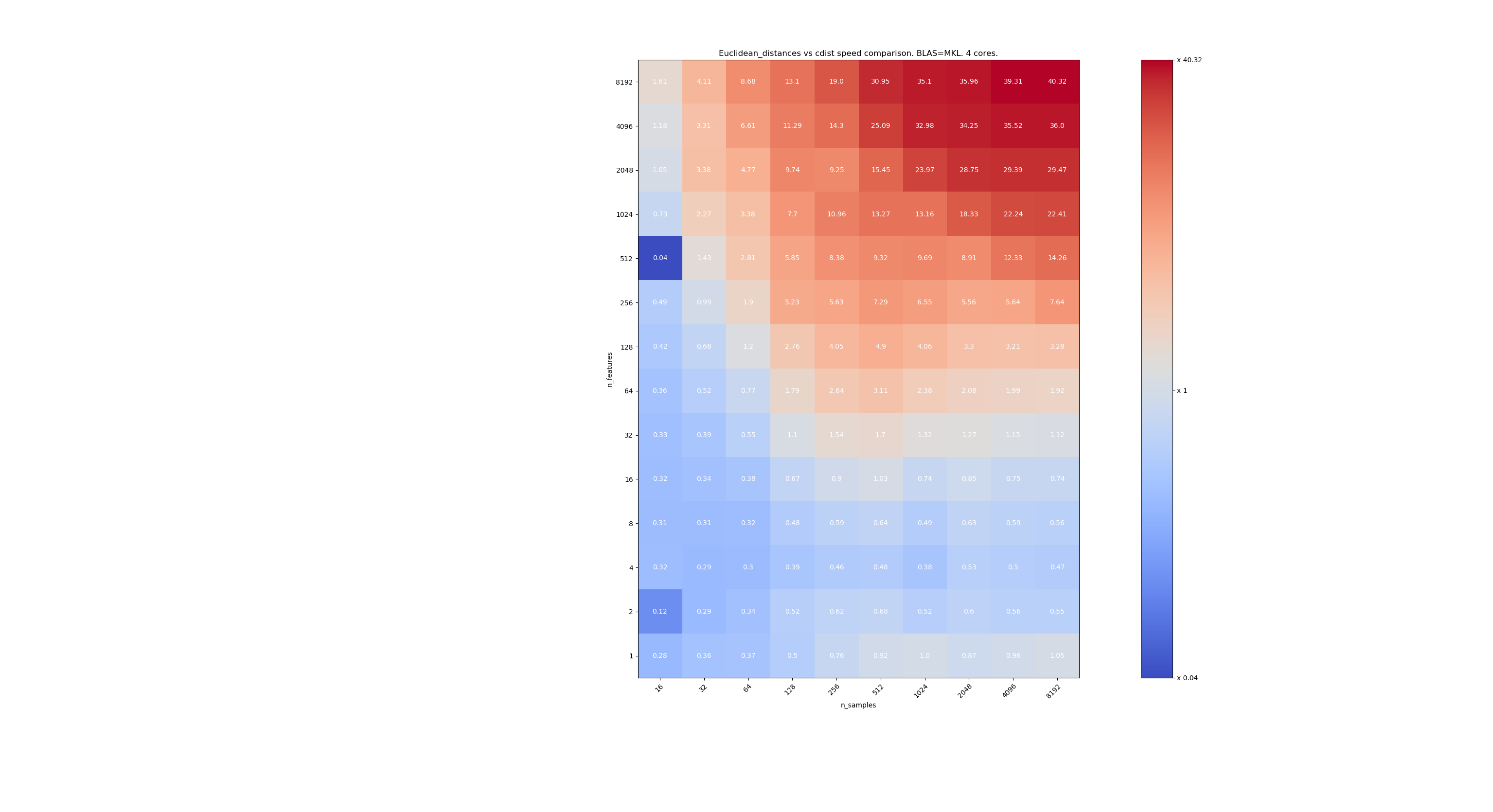
Ketika n_samples tidak terlalu rendah (> 100), nampaknya frontier sekitar 32 fitur. Kami dapat memutuskan untuk menggunakan cdist ketika n_features <32 dan euclidean_distances when n_features> 32. Ini lebih cepat dan tidak ada masalah presisi. Ini juga memiliki keuntungan bahwa ketika n_features kecil, ambang julia menyebabkan banyak penghitungan ulang. Menggunakan cdist menghindari hal itu.
Jika n_features> 32, kita dapat mempertahankan implementasi euclidean_distances , diperbarui dengan ambang julia. Menambahkan ambang seharusnya tidak memperlambat euclidean_distances terlalu banyak karena jumlah fiturnya cukup tinggi sehingga hanya diperlukan beberapa penghitungan ulang.
@jeremiedbb bagus, terima kasih atas analisisnya. Kesimpulannya terdengar seperti cara yang bagus untuk saya.
Oh, saya anggap ini semua untuk float64, bukan? Apa yang kita lakukan dengan float32? selalu marah? upcast untuk> 32 fitur?
Saya belum membaca komentar dengan seksama (akan segera), hanya FYI yang float64 memiliki batasan, lihat # 12128
@ qinhanmin2014 ya, presisi float64 memiliki batasan, tetapi cukup tepat untuk menghasilkan hasil fp32 yang andal untuk semua yang saya tahu. Pertanyaannya adalah di parameter mana upcast ke fp64 sebenarnya lebih murah daripada menggunakan cdist dari scipy.
Seperti yang terlihat pada tolok ukur di atas, bahkan BLAS multi-core umumnya tidak lebih cepat. Hal ini tampaknya sebagian besar berlaku untuk data dimensi tinggi (lebih dari 64 dimensi; sebelum itu manfaatnya biasanya tidak sebanding dengan upaya IMHO) - dan karena jarak Euclidean tidak dapat diandalkan dalam data dimensi tinggi yang padat, kasus penggunaan IMHO tidak terlalu penting . Banyak pengguna akan memiliki kurang dari 10 dimensi. Dalam kasus ini, cdist biasanya lebih cepat?
Oh, saya anggap ini semua untuk float64, bukan?
Sebenarnya itu untuk float32 dan float64 (maksud saya sangat mirip). Saya menyarankan untuk selalu menggunakan cdist ketika n_features <32.
Pertanyaannya adalah di parameter mana upcast ke fp64 sebenarnya lebih murah daripada menggunakan cdist dari scipy.
Upcasting akan melambat dengan faktor ~ 2 jadi saya kira sekitar n_features = 64.
Banyak pengguna akan memiliki kurang dari 10 dimensi.
Tapi tidak semua orang, jadi kita masih perlu mencari solusi untuk data berdimensi tinggi.
Analisis yang sangat bagus @jeremiedbb !
Untuk data berdimensi rendah, sebaiknya gunakan cdist.
Juga, FYI scipy's cdist upcasts float32 menjadi float64 https://github.com/scipy/scipy/issues/8771#issuecomment -384015674, saya tidak yakin apakah ini karena masalah akurasi atau hal lain.
Secara keseluruhan, menurut saya masuk akal untuk menambahkan parameter "algoritme" ke euclidean_distance seperti yang disarankan di https://github.com/scikit-learn/scikit-learn/pull/12601#pullrequestreview -176076355, mungkin dengan default ke "Tidak Ada" sehingga ini juga bisa disetel melalui opsi global seperti di https://github.com/scikit-learn/scikit-learn/pull/12136.
Ada juga pendekatan menarik di Eigen3 untuk menghitung norma yang stabil: https://eigen.tuxfamily.org/dox/StableNorm_8h_source.html (yang belum saya benar-benar grokk)
Penjelasan Yang Baik, Meningkatkan pemahaman saya
Kami belum membuat kemajuan apa pun dalam hal ini di sprint dan kami mungkin harus ... dan @rth tidak ada hari ini.
Saya dapat bergabung dari jarak jauh jika Anda mengatur waktu. Mungkin di awal sore?
Untuk meringkas situasinya,
Untuk masalah presisi dalam penghitungan jarak Euclidean,
- dalam kasus dimensi rendah, seperti yang ditunjukkan @jeremiedbb di atas, kita mungkin harus menggunakan cdist
- dalam kasus dimensi tinggi dan float32, kita dapat memilih antara,
- chunking, menghitung jarak dalam 64 bit dan menggabungkan
- kembali ke cdist jika presisi adalah masalah (bagaimana pertanyaan terbuka - menghubungi misalnya ke scipy mungkin berguna https://github.com/scikit-learn/scikit-learn/issues/9354#issuecomment-438522881 )
Lalu ada semua masalah ketidakkonsistenan antara euclidean, sqeuclidean, minkowski, dll.
Dalam hal ketepatan, @jeremiedbb , @amueller dan saya mengobrol singkat, kebanyakan hanya memerah Jeremie untuk keahliannya. Ia berpendapat bahwa kita tidak perlu terlalu khawatir tentang masalah ketidakstabilan dalam konteks ML dalam dimensi tinggi di float64. Jeremie juga menyiratkan bahwa sulit untuk menemukan tes yang efisien untuk mengetahui apakah hasil yang baik telah dikembalikan (lih. # 12142)
Jadi saya pikir kami senang dengan komentar sebelumnya @rth dengan upcasting untuk float32. Karena cdist juga meng-upcast ke float64, kita dapat menerapkan ulang cdist untuk mengambil float32 (tetapi dengan akumulator float64?), Atau dapat menggunakan chunking, jika kita ingin lebih sedikit menyalin di float32 redup rendah.
Apakah @Celibi ingin mengubah PR di # 11271, atau haruskah orang lain (salah satu dari kita?) Membuat pull request lengkap?
Dan setelah ini diperbaiki, saya pikir kita harus membuat sqeuclidean dan minkowski (p dalam {0,1}) menggunakan implementasi kita. Kami tidak membahas perbedaan dengan Tetangga Terdekat. Sprint lagi :)
Setelah diskusi singkat di sprint, kami berakhir dengan cara berikut:
dalam kasus dimensi tinggi (> 32 atau> 64 pilih yang terbaik): upcast demi potongan menjadi float64 saat float32 dan pertahankan metode 'cepat'. Untuk jenis data ini, masalah numerik, pada float64, hampir dapat diabaikan (saya akan memberikan tolok ukur untuk itu)
dalam kasus dimensi rendah: terapkan komputasi yang aman (daripada menggunakan cdist scipy karena upcast) di sklearn.
(Sangat menggoda untuk melempar float32 ke 0.20.3 juga)
Berikut adalah beberapa tolok ukur untuk perbandingan kecepatan antara scipy dan sklearn.
[... snip ...]
Ini sangat menarik. Saya sebenarnya tidak mengharapkan hasil ini. Saya mengulangi benchmark Anda dan menemukan hasil yang sangat mirip. Kecuali saya akan menganjurkan batas keputusan yang lebih rendah. Tolok ukur saya akan menyarankan 8 fitur.

Kerugian menjadi salah tidak simetris. cdist lebih baik hanya untuk komputasi yang berlangsung kurang dari beberapa detik dan menjadi lambat sangat cepat ketika jumlah fitur meningkat. Jadi, lebih baik gunakan implementasi BLAS jika ragu.
Sunting: Tolok ukur ini adalah untuk float64, tetapi saya juga menemukan bahwa matriks float32 upcasting ke float64 hanya menambah beberapa persen ke total waktu dan tidak mengubah kesimpulan.
Saya perhatikan bahwa ambang batas bergantung pada mesin tempat Anda menjalankan tolok ukur. Saya menduga itu mungkin ada hubungannya dengan instruksi AVX. Saya menyadari tolok ukur yang saya terbitkan dijalankan pada mesin yang tidak memiliki instruksi AVX2, hanya AVX. Dan pada mesin yang memiliki AVX2, saya mendapatkan hasil yang serupa dengan milik Anda.
Tetapi pertanyaannya bukan hanya tentang kinerja tetapi juga tentang presisi dan kemungkinan besar memiliki masalah presisi ketika dimensinya kecil. Mungkin 16 adalah kompromi yang bagus. Bagaimana menurut anda ?
Sehubungan dengan diskusi ini, saya akan mengatakan bahwa kita perlu mengukur keakuratan untuk mengambil keputusan yang tepat.
Namun, terkait dengan PR Anda, keakuratan seharusnya tidak menjadi masalah lagi. Tetapi dengan biaya komputasi yang sedikit lebih mahal. Oleh karena itu, ambang batas mungkin harus ditentukan dengan membandingkan PR Anda.
Akurasi benchmarking tidak semudah itu. Karena kasus-kasus sulit tidak akan merata.
Dan itu mungkin bermasalah jika terjadi tanpa terdeteksi di kasus sudut. Biasanya, Anda ingin memastikan akurasi numerik dalam batas CPU yang terjangkau.
Tetapi seperti yang disebutkan di tempat lain, satu fitur dengan 10000000.01 dan 10000000.00 seharusnya cukup untuk memicu ketidakstabilan numerik dengan fp64 saat menggunakan persamaan bermasalah yang diketahui, 10000 dan 10001 dengan fp32. Dengan 1024 fitur, coba
>>> import sklearn.metrics.pairwise as sk, scipy.spatial.distance as sp
>>> X = [[10000.01] * 1024, [10000.00] * 1024]
>>> print(sk.euclidean_distances(X,X), "\n", sp.cdist(X,X))
[[ 0. 0.31895195]
[ 0.31895195 0. ]]
[[ 0. 0.32]
[ 0.32 0. ]]
(ini menggunakan 0.19.1) Jarak yang benar adalah 0.32.
Seperti yang Anda lihat, ketidakstabilan numerik cenderung menjadi lebih buruk dengan jumlah fitur (kecuali data Anda jarang). Di sini, hasil presisi kurang dari dua digit dengan FP64.
13410 tidak memperbaiki kasus khusus ini. yaitu float64 + dimensi tinggi.
Ini memperbaikinya untuk float32.
Namun kami memutuskan bahwa untuk float64 + high dim, kami tetap seperti itu, karena masalah akurasi sangat tidak mungkin terjadi dan tidak benar-benar berlaku untuk kasus penggunaan machine learning.
Dalam contoh Anda, X [0] dan X [1] memiliki norma sama dengan 320000,32 dan 320000 dan jaraknya 0,32, yaitu 1e-6 kali norma mereka. Dalam pembelajaran mesin, 16 digit signifikan (dalam float64) tidak semuanya relevan.
Namun kami memutuskan bahwa untuk float64 + high dim, kami tetap seperti itu, karena masalah akurasi sangat tidak mungkin terjadi dan tidak benar-benar berlaku untuk kasus penggunaan machine learning.
Saya akan lebih moderat dalam hal ini. Mengurangi dimensi adalah langkah pertama yang biasa dilakukan di ML. MDS dapat digunakan untuk itu, dan itu membuat penggunaan matriks jarak euclidean sangat banyak.
Jika seseorang ingin melihat peningkatan akurasi kasus float64, ada cara menggunakan dua float untuk mewakili hasil antara. Meskipun saya pikir itu mulai berada di luar cakupan scikit-learn.
ftp://ftp.math.ethz.ch/users/wpp/CELL/qd.pdf
Saya tidak jelas. Saya tidak mengatakan data dimensi tinggi tidak berlaku untuk pembelajaran mesin. Saya mengatakan bahwa jenis masalah presisi yang terjadi di float64 melibatkan titik-titik yang jaraknya 6 kali lipat lebih kecil dari normalnya. Memiliki presisi seperti itu tidak ada artinya dalam model pembelajaran mesin yang realistis
Dalam pembelajaran mesin, 16 digit signifikan (dalam float64) tidak semuanya relevan.
Saya sama sekali tidak yakin bahwa ini secara umum benar.
Dalam contoh ini, kami kehilangan presisi 15 dari 16 digit. Saya setuju jika kami akan menggunakan setengah dari presisi, tetapi kami tidak memiliki hubungan seperti itu. Kerugian dari downcasting FP64 ke FP32 seringkali dapat ditoleransi karena ketepatan pengukuran. Dan GPU tingkat konsumen jauh lebih cepat dengan FP32 daripada dengan FP64, misalnya (dalam beberapa kasus, mereka mengizinkan data FP32 dan akumulator FP64 sekarang), dan untuk inferensi jaringan saraf, Anda bahkan dapat melihat int8 sekarang. Tapi itu tidak berlaku di mana-mana.
Misalnya di k-means clustering, ada asumsi bahwa kelompok berbeda secara substansial dalam cara mereka (dan bahwa kita tidak tahu cara sebelumnya), dan karenanya kita memiliki kerugian dalam presisi sini. Jika Anda memiliki banyak kelompok, beberapa norma mereka dapat menjadi besar dibandingkan dengan pemisahan mereka.
Selanjutnya, setelah iterasi awal pertama, seringkali perbedaan jarak yang kecil membuat satu titik beralih ke cluster lain. Kehilangan presisi di sini dapat memengaruhi hasil, dan dapat menyebabkan ketidakstabilan.
Sekarang pertimbangkan k-means pada fragmen deret waktu dengan banyak variabel.
Dengan bertambahnya ukuran data, kita harus mengasumsikan bahwa jarak ke tetangga terdekat semakin kecil, dan kecuali norma Anda 0, pada akhirnya akan lebih kecil dari norma vektor dan menyebabkan masalah. Jadi, ini kemungkinan akan menjadi lebih parah dengan meningkatnya ukuran kumpulan data. Kutukan dimensi mengatakan bahwa jarak terbesar dan jarak terkecil semakin mirip; jadi untuk menghitung peringkat tetangga terdekat yang benar, kita mungkin membutuhkan ketepatan yang baik dalam data berdimensi tinggi. Pada kumpulan data 20news, jarak bukan nol terkecil adalah sekitar 0,02 (semua normanya 1). Tapi itu baru 10k contoh, dan isinya cukup beragam. Sekarang asumsikan kumpulan data tentang deteksi hampir duplikat sebagai gantinya ...
Saya tidak yakin ini "tidak mungkin" terjadi di ML ... tentu saja itu tidak akan mempengaruhi semua orang.
Saat saya mengatakan "Dalam pembelajaran mesin, 16 digit signifikan (dalam float64) tidak semuanya relevan.", Saya tidak berbicara tentang jarak yang dihitung, saya berbicara tentang data X.
Dalam pembelajaran mesin, data Anda berasal dari suatu ukuran, dan tidak ada pengukuran yang tepat sampai digit ke-9 (selain sangat sedikit dalam fisika partikel).
Jadi dalam contoh Anda 10000000.01 dan 10000000.00 , bagaimana Anda akan mementingkan jarak 0,01 ketika ketidakpastian Anda pada nilai X jauh lebih besar?
Untuk KMeans, pertama-tama ada cara untuk mengatasi sebagian besar kehilangan presisi. Saat Anda mencari pusat terdekat dari sebuah observasi x, Anda tidak perlu menambahkan norma x ke penghitungan jarak yang menghindari pembatalan katastropik dalam banyak kasus.
Kemudian, klaster kmean berdasarkan jarak euclidean. Tetapi Anda tidak tahu apakah ini cara yang tepat untuk mengumpulkan data Anda. Faktanya, ada 0 kemungkinan bahwa data Anda dikelompokkan seperti itu. Kmeans memberikan perkiraan tentang bagaimana data Anda dapat dikelompokkan dan titik-titik yang berada di perbatasan 2 cluster pasti tidak dapat dianggap sebagai milik satu atau lainnya secara pasti. Apa interpretasi Anda tentang suatu titik pada jarak yang sama dari 2 cluster? Milik saya adalah 2 cluster yang seharusnya hanya satu cluster atau KMeans bukan yang terbaik untuk mengelompokkan data saya (atau bahkan kmean memberi saya gambaran yang cukup bagus tentang bagaimana data saya dikelompokkan tetapi saya tahu bahwa batas cluster tidak relevan).
Penggunaan hanya "| b | ^ 2-2ab" tidak memiliki pembatalan bencana - tetapi hilangnya presisi yang sama dalam digit yang membuat perbedaan. Hasilnya sama seperti jika Anda menambahkan norma a ke setiap jarak sesudahnya; jika jarak jauh lebih kecil dari norma a, maka Anda mendapatkan kerugian dalam presisi yang dapat dihindari dengan melakukan perhitungan dengan cara tradisional tanpa hack BLAS.
Jadi Anda sebenarnya TIDAK bisa mengatasi masalah numerik dengan cara ini!
K-means adalah masalah pengoptimalan. Jadi peretasan semacam itu dapat berarti bahwa sklearn hanya menemukan solusi yang lebih buruk daripada alat lain. Dan seperti yang ditunjukkan sebelumnya, ini juga dapat menyebabkan ketidakstabilan. Dalam kasus terburuk, ini dapat menyebabkan sklearn kmeans beralih melalui status yang sama hingga max_iter tanpa perbaikan (dengan asumsi tol = 0, jika Anda ingin menemukan optimal lokal), yang menurut teori tidak mungkin dilakukan.
Sampai k-means bertemu, Anda tidak dapat banyak bicara tentang titik-titik dengan jarak "sama" ke dua cluster. Iterasi berikutnya, sarana mungkin telah berpindah dan perbedaannya bisa menjadi jauh lebih besar dan materi!
Saya bukan penggemar berat k-means karena tidak berfungsi dengan baik pada data yang berisik. Tetapi ada variasi yang menangani kasus seperti itu dengan lebih baik. Namun demikian, jika Anda menggunakannya, Anda mungkin harus mencoba untuk mendapatkan kualitas penuh (itulah sebabnya saya juga selalu menggunakan tol=0 ) dan tidak membuatnya lebih buruk dari yang diperlukan. Ini cukup murah untuk melakukan penghitungan yang tepat (dan, seperti yang disebutkan, masalah menjadi lebih buruk dengan ukuran data - jadi untuk data kecil, runtime yang lebih lambat biasanya tidak menjadi masalah, untuk kumpulan data yang lebih besar, presisi menjadi lebih penting).
Bergantung pada aplikasinya, perbedaan antara 10000000.01 dan 10000000.00 dapat berpengaruh. Dan seperti yang saya tunjukkan sebelumnya, jika Anda menggunakan banyak fitur, masalah muncul lebih awal. Dengan fp32 minimal 10000 dan 10001 dengan satu fitur dan 100 vs. 101 dengan 100 fitur, saya kira:
Seperti disebutkan, mean mungkin memiliki makna fisik yang tidak ingin Anda hilangkan. Jika Anda memiliki data dengan suhu dalam Kelvin, Anda tidak ingin 0: 1 menskalakannya atau memusatkannya; itu akan merusak skala rasio Anda. Sekarang jika Anda ingin membandingkan, misalnya, deret waktu suhu beberapa produk baja saat mendingin, dan mencari tahu apakah proses pendinginan memengaruhi keandalan produk baja Anda. Anda mungkin memiliki suhu lebih dari 700 K, dan deret waktu mungkin memiliki ratusan titik data jika Anda ingin menganalisis proses cooldown. Bahkan dengan hanya 5 digit presisi input (0,01K) dengan panjang deret waktu masalah numerik dapat terjadi. Anda mungkin akan mendapatkan hanya 1-2 digit lagi. Saya tidak berpikir Anda bisa mengesampingkan bahwa presisi penting dalam ML jika Anda memiliki jenis efek bencana ini. Ini berbeda jika Anda bisa menjamin untuk selalu mendapatkan, katakanlah 10 dari 16 digit dengan presisi. Di sini Anda tidak dapat melakukan itu, Anda mungkin memiliki 0 digit tepat dalam kasus terburuk (itulah mengapa itu bencana).
Dalam pembelajaran mesin, data Anda berasal dari suatu ukuran, dan tidak ada pengukuran yang tepat sampai digit ke-9 (selain sangat sedikit dalam fisika partikel).
Nilai mentah dari dunia nyata jarang memiliki akurasi seperti itu, itu benar. Tetapi ML tidak terbatas pada jenis masukan tersebut. Seseorang mungkin ingin menerapkan ML ke masalah matematika, seperti menerapkan MDS pada grafik puzzle mirip kubus rubik atau mengelompokkan strategi sukses yang ditemukan oleh sekumpulan agen RL yang bermain pacman.
Meskipun sumber informasi awal adalah dunia nyata, mungkin ada beberapa pemrosesan tengah jalan yang membuat sebagian besar digit relevan dengan algoritme pengelompokan. Seperti hasil dari penurunan gradien pada fungsi yang parameternya diambil sampelnya secara statistik di dunia nyata.
Saya sebenarnya bertanya-tanya mengapa kita masih membahas ini. Saya kira kita semua setuju bahwa scikit-learn harus mencoba yang terbaik dalam akurasi trade-off vs. waktu komputasi. Dan siapa pun yang tidak senang dengan keadaan saat ini harus mengajukan permintaan penarikan.
Penggunaan hanya "| b | ^ 2-2ab" tidak memiliki pembatalan bencana - tetapi hilangnya presisi yang sama dalam digit yang membuat perbedaan. Hasilnya sama seperti jika Anda menambahkan norma a ke setiap jarak sesudahnya; jika jarak jauh lebih kecil dari norma a, maka Anda mendapatkan kerugian dalam presisi yang dapat dihindari dengan melakukan perhitungan dengan cara tradisional tanpa hack BLAS.
Jadi Anda sebenarnya TIDAK bisa mengatasi masalah numerik dengan cara ini!
Ada kehilangan presisi, tetapi itu tidak dapat menyebabkan pembatalan bencana (setidaknya saat a dan b dekat), dan Anda dapat menunjukkan bahwa kesalahan relatif pada jarak (yang bukan jarak) tetap kecil.
Dalam kasus KMean di mana Anda hanya tertarik untuk menemukan pusat terdekat, Anda memiliki cukup presisi untuk menjaga urutannya tetap benar. Jika pada akhirnya Anda menginginkan inersia, maka Anda tinggal menghitung jarak setiap titik ke pusat klusternya dengan rumus yang tepat.
Selain itu, KMeans bukanlah masalah pengoptimalan konveks, jadi meskipun Anda membiarkannya berjalan dengan tol = 0 hingga konvergensi, Anda akan berakhir di minimum lokal yang bisa jauh dari minimum global (bahkan dengan inisialisasi kmeans ++). Jadi saya lebih suka menjalankan kmean berkali-kali dengan init berbeda dan jumlah iterasi yang cukup kecil. Anda akan memiliki kesempatan yang lebih baik untuk mendapatkan minimum lokal yang lebih baik. Kemudian Anda dapat menjalankan kembali yang terbaik sampai konvergensi.
Kesalahan relatif dibandingkan dengan jarak sebenarnya bisa sangat besar, dan karenanya menyebabkan tetangga terdekat yang salah. Perhatikan kasus di mana | a | ² = | b | ² = 1, misalnya pada tf-idf. Asumsikan bahwa vektor sangat dekat. Kemudian ab juga mendekati 1, dan pada titik ini Anda sudah kehilangan banyak ketepatan.
Seperti yang saya tulis di atas, kesalahannya ada, bahkan jika Anda tidak memiliki pembatalan bencana. Pertimbangkan presisi 8 digit. Jarak sebenarnya mungkin 0,000012345678 dan dapat dihitung dengan delapan digit menggunakan FP32 dan jarak Euclidean reguler. Tetapi dengan persamaan ini, Anda malah menghitung nilai ab = 0,99998765432, yang dengan FP32 akan dipotong paling banyak menjadi sekitar 0,9998765, jadi Anda kehilangan tiga digit ketepatan yang tidak perlu dalam contoh ini. Kerugian sebesar yang terjadi pada kasus bencana. Jika jaraknya jauh lebih kecil dari biasanya, presisi Anda bisa menjadi sangat buruk dengan pendekatan ini.
Ya, kmeans bukan cembung. Tetapi kemudian Anda akan ingin setidaknya menemukan optimal lokal , dan tidak macet (atau bahkan berosilasi karena kesalahan yang dihasilkan berperilaku tidak menentu) karena presisi Anda terlalu rendah. Jadi Anda setidaknya mendapat kesempatan untuk menemukan kasus global dalam kasus yang berperilaku baik dan dengan banyak upaya.
Saya menghargai diskusi ini, tetapi yang benar-benar kami butuhkan adalah solusi yang tidak lebih buruk dari apa yang kami lakukan sebelum kami berhenti membuat hal-hal mengambang64. Dalam hal ini, solusi menyiarkan @Celibi sudah cukup. Menggunakan solusi yang tepat dalam dimensi rendah merupakan peningkatan tambahan dari apa yang biasa kami lakukan.
Mengenai versi yang akan datang, apakah Anda merasa lebih percaya diri untuk mendeteksi secara efisien ketika kami dapat mempertimbangkan penghitungan yang tepat dalam dimensi tinggi?
Saya telah menjalankan patokan untuk mengevaluasi akurasi rata-rata kasus float64 dengan angka acak. Saya membandingkan 3 algoritma: neumaier_sum((x-y)**2) , numpy.sum((x-y)**2) dan X2 - 2*X.dot(Y.T) + Y2.T . Hasil yang tepat untuk dibandingkan telah diperoleh dengan menggunakan mpmath dengan presisi 256 bit.
X dan Y memiliki 100 sampel dan sejumlah fitur yang bervariasi dan diisi dengan nomor acak antara -2 dan 2.
Di gif berikut, ada satu gambar per jumlah fitur (antara 1 dan 200). Pada setiap gambar, setiap titik mewakili kesalahan relatif jarak euclidean kuadrat antara salah satu dari 10.000 pasang vektor X dan Y . Kesalahan relatif dikalikan dengan 2 ^ 53 untuk keterbacaan, yang kira-kira sesuai dengan unit ULP.
Kurva di atas adalah perkiraan distribusi (menggunakan perkiraan kerapatan kernel).

Perhatikan bahwa grafik dipotong pada 6 ULP agar mudah dibaca. Ini menunjukkan kasus rata-rata, bukan kasus yang lebih buruk. Kesalahan rumus yang diperluas bisa bertambah besar.
Analisis saya tentang hasil ini adalah bahwa secara rata-rata, kesalahan relatif dari rumus yang diperluas bisa sangat besar dengan sedikit fitur, tetapi dengan cepat menjadi mirip dengan perbedaan dan jumlah numpy. Ambang batasnya berada di antara 5 dan 10 fitur.
Saat ini saya juga mencoba menemukan batas atas untuk kesalahan rumus yang diperluas serta contoh patologis.
Saya pikir perhatian @ kno10 adalah bahwa kami sering tertarik pada kasus di mana
titik tidak didistribusikan secara acak, tetapi berdekatan satu sama lain atau memiliki unit
norma.
Memang, tapi saya perlu diyakinkan bahwa dalam praktiknya, itu bukan BS lengkap. ^^
Untuk melengkapi komentar di atas: kesalahan relatif dari rumus x²+y²-2ab tampaknya tidak dibatasi. Kecuali jika analisis saya salah, ketika x dan y saling berdekatan, kesalahan relatif bisa mencapai 2^(52*2) . Setidaknya secara teoritis. Dalam praktiknya, kasus terburuk yang saya temukan adalah kesalahan relatif 2^52+1 .
>>> a, b = (0xfffffec4d6282+1) * 2.0**(511-52), 0xfffffec4d6282 * 2.0**(511-52)
>>> a, b
(6.703903473040778e+153, 6.7039034730407766e+153)
>>> exactdiff = (a-b)**2
>>> exactdiff
2.2158278651204453e+276
>>> computeddiff = a**2 + b**2 - 2*a*b
>>> computeddiff
-9.9792015476736e+291
>>> abs((computeddiff - exactdiff) / exactdiff)
4503599627370497.0
>>> bin(int(abs((computeddiff - exactdiff) / exactdiff)))
'0b10000000000000000000000000000000000000000000000000001'
Membalik tanda hasil sebenarnya akan membuatnya lebih dekat dengan kebenaran. Ini adalah contoh paling dramatis yang dapat saya temukan, tetapi sebenarnya mengubah eksponen dalam nilai a dan b tidak mengubah kesalahan relatif.
>>> a, b = (0xfffffec4d6282+1) * 2.0**(-52), 0xfffffec4d6282 * 2.0**(-52)
>>> a, b
(0.9999999266202912, 0.999999926620291)
>>> exactdiff = (a-b)**2
>>> computeddiff = a**2 + b**2 - 2*a*b
>>> abs((computeddiff - exactdiff) / exactdiff)
4503599627370497.0
Saya pikir plot histogram di ULP akan lebih masuk akal daripada animasi di atas dengan distribusi kesalahan dalam-ULP. Jadi 0 kesalahan ULP dan 1 kesalahan ULP adalah "sebaik yang didapat". 2 ULP sepertinya tidak dapat dihindari karena sqrt. Saya berasumsi bahwa kesalahan yang lebih besar patut diselidiki.
Menggunakan (computed - exact) / exact masuk akal selama pastinya besar. Tapi begitu kita mendapatkan tantangan numerik untuk nilai pastinya, ini menjadi sangat tidak stabil. Dalam kasus seperti itu, (computed-exact)/norm mungkin layak digunakan, yaitu melihat ketepatan perhitungan jarak kita dibandingkan dengan data input, tidak dibandingkan dengan jarak yang diturunkan.
Jika kita memiliki dua nilai satu dimensi yang hanya berbeda 1 ULP, dan kesalahan 2 ULP mungkin tampak besar; tetapi kami sudah berada pada resolusi data input, jadi hasilnya cukup tidak stabil.
Perhatikan bahwa dengan beberapa dimensi, kami mungkin mendapatkan resolusi yang lebih tinggi dalam data masukan.
Pertimbangkan input data tipe (1, 1e-16) vs. (1, 2e-16) . Misalnya jika kita memiliki atribut konstan dalam data masukan, katakanlah, piksel putih di MNIST.
Dengan persamaan berbasis perbedaan ini akan baik-baik saja, tetapi versi titik mendapat masalah, bukan? Itulah sebabnya eksperimen satu dimensi mungkin tidak cukup untuk mempelajari ini.
Saya pikir plot histogram di ULP akan lebih masuk akal daripada animasi di atas dengan distribusi kesalahan dalam-ULP.
Saya tidak yakin saya mengerti bagaimana Anda akan merepresentasikannya. Akan ada satu histogram per jumlah fitur dan per algoritma. Tidak banyak yang bisa saya lakukan selain plot 3D atau animasi.
Menggunakan
(computed - exact) / exactmasuk akal selama pastinya besar. Tapi begitu kita mendapatkan tantangan numerik untuk nilai pastinya, ini menjadi sangat tidak stabil.
Saya tidak yakin apa yang Anda maksud dengan tidak stabil dalam konteks ini. Nilai pasti harus dihitung dengan apa pun yang diperlukan untuk membuatnya tepat.
(Ngomong-ngomong, saya seharusnya juga menghitung kesalahan relatif dengan presisi sewenang-wenang dalam plot saya, alih-alih membandingkan dengan hasil yang benar-benar bulat. Saya memperbarui plot saya, gelombang aneh menghilang.)
Dalam kasus seperti itu,
(computed-exact)/normmungkin layak digunakan, yaitu melihat ketepatan perhitungan jarak kita dibandingkan dengan data input, tidak dibandingkan dengan jarak yang diturunkan.
Jika saya memahami ide Anda dengan benar, Anda lebih suka membandingkan kesalahan absolut dengan besarnya data masukan. Menggunakan norma vektor sebagai ukuran agregat dari besarnya input. Sedangkan kesalahan relatif standar membandingkannya dengan besarnya hasil yang tepat.
Saya pikir dengan metrik ini Anda mencoba untuk menangkap seberapa banyak kesalahan algoritma. Tetapi sebenarnya itu tidak terlalu berguna karena beberapa alasan.
- Itu tidak benar-benar mengatakan berapa digit hasil yang tidak tepat.
- Sebenarnya, sebagian besar algoritme akan memiliki skor kurang dari 1e-15. Bahkan rumus yang diperluas (algoritma berbasis titik) akan memiliki skor yang dibatasi oleh sesuatu seperti 5 ULP (masukan) (perkiraan kasar, saya tidak menulis bukti lengkapnya).
- Dan karena kedua metrik hanyalah versi skala ulang dari kesalahan absolut
computed - exact, mereka akan memberi peringkat algoritme dalam urutan yang sama saat dievaluasi pada input yang sama.
Jadi sama saja dengan relative error biasa, hanya saja dengan nilai interpretasi kurang bermanfaat (IMO).
Pertimbangkan input data tipe
(1, 1e-16)vs.(1, 2e-16). Misalnya jika kita memiliki atribut konstan dalam data masukan, katakanlah, piksel putih di MNIST.
Dengan persamaan berbasis perbedaan ini akan baik-baik saja, tetapi versi titik mendapat masalah, bukan? Itulah sebabnya eksperimen satu dimensi mungkin tidak cukup untuk mempelajari ini.
Algoritme berbasis titik akan memiliki kesalahan relatif 1 , yang berarti bahwa kesalahan tersebut sebesar hasil yang tepat, dan dengan demikian, tidak ada digit hasil yang benar. Dan metrik Anda akan memiliki nilai 1e-16 berarti relatif terhadap skala norma vektor, hanya digit ke-16 yang tidak aktif.
Saya tidak yakin apa yang Anda coba tunjukkan dengan contoh ini.
Jika kita masih prihatin tentang ketepatan jarak_ euclidean dengan float64, mungkin lebih baik untuk meringkas diskusi ini dalam edisi baru karena ada 100 komentar di sini.
Komentar yang paling membantu
Nilai mentah dari dunia nyata jarang memiliki akurasi seperti itu, itu benar. Tetapi ML tidak terbatas pada jenis masukan tersebut. Seseorang mungkin ingin menerapkan ML ke masalah matematika, seperti menerapkan MDS pada grafik puzzle mirip kubus rubik atau mengelompokkan strategi sukses yang ditemukan oleh sekumpulan agen RL yang bermain pacman.
Meskipun sumber informasi awal adalah dunia nyata, mungkin ada beberapa pemrosesan tengah jalan yang membuat sebagian besar digit relevan dengan algoritme pengelompokan. Seperti hasil dari penurunan gradien pada fungsi yang parameternya diambil sampelnya secara statistik di dunia nyata.
Saya sebenarnya bertanya-tanya mengapa kita masih membahas ini. Saya kira kita semua setuju bahwa scikit-learn harus mencoba yang terbaik dalam akurasi trade-off vs. waktu komputasi. Dan siapa pun yang tidak senang dengan keadaan saat ini harus mengajukan permintaan penarikan.