Scikit-learn: Numerische Genauigkeit von euklidischen Abständen mit float32
Beschreibung
Ich habe festgestellt, dass die Funktion sklearn.metrics.pairwise.pairwise_distances mit np.linalg.norm übereinstimmt, wenn np.float64-Arrays verwendet werden, bei Verwendung von np.float32-Arrays jedoch nicht übereinstimmt. Siehe das Code-Snippet unten.
Schritte / Code zum Reproduzieren
import numpy as np
import scipy
import sklearn.metrics.pairwise
# create 64-bit vectors a and b that are very similar to each other
a_64 = np.array([61.221637725830078125, 71.60662841796875, -65.7512664794921875], dtype=np.float64)
b_64 = np.array([61.221637725830078125, 71.60894012451171875, -65.72847747802734375], dtype=np.float64)
# create 32-bit versions of a and b
a_32 = a_64.astype(np.float32)
b_32 = b_64.astype(np.float32)
# compute the distance from a to b using numpy, for both 64-bit and 32-bit
dist_64_np = np.array([np.linalg.norm(a_64 - b_64)], dtype=np.float64)
dist_32_np = np.array([np.linalg.norm(a_32 - b_32)], dtype=np.float32)
# compute the distance from a to b using sklearn, for both 64-bit and 32-bit
dist_64_sklearn = sklearn.metrics.pairwise.pairwise_distances([a_64], [b_64])
dist_32_sklearn = sklearn.metrics.pairwise.pairwise_distances([a_32], [b_32])
# note that the 64-bit sklearn results agree exactly with numpy, but the 32-bit results disagree
np.set_printoptions(precision=200)
print(dist_64_np)
print(dist_32_np)
print(dist_64_sklearn)
print(dist_32_sklearn)
erwartete Ergebnisse
Ich gehe davon aus, dass die Ergebnisse von sklearn.metrics.pairwise.pairwise_distances sowohl für 64-Bit als auch für 32-Bit mit np.linalg.norm übereinstimmen. Mit anderen Worten, ich erwarte die folgende Ausgabe:
[ 0.0229059506440019884643266578905240749008953571319580078125]
[ 0.02290595136582851409912109375]
[[ 0.0229059506440019884643266578905240749008953571319580078125]]
[[ 0.02290595136582851409912109375]]
Tatsächliche Ergebnisse
Das obige Code-Snippet erzeugt für mich die folgende Ausgabe:
[ 0.0229059506440019884643266578905240749008953571319580078125]
[ 0.02290595136582851409912109375]
[[ 0.0229059506440019884643266578905240749008953571319580078125]]
[[ 0.03125]]
Versionen
Darwin-16.6.0-x86_64-i386-64bit
('Python', '2.7.11 | 64-bit | (default, Jun 11 2016, 03:41:56) \n[GCC 4.2.1 Compatible Apple LLVM 6.0 (clang-600.0.57)]')
('NumPy', '1.11.3')
('SciPy', '0.19.0')
('Scikit-Learn', '0.18.1')
Alle 102 Kommentare
Gleiche Ergebnisse mit Python 3.5:
Darwin-15.6.0-x86_64-i386-64bit
Python 3.5.1 (v3.5.1:37a07cee5969, Dec 5 2015, 21:12:44)
[GCC 4.2.1 (Apple Inc. build 5666) (dot 3)]
NumPy 1.11.0
SciPy 0.18.1
Scikit-Learn 0.17.1
Es kommt nur mit euklidischer Distanz vor und kann direkt mit sklearn.metrics.pairwise.euclidean_distances reproduziert werden:
import scipy
import sklearn.metrics.pairwise
# create 64-bit vectors a and b that are very similar to each other
a_64 = np.array([61.221637725830078125, 71.60662841796875, -65.7512664794921875], dtype=np.float64)
b_64 = np.array([61.221637725830078125, 71.60894012451171875, -65.72847747802734375], dtype=np.float64)
# create 32-bit versions of a and b
a_32 = a_64.astype(np.float32)
b_32 = b_64.astype(np.float32)
# compute the distance from a to b using sklearn, for both 64-bit and 32-bit
dist_64_sklearn = sklearn.metrics.pairwise.euclidean_distances([a_64], [b_64])
dist_32_sklearn = sklearn.metrics.pairwise.euclidean_distances([a_32], [b_32])
np.set_printoptions(precision=200)
print(dist_64_sklearn)
print(dist_32_sklearn)
Ich konnte den Fehler nicht weiter aufspüren.
Ich hoffe das kann helfen.
numpy verwendet möglicherweise einen Akku mit höherer Genauigkeit. Ja, es sieht so aus
verdient es, repariert zu werden.
Am 19. Juli 2017 um 00:05 Uhr schrieb "nvauquie" [email protected] :
Gleiche Ergebnisse mit Python 3.5:
Darwin-15.6.0-x86_64-i386-64bit
Python 3.5.1 (v3.5.1: 37a07cee5969, 5. Dezember 2015, 21:12:44)
[GCC 4.2.1 (Apple Inc. Build 5666) (Punkt 3)]
NumPy 1.11.0
SciPy 0.18.1
Scikit-Learn 0.17.1Es kommt nur mit euklidischer Distanz vor und kann mit reproduziert werden
direkt sklearn.metrics.pairwise.euclidean_distances:scipy importieren
importiere sklearn.metrics.pairwiseErstellen Sie 64-Bit-Vektoren a und b, die einander sehr ähnlich sind
a_64 = np.array ([61.221637725830078125, 71.60662841796875, -65.7512664794921875], dtype = np.float64)
b_64 = np.array ([61.221637725830078125, 71.60894012451171875, -65.72847747802734375], dtype = np.float64)Erstellen Sie 32-Bit-Versionen von a und b
a_32 = a_64.astype (np.float32)
b_32 = b_64.astype (np.float32)Berechnen Sie den Abstand von a nach b mit sklearn für 64-Bit und 32-Bit
dist_64_sklearn = sklearn.metrics.pairwise.euclidean_distances ([a_64], [b_64])
dist_32_sklearn = sklearn.metrics.pairwise.euclidean_distances ([a_32], [b_32])np.set_printoptions (Genauigkeit = 200)
print (dist_64_sklearn)
print (dist_32_sklearn)Ich konnte den Fehler nicht weiter aufspüren.
Ich hoffe das kann helfen.- -
Sie erhalten dies, weil Sie diesen Thread abonniert haben.
Antworte direkt auf diese E-Mail und sieh sie dir auf GitHub an
https://github.com/scikit-learn/scikit-learn/issues/9354#issuecomment-316074315 ,
oder schalten Sie den Thread stumm
https://github.com/notifications/unsubscribe-auth/AAEz65yy8Aq2FcsDAcWHT8qkkdXF_MfPks5sPLu_gaJpZM4OXbpZ
.
Ich würde gerne daran arbeiten, wenn möglich
Tue es!
Ich denke, das Problem liegt in der Tatsache, dass wir sqrt(dot(x, x) - 2 * dot(x, y) + dot(y, y)) für die Berechnung der euklidischen Distanz verwenden
Denn wenn ich versuche - (-2 * np.dot(X, Y.T) + (X * X).sum(axis=1) + (Y * Y).sum(axis=1) bekomme ich die Antwort 0 für np.float32, während ich die richtige Antwort für np.float 64 bekomme.
@jnothman Was denkst du sollte ich dann tun? Wie in meinem obigen Kommentar erwähnt, besteht das Problem wahrscheinlich darin, die euklidische Entfernung mit sqrt(dot(x, x) - 2 * dot(x, y) + dot(y, y)) berechnen
Sie sagen also, dass dot ein weniger genaues Ergebnis liefert als Produkt-dann-Summe?
Nein, ich versuche zu sagen, dass dot ein genaueres Ergebnis liefert als Produkt-dann-Summe
-2 * np.dot(X, Y.T) + (X * X).sum(axis=1) + (Y * Y).sum(axis=1) gibt die Ausgabe [[0.]]
während np.sqrt(((X-Y) * (X-Y)).sum(axis=1)) die Ausgabe [ 0.02290595] ergibt
Es ist nicht klar, was Sie tun, auch weil Sie kein vollständig eigenständiges Snippet veröffentlichen.
Wenn Sie sich Ihren letzten Beitrag kurz ansehen, haben die beiden Dinge, die Sie versuchen, [[0.]] und [0.022...] , nicht die gleichen Abmessungen (möglicherweise ein Problem beim Kopieren und Einfügen, aber wiederum schwer zu wissen, weil wir dies nicht tun habe einen vollständigen Ausschnitt).
Ok, tut mir leid, mein schlechtes
import numpy as np
import scipy
from sklearn.metrics.pairwise import check_pairwise_arrays, row_norms
from sklearn.utils.extmath import safe_sparse_dot
# create 64-bit vectors a and b that are very similar to each other
a_64 = np.array([61.221637725830078125, 71.60662841796875, -65.7512664794921875], dtype=np.float64)
b_64 = np.array([61.221637725830078125, 71.60894012451171875, -65.72847747802734375], dtype=np.float64)
# create 32-bit versions of a and b
X = a_64.astype(np.float32)
Y = b_64.astype(np.float32)
X, Y = check_pairwise_arrays(X, Y)
XX = row_norms(X, squared=True)[:, np.newaxis]
YY = row_norms(Y, squared=True)[np.newaxis, :]
#Euclidean distance computed using product-then-sum
distances = safe_sparse_dot(X, Y.T, dense_output=True)
distances *= -2
distances += XX
distances += YY
print(np.sqrt(distances))
#Euclidean distance computed using (X-Y)^2
print(np.sqrt(row_norms(X-Y, squared=True)[:, np.newaxis]))
AUSGABE
[[ 0.03125]]
[[ 0.02290595136582851409912109375]]
Die erste Methode ist die Berechnung durch die euklidische Distanzfunktion.
Um zu verdeutlichen, was ich oben gemeint habe, war auch die Tatsache, dass Summe-dann-Produkt eine geringere Genauigkeit aufweist, selbst wenn wir dazu Numpy-Funktionen verwenden
Ja, ich kann das replizieren. Ich sehe das zunächst bei der Subtraktion
ermöglicht die Aufrechterhaltung der Genauigkeit der Differenz. Den Punkt machen
Produkt und dann subtrahieren (oder negieren und addieren), wie wir es derzeit tun,
verliert diese Präzision, da die wichtigsten Zahlen viel größer sind als
die Unterschiede.
Die aktuelle Implementierung ist speichereffizienter für eine hohe Anzahl von
Eigenschaften. Aber ich nehme an, dass die euklidische Distanz zunehmend irrelevant wird
In hohen Dimensionen wird der Speicher also von der Anzahl der Ausgaben dominiert
Werte.
Deshalb stimme ich für die Übernahme der zahlenmäßig stabileren Implementierung gegenüber dem
d-asymptotisch effiziente Implementierung, die wir derzeit haben. Eine Meinung,
@ogrisel? @agramfort?
Und das ist natürlich ein größeres Problem, da wir kürzlich float32s zugelassen haben
über Schätzer hinweg alltäglicher zu sein.
In diesem Beispiel funktioniert product-then-sum für np.float64 einwandfrei. Eine mögliche Lösung könnte darin bestehen, die Eingabe in float64 zu konvertieren, dann das Ergebnis zu berechnen und das in float32 konvertierte Ergebnis zurückzugeben. Ich denke, dies wäre effizienter, bin mir aber nicht sicher, ob dies für ein anderes Beispiel gut funktionieren würde.
Die Konvertierung in float64 ist bei der Speichernutzung nicht effizienter als
Subtraktion.
Oh ja, das tut Ihnen zu Recht leid, aber ich denke, dass die Verwendung von float64 und die anschließende Ausführung von Produkt-dann-Summe rechnerisch effizienter wäre, wenn nicht in Bezug auf den Speicher.
Der Grund für die Verwendung von Produkt-dann-Summe war eine höhere Recheneffizienz und keine Speichereffizienz.
Sicher, aber ich glaube nicht, dass es einen Grund gibt anzunehmen, dass es tatsächlich so ist
rechnerisch effizienter, außer um nicht realisieren zu müssen
Zwischenarray. Angenommen, wir begrenzen den absoluten Arbeitsspeicher (z. B. durch
Chunking), warum sollte man das Punktprodukt nehmen, Normen verdoppeln und subtrahieren?
viel effizienter sein als subtrahieren und quadrieren?
Benchmarks bereitstellen?
Ok, also habe ich ein Python-Skript erstellt, um die Zeit zu vergleichen, die durch Subtraktion, Quadrieren und Konvertieren in float64 und dann Produkt-dann-Summe benötigt wird. Wenn wir X und Y als sehr große Vektoren auswählen, sind die beiden Ergebnisse sehr unterschiedlich . Auch @jnothman du
Hier ist das Skript, das ich geschrieben habe. Wenn es ein Problem gibt, lass es mich wissen
import numpy as np
import scipy
from sklearn.metrics.pairwise import check_pairwise_arrays, row_norms
from sklearn.utils.extmath import safe_sparse_dot
from timeit import default_timer as timer
for i in range(9):
X = np.random.rand(1,3 * (10**i)).astype(np.float32)
Y = np.random.rand(1,3 * (10**i)).astype(np.float32)
X, Y = check_pairwise_arrays(X, Y)
XX = row_norms(X, squared=True)[:, np.newaxis]
YY = row_norms(Y, squared=True)[np.newaxis, :]
#Euclidean distance computed using product-then-sum
distances = safe_sparse_dot(X, Y.T, dense_output=True)
distances *= -2
distances += XX
distances += YY
ans1 = np.sqrt(distances)
start = timer()
ans2 = np.sqrt(row_norms(X-Y, squared=True)[:, np.newaxis])
end = timer()
if ans1 != ans2:
print(end-start)
start = timer()
X = X.astype(np.float64)
Y = Y.astype(np.float64)
X, Y = check_pairwise_arrays(X, Y)
XX = row_norms(X, squared=True)[:, np.newaxis]
YY = row_norms(Y, squared=True)[np.newaxis, :]
distances = safe_sparse_dot(X, Y.T, dense_output=True)
distances *= -2
distances += XX
distances += YY
distances = np.sqrt(distances)
end = timer()
print(end-start)
print('')
if abs(ans2 - distances) > 1e-3:
# np.set_printoptions(precision=200)
print(ans2)
print(np.sqrt(distances))
print(X, Y)
break
Es lohnt sich zu testen, wie es mit der Anzahl der Proben skaliert, nicht nur mit der
Anzahl der Funktionen ... Normen zu übernehmen kann den Vorteil haben, einige zu berechnen
Dinge einmal pro Probe, nicht einmal pro Probenpaar
Am 20. Oktober 2017, 02:39 Uhr, "Osaid Rehman Nasir" [email protected]
schrieb:
Ok, also habe ich ein Python-Skript erstellt, um die benötigte Zeit zu vergleichen
Subtraktion-dann-Quadrieren und Umwandlung in float64, dann Produkt-dann-Summe
und es stellt sich heraus, wenn wir ein X und ein Y als sehr große Vektoren wählen, dann die 2
Ergebnisse sind sehr unterschiedlich. Auch @jnothman https://github.com/jnothman
Sie hatten Recht Subtraktion-dann-Quadrieren ist schneller.
Hier ist das Skript, das ich geschrieben habe. Wenn es ein Problem gibt, lass es mich wissenimportiere numpy als np
scipy importieren
Importieren Sie aus sklearn.metrics.pairwise check_pairwise_arrays, row_norms
aus sklearn.utils.extmath importiere safe_sparse_dot
aus timeit importiere default_timer als timerfür i im Bereich (9):
X = np.random.rand (1,3 * (10 i)). Astype (np.float32)Y = np.random.rand (1,3 * (10 i)). Astype (np.float32)X, Y = check_pairwise_arrays (X, Y)
XX = row_norms (X, squared = True) [:, np.newaxis]
YY = row_norms (Y, squared = True) [np.newaxis ,:]#Euklidischer Abstand berechnet mit Produkt-dann-Summe
Entfernungen = safe_sparse_dot (X, YT, dens_output = True)
Abstände * = -2
Entfernungen + = XX
Entfernungen + = JJans1 = np.sqrt (Entfernungen)
start = timer ()
ans2 = np.sqrt (row_norms (XY, squared = True) [:, np.newaxis])
Ende = Timer ()
wenn ans1! = ans2:
Drucken (Ende-Start)start = timer() X = X.astype(np.float64) Y = Y.astype(np.float64) X, Y = check_pairwise_arrays(X, Y) XX = row_norms(X, squared=True)[:, np.newaxis] YY = row_norms(Y, squared=True)[np.newaxis, :] distances = safe_sparse_dot(X, Y.T, dense_output=True) distances *= -2 distances += XX distances += YY distances = np.sqrt(distances) end = timer() print(end-start) print('') if abs(ans2 - distances) > 1e-3: # np.set_printoptions(precision=200) print(ans2) print(np.sqrt(distances)) print(X, Y) break- -
Sie erhalten dies, weil Sie erwähnt wurden.
Antworte direkt auf diese E-Mail und sieh sie dir auf GitHub an
https://github.com/scikit-learn/scikit-learn/issues/9354#issuecomment-337948154 ,
oder schalten Sie den Thread stumm
https://github.com/notifications/unsubscribe-auth/AAEz6z5o2Ao_7V5-Lflb4HosMrHCeOrVks5st209gaJpZM4OXbpZ
.
Wie auch immer, möchten Sie eine PR einreichen, @ragnerok?
Ja klar, was soll ich tun?
bieten eine stabilere Implementierung, auch einen Test, der unter dem fehlschlagen würde
aktuelle Implementierung und im Idealfall ein Benchmark, der zeigt, dass wir nicht verlieren
viel von der Änderung, in vernünftigen Fällen.
Ich wollte fragen, ob es möglich ist, mit Vektorisierung einen Abstand zwischen jedem Zeilenpaar zu finden. Ich kann nicht darüber nachdenken, wie ich es vektorisiert machen soll.
Du meinst Unterschied (nicht Abstand) zwischen Reihenpaaren? Sicher können Sie das tun, wenn Sie mit numpy Arrays arbeiten. Wenn Sie Arrays mit Formen (n_samples1, n_features) und (n_samples2, n_features) haben, müssen Sie sie nur in (n_samples1, 1, n_features) und (1, n_samples2, n_features) umformen und die Subtraktion durchführen:
>>> X = np.random.randint(10, size=(10, 5))
>>> Y = np.random.randint(10, size=(11, 5))
X.reshape(-1, 1, X.shape[1]) - Y.reshape(1, -1, X.shape[1])
Ja danke, das hat wirklich geholfen 😄
Ich wollte auch fragen, ob ich eine stabilere Implementierung zur Verfügung stelle. Ich werde X_norm_squared und Y_norm_squared nicht verwenden. Entferne ich sie auch aus den Argumenten oder sollte ich davor warnen, dass sie keinen Nutzen haben?
Ich denke, sie werden veraltet sein, aber das muss uns vielleicht erst versichert werden
Es gibt keinen Fall, in dem wir diese Version behalten sollten.
Wir werden sehr vorsichtig sein, wenn wir das ändern. es ist ein weit verbreitetes und
langjährige Umsetzung. Wir sollten sicher sein, nichts Wichtiges zu verlangsamen
Fälle. Möglicherweise müssen wir die Operation in Blöcken ausführen, um einen hohen Arbeitsspeicher zu vermeiden
Verwendung (was vielleicht dadurch schwieriger wird, dass dies aufgerufen wird
innerhalb von Funktionen, die Chunk, um den Ausfall des Ausgabespeichers von zu minimieren
paarweise Abstände).
Ich würde wirklich gerne von anderen Kernentwicklern hören, die sich mit Computern auskennen
Kosten und numerische Genauigkeit ... @ogrisel , @lesteve , @rth ...
Am 5. November 2017, 05:27 Uhr, "Osaid Rehman Nasir" [email protected]
schrieb:
Ich wollte auch fragen, ob ich eine stabilere Implementierung bereitstellen werde, die ich nicht sein werde
mit X_norm_squared und Y_norm_squared. Also entferne ich sie aus dem
Argumente auch oder sollte ich davor warnen, dass es nicht von Nutzen ist?- -
Sie erhalten dies, weil Sie erwähnt wurden.
Antworte direkt auf diese E-Mail und sieh sie dir auf GitHub an
https://github.com/scikit-learn/scikit-learn/issues/9354#issuecomment-341919282 ,
oder schalten Sie den Thread stumm
https://github.com/notifications/unsubscribe-auth/AAEz63izdpQGDEuW32m8Aob6rrsvV6q-ks5szKyHgaJpZM4OXbpZ
.
Es wäre jedoch einfacher, genau zu diskutieren, wenn Sie eine PR eröffnen
Ok, ich werde dann eine PR mit einer sehr grundlegenden Implementierung dieser Funktion eröffnen
Die Frage ist, was diesbezüglich für die Version 0.20 getan werden sollte. Könnte es einige einfache / vorübergehende Verbesserungen geben (Ereignis auf Kosten, z. B. der Speichernutzung), die in Betracht gezogen werden könnten?
Die in # 11271 vorgeschlagene Lösung und Analyse sind definitiv sehr wertvoll, aber es könnte weitere Diskussionen erfordern, um sicherzustellen, dass dies die optimale Lösung ist. Insbesondere bin ich besorgt über die Tatsache, dass derzeit einige Diskussionen über den optimalen globalen Arbeitsspeicher in https://github.com/scikit-learn/scikit-learn/issues/11506 in Abhängigkeit vom CPU-Typ usw. anstehen würde noch eine weitere Ebene des Chunking hinzufügen und die Komplexität des Ganzen würde ein bisschen Kontrolle IMO bekommen. Aber vielleicht bin ich es nur, der nach einer zweiten Meinung sucht.
Was sollte Ihrer Meinung nach in Bezug auf dieses Problem für die Veröffentlichung @jnothman @amueller @ogrisel getan werden ?
Stabilität übertrifft Effizienz. Stabilitätsprobleme sollten auch dann behoben werden, wenn
Effizienz muss noch verbessert werden.
Der Fokus von working_memory lag darauf, Dinge wie eine Silhouette mit einer großen Stichprobe zu erstellen
Größen funktionieren. Es hat auch die Effizienz verbessert, aber das kann optimiert werden
Linie.
Ich bin der festen Überzeugung, dass wir versuchen sollten, eine Lösung für euklidische Abstände mit zu finden
float32 in. Wir haben es in 0.19 gebrochen, indem wir angenommen haben, dass wir machen könnten
euclidean_distances arbeiten auf naive Weise mit 32 Bit.
Ich bin damit einverstanden, dass wir eine Lösung brauchen. Mein Anliegen ist hier nicht die Effizienz, sondern die zusätzliche Komplexität der Codebasis.
Wenn Sie einen Schritt zurücktreten, scheint die euklidische Implementierung von scipy aus 10 Zeilen C-Code zu bestehen, und für 32 Bit wandeln Sie sie einfach in 64 Bit um. Ich verstehe, dass es nicht das schnellste ist, aber es ist konzeptionell einfach zu folgen und zu verstehen. Beim Scikit-Lernen verwenden wir den Trick, um Berechnungen in BLAS zu beschleunigen. Dann gibt es mögliche Verbesserungen aufgrund von https://github.com/scikit-learn/scikit-learn/pull/10212 und jetzt die mögliche Chunked-Lösung für Euklidisch Abstand in 32 Bit.
Ich bin nur auf der Suche nach Informationen darüber, wie die allgemeine Richtung zu diesem Thema lauten soll (z. B. versuchen Sie, einen Teil davon auf scipy usw. zu übertragen).
scipy scheint sich keine Sorgen um das Kopieren der Daten zu machen ...
Gehen Sie nach der PR auf 0,21.
Blocker entfernen?
sqrt(dot(x, x) - 2 * dot(x, y) + dot(y, y))
ist numerisch instabil, wenn Punkt (x, x) und Punkt (y, y) aufgrund der sogenannten katastrophalen Löschung eine ähnliche Größe wie Punkt (x, y) haben.
Dies wirkt sich nicht nur auf die Genauigkeit von FP32 aus, sondern ist natürlich auch wichtiger und wird viel früher fehlschlagen.
Hier ist ein einfacher Testfall, der zeigt, wie schlimm dies auch bei doppelter Genauigkeit ist:
import numpy
from sklearn.metrics.pairwise import euclidean_distances
a = numpy.array([[100000001, 100000000]])
b = numpy.array([[100000000, 100000001]])
print "skelarn:", euclidean_distances(a, b)[0,0]
print "correct:", numpy.sqrt(numpy.sum((a-b)**2))
a = numpy.array([[10001, 10000]], numpy.float32)
b = numpy.array([[10000, 10001]], numpy.float32)
print "skelarn:", euclidean_distances(a, b)[0,0]
print "correct:", numpy.sqrt(numpy.sum((a-b)**2))
sklearn berechnet hier beide Male einen Abstand von 0 anstelle von sqrt (2).
Eine Diskussion der numerischen Probleme für Varianz und Kovarianz - und dies überträgt sich trivial auf diesen Ansatz der Beschleunigung der euklidischen Distanz - finden Sie hier:
Erich Schubert und Michael Gertz.
Numerisch stabile parallele Berechnung der (Co-) Varianz.
In: Tagungsband der 30. Internationalen Konferenz für wissenschaftliches und statistisches Datenbankmanagement (SSDBM), Bozen, Italien. 2018, 10: 1–10: 12
Tatsächlich kann die y-Koordinate aus diesem Testfall entfernt werden, der richtige Abstand wird dann trivial zu 1. Ich habe eine Pull-Anfrage gestellt, die dieses numerische Problem auslöst:
XA = np.array([[10000]], np.float32)
XB = np.array([[10001]], np.float32)
assert_equal(euclidean_distances(XA, XB)[0,0], 1)
Ich glaube nicht, dass mein oben erwähntes Papier eine Lösung für dieses Problem bietet - berechnen Sie einfach den euklidischen Abstand als sqrt (sum (power ())) und es ist ein Durchgang und hat eine angemessene Genauigkeit. Der Verlust besteht darin, dass die Quadrate bereits verwendet werden, dh der Punkt (x, x) selbst verliert bereits die Genauigkeit.
@amueller Da das Problem
Vielen Dank für dieses sehr einfache Beispiel.
Der Grund, warum es so implementiert wird, ist, dass es viel schneller ist. Siehe unten:
x = np.random.random_sample((1000, 1000))
%timeit euclidean_distances(x,x)
20.6 ms ± 452 µs per loop (mean ± std. dev. of 7 runs, 10 loops each)
%timeit cdist(x,x)
695 ms ± 4.06 ms per loop (mean ± std. dev. of 7 runs, 1 loop each)
Obwohl die Anzahl der Operationen in beiden Methoden in der gleichen Größenordnung liegt (1,5x mehr in der zweiten), ergibt sich die Beschleunigung aus der Möglichkeit, gut optimierte BLAS-Bibliotheken für die Matrixmatrixmultiplikation zu verwenden.
Dies wäre eine enorme Verlangsamung für mehrere Schätzer beim Scikit-Lernen.
Ja, aber nur 3-4 Ziffern der Präzision , mit FP32 und 7-8 Ziffern mit FP64 verursacht erheblichen Unschärfen, nicht wahr? Insbesondere, da solche Fehler dazu neigen, sich zu verstärken ...
Nun, ich sage nicht, dass es jetzt in Ordnung ist. :) :)
Ich sage, wir müssen dazwischen eine Lösung finden.
Es gibt eine PR (# 11271), die vorschlägt, float64 zu verwenden, um die Berechnungen durchzuführen. In behebt das Problem für float64 nicht, bietet jedoch eine bessere Genauigkeit für float32.
Haben Sie ein Beispiel, in dem die Verwendung eines Schätzers, der euklidische_Distanzen verwendet, aufgrund des Genauigkeitsverlusts zu falschen Ergebnissen führt?
Ich denke sicherlich immer noch, dass dies eine große Sache ist und ein Blocker für 0,21 sein sollte. Es war ein Problem, das für 32 Bit in 0.19 eingeführt wurde, und es ist kein guter Zustand, es zu verlassen. Ich wünschte, wir hätten es früher in 0.20 gelöst, und ich wäre in Ordnung oder sogar gespannt darauf, dass # 11271 in der Zwischenzeit zusammengeführt wird. Die einzigen Probleme in dieser PR, die ich von der Surround-Optimierung der Speichereffizienz kenne, sind ein tiefes Kaninchenloch.
Wir haben diese "schnelle" Version schon lange, aber immer in float64. Ich weiß, @ kno10 , dass es Probleme mit der Präzision gibt. Haben Sie eine gute und schnelle Heuristik, mit der wir herausfinden können, wann dies ein Problem sein könnte, und eine langsamere, aber sicherere Lösung verwenden können?
Ja, aber nur 3-4 Stellen Genauigkeit mit FP32 und 7-8 Stellen mit FP64 verursachen erhebliche Ungenauigkeiten, nicht wahr?
Vielen Dank, dass Sie dieses Problem anhand eines sehr einfachen Beispiels veranschaulicht haben!
Ich denke jedoch nicht, dass das Problem so weit verbreitet ist, wie Sie es vorschlagen - es betrifft hauptsächlich Stichproben, deren gegenseitiger Abstand in Bezug auf ihre Normen gering ist.
Die folgende Abbildung veranschaulicht dies für 2e6-Zufallsstichprobenpaare, bei denen jede 1D-Stichprobe im Intervall [-100, 100] liegt. Der relative Fehler zwischen der Scikit-Learn- und der Scipy-Implementierung wird als Funktion des Abstands zwischen den Proben aufgetragen, normalisiert durch ihre L2-Normen, d. H.
d_norm(A, B) = d(A, B) / sqrt(‖A‖₂*‖B‖₂)
(nicht sicher, ob es die richtige Parametrisierung ist, aber nur um Ergebnisse zu erhalten, die für die Datenskala etwas unveränderlich sind),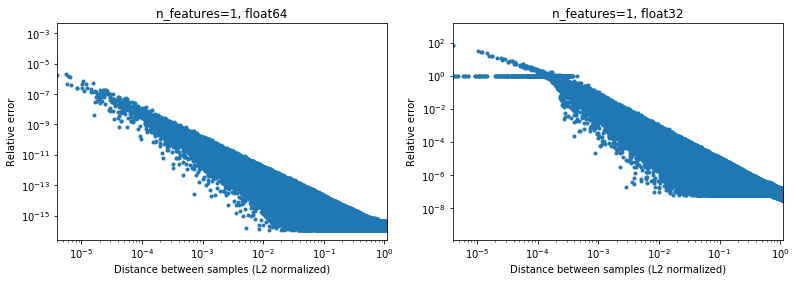
Zum Beispiel,
- Wenn man
[10000]und[10001]der normalisierte L2-Abstand 1e-4 und der relative Fehler bei der Entfernungsberechnung ist 1e-8 in 64 Bit und> 1 in 32 Bit (oder 1e) -8 bzw.> 1 in absoluten Werten). In 32 Bit ist dieser Fall in der Tat ziemlich schrecklich. - Auf der anderen Seite beträgt der relative Fehler für
[1]und[10001]~ 1e-7 in 32 Bit oder die maximal mögliche Genauigkeit.
Die Frage ist, wie oft der Fall 1. in der Praxis in ML-Anwendungen auftreten wird.
Interessanterweise wird es schwierig sein, Punkte zu finden, die sehr nahe beieinander liegen, wenn wir wieder mit einer gleichmäßigen Zufallsverteilung zu 2D gehen.
In der Realität werden unsere Daten natürlich nicht einheitlich abgetastet, aber bei jeder Verteilung aufgrund des Fluches der Dimensionalität konvergiert der Abstand zwischen zwei beliebigen Punkten langsam zu sehr ähnlichen Werten (von 0 verschieden), wenn die Dimentionalität zunimmt. Während es sich um ein allgemeines ML-Problem handelt, kann es dieses Genauigkeitsproblem hier selbst bei relativ geringer Dimensionalität etwas abschwächen. Unter den Ergebnissen für n_features=5 ,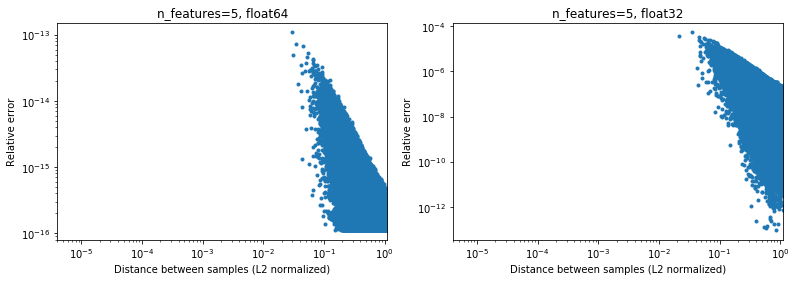 .
.
Für zentrierte Daten, zumindest in 64-Bit, ist dies in der Praxis möglicherweise kein so großes Problem (vorausgesetzt, es gibt mehr als zwei Funktionen). Die 50-fache Rechenbeschleunigung (wie oben dargestellt) kann sich lohnen (in 64 Bit). Natürlich kann man einigen in [-1, 1] normalisierten Daten immer 1e6 hinzufügen und sagen, dass die Ergebnisse nicht genau sind, aber ich würde argumentieren, dass dies auch für eine Reihe von numerischen Algorithmen gilt und mit Daten arbeitet, die im 6. ausgedrückt werden signifikante Ziffer sucht nur nach Ärger.
(Den Code für die obigen Abbildungen finden Sie hier ).
Jeder schnelle Ansatz mit der Version Punkt (x, x) + Punkt (y, y) -2 Ich glaube, Sie müssen die Präzision der Punktprodukte verdoppeln, um ca.
Ich kann nicht sagen, wie viel Aufwand Sie durch die sofortige Konvertierung von float32 in float64 für die Verwendung dieses Ansatzes erhalten. Zumindest für float32 sollte es meines Wissens in Ordnung sein, alle Berechnungen durchzuführen und die Punktprodukte als float64 zu speichern.
Meiner Meinung nach sind die Leistungssteigerungen (die nicht exponentiell sind, sondern nur ein konstanter Faktor) den Präzisionsverlust (der Sie unerwartet beißen kann) nicht wert, und der richtige Weg ist, diesen problematischen Trick nicht anzuwenden. Es kann jedoch durchaus möglich sein, Code, der die "traditionelle" Berechnung durchführt, weiter zu optimieren, beispielsweise um AVX zu verwenden. Weil sum ((xy) * 2) in AVX so gut wie schwer zu implementieren ist.Zumindest würde ich vorschlagen, die Methode wegen der manchmal geringen Genauigkeit in approximate_euclidean_distances umzubenennen (was umso schlimmer wird, je näher zwei Werte liegen, die * zunächst in Ordnung sein
@rth danke für die Illustrationen. Aber was ist, wenn Sie versuchen, z. B. x in Richtung eines Optimums zu optimieren? Höchstwahrscheinlich liegt das Optimum nicht bei Null (wenn es immer Ihr Rechenzentrum wäre, wäre das Leben großartig), und schließlich können die Deltas, die Sie für Gradienten usw. berechnen, einige sehr kleine Unterschiede aufweisen.
In ähnlicher Weise werden beim Clustering nicht alle Cluster ihre Zentren nahe Null haben, aber insbesondere bei vielen Clustern ist ein x ≈ -Zentrum mit wenigen Ziffern durchaus möglich.
Insgesamt stimme ich jedoch zu, dass dieses Problem behoben werden muss. In jedem Fall müssen wir die Präzisionsprobleme der aktuellen Implementierung so schnell wie möglich dokumentieren.
Im Allgemeinen denke ich jedoch nicht, dass diese Diskussion in Scikit-Learn stattfinden sollte. Die euklidische Distanz wird in verschiedenen Bereichen des wissenschaftlichen Rechnens verwendet, und IMO-Scipy-Mailinglisten oder -Probleme sind ein besserer Ort, um darüber zu diskutieren: Diese Community hat auch mehr Erfahrung mit solchen numerischen Präzisionsproblemen. Tatsächlich haben wir hier einen schnellen, aber etwas ungefähren Algorithmus. Möglicherweise müssen wir kurzfristig einige Problemumgehungen für Korrekturen implementieren, aber langfristig wäre es gut zu wissen, dass dies dort dazu beitragen wird.
Für 32-Bit ist https://github.com/scikit-learn/scikit-learn/pull/11271 in der Tat eine Lösung. Ich bin einfach nicht so begeistert von mehreren Chunking-Ebenen in der gesamten Bibliothek, da dies die Codekomplexität erhöht und möchten sicherstellen, dass es keinen besseren Weg gibt, um es zu umgehen.
Danke für deine Antwort @ kno10! (Meine obigen Kommentare berücksichtigen dies noch nicht.) Ich werde etwas später antworten.
Ja, die Konvergenz zu einem Punkt außerhalb des Ursprungs kann ein Problem sein.
Meiner Meinung nach sind die Leistungssteigerungen (die nicht exponentiell sind, sondern nur ein konstanter Faktor) den Präzisionsverlust (der Sie unerwartet beißen kann) nicht wert, und der richtige Weg ist, diesen problematischen Trick nicht anzuwenden.
Nun, eine> 10-fache Verlangsamung für ihre Berechnung in 64 Bit hat einen sehr realen Effekt auf die Benutzer.
Es kann jedoch durchaus möglich sein, Code, der die "traditionelle" Berechnung durchführt, weiter zu optimieren, beispielsweise um AVX zu verwenden. Weil sum ((xy) ** 2) in AVX so gut wie schwer zu implementieren ist.
Versuchte eine schnelle naive Implementierung mit numba (die SSE verwenden sollte),
@numba.jit(nopython=True, fastmath=True)
def pairwise_distance_naive(A, B):
n_samples_a, n_features_a = A.shape
n_samples_b, n_features_b = B.shape
assert n_features_a == n_features_b
distance = np.empty((n_samples_a, n_samples_b), dtype=A.dtype)
for i in range(n_samples_a):
for j in range(n_samples_b):
psum = 0.0
for k in range(n_features_a):
psum += (A[i, k] - B[j, k])**2
distance[i, j] = math.sqrt(psum)
return distance
eine ähnliche Geschwindigkeit zu erreichen wie bisher cdist (aber ich bin kein Numba-Experte) und auch nicht sicher über die Wirkung von fastmath .
Verwenden der Version Punkt (x, x) + Punkt (y, y) -2 * Punkt (x, y)
Zur späteren Bezugnahme tun wir derzeit ungefähr Folgendes (da es eine Dimension gibt, die in der obigen Notation nicht angezeigt wird):
def quadratic_pairwise_distance(A, B):
A2 = np.einsum('ij,ij->i', A, A)
B2 = np.einsum('ij,ij->i', B, B)
return np.sqrt(A2[:, None] + B2[None, :] - 2*np.dot(A, B.T))
Wobei sowohl einsum als auch dot jetzt BLAS verwenden. Ich frage mich, ob dies neben der Verwendung von BLAS auch die gleiche Anzahl mathematischer Operationen ausführt wie die erste Version oben.
Ich frage mich, ob dies neben der Verwendung von BLAS auch die gleiche Anzahl mathematischer Operationen ausführt wie die erste Version oben.
Nein. Die ((x - y) * 2.sum ()) wird ausgeführt* n_samples_x * n_samples_y * n_features * (1 Subtraktion + 1 Addition + 1 Multiplikation)
wohingegen xx + yy -2x.y ausgeführt wird
n_samples_x * n_samples_y * n_features * (1 Addition + 1 Multiplikation) .
Es gibt ein Verhältnis von 2/3 für die Anzahl der Operationen zwischen den beiden Versionen.
Nach der obigen Diskussion
- PR erstellt, um optional die Berechnung euklidischer Entfernungen genau zu ermöglichen https://github.com/scikit-learn/scikit-learn/pull/12136
- Einige WIP, um zu sehen, ob wir die problematischen Punkte in https://github.com/scikit-learn/scikit-learn/pull/12142 erkennen und abmildern können
Für 32-Bit müssen wir noch https://github.com/scikit-learn/scikit-learn/pull/11271 in irgendeiner Form zusammenführen, obwohl IMO, die obigen PRs etwas orthogonal dazu sind.
Zu Ihrer Information: Wenn Sie einige Probleme in OPTICS beheben und den Test aktualisieren, um Referenzergebnisse von ELKI zu verwenden, schlagen diese mit metric="euclidean" fehl, sind jedoch mit metric="minkowski" . Die numerischen Unterschiede sind groß genug, um eine andere Verarbeitungsreihenfolge zu verursachen (nur das Verringern des Schwellenwerts reicht nicht aus).
Ich bin wirklich nicht davon überzeugt, aber ich bin überrascht, dass es keine Lösung gibt. Dies scheint eine sehr häufige Berechnung zu sein und es sieht so aus, als würden wir das Rad neu erfinden. Hat jemand versucht, die breitere wissenschaftliche Computergemeinschaft zu erreichen?
Noch nicht, aber ich stimme zu, wir sollten. Das einzige, was ich in scipy darüber gefunden habe, war https://github.com/scipy/scipy/pull/2815 und verknüpfte Probleme.
Ich glaube, @jeremiedbb könnte eine Idee haben?
Leider noch nicht zufriedenstellend :(
Wir möchten uns für diese Art der Berechnung auf eine hochoptimierte Bibliothek verlassen, wie wir es für die lineare Algebra mit BLAS-Bibliotheken wie OpenBLAS oder MKL tun. Aber die euklidische Distanz gehört nicht dazu. Der Punkttrick ist ein Versuch, dies unter Verwendung der Matrix-Matrix-Multiplikations-Subroutine BLAS Level 3 zu tun. Dies ist jedoch nicht präzise und es gibt keine Möglichkeit, es mit derselben Methode präziser zu gestalten. Wir müssen unsere Erwartung entweder in Bezug auf Geschwindigkeit oder Präzision senken.
Ich denke, in einigen Situationen ist volle Präzision nicht zwingend erforderlich und die Beibehaltung der schnellen Methode ist in Ordnung. In diesem Fall werden die Entfernungen verwendet, um die nächstgelegenen Aufgaben zu finden. Die Präzisionsprobleme bei der schnellen Methode treten auf, wenn der Abstand zwischen Punkten im Vergleich zu ihrer Norm gering ist (in einem Verhältnis von ~ <1e-4 für Float 32 und ~ <1e-8 für Float64). Damit diese Situation eintreten kann, muss der Datensatz zunächst ziemlich dicht sein. Um einen Bestellfehler zu erhalten, müssen sich die beiden nächstgelegenen Punkte in nahezu gleicher Entfernung befinden. Darüber hinaus würden in diesem Fall aus ML-Sicht beide zu fast gleich guten Passungen führen.
In der obigen Situation können wir etwas tun, um die Häufigkeit dieser falschen Reihenfolge zu verringern (auf 0?). In der paarweisen Distanz argmin Situation. Wir können die Fehlordnung auf Punkte verschieben, die nicht am nächsten sind. Im Wesentlichen unter Verwendung der Tatsache, dass eine der Normen nicht erforderlich ist, um den Argmin zu finden, siehe Kommentar . Es hat 2 Vorteile. Es ist robuster (bis jetzt habe ich noch keine falsche Reihenfolge gefunden) und es ist noch schneller, weil es einige Berechnungen vermeidet.
Ein Nachteil, immer noch in der gleichen Situation, wenn wir am Ende die tatsächlichen Entfernungen zu den nächstgelegenen Punkten wollen, können die mit der obigen Methode berechneten Entfernungen nicht verwendet werden. Sie werden nur teilweise berechnet und sind sowieso nicht präzise. Wir müssen die Entfernungen von jedem Punkt zum nächstgelegenen Punkt neu berechnen. Dies ist jedoch schnell, da für jeden Punkt nur eine Entfernung berechnet werden muss.
Ich frage mich, was ich oben beschrieben habe, deckt den gesamten Anwendungsfall von euklidischen Abständen in sklearn ab. Aber ich schlage vor, dies überall dort zu tun, wo es angewendet werden kann. Dazu können wir euclidean_distances einen neuen Parameter hinzufügen, um nur den erforderlichen Teil zu berechnen, um ihn mit argmin zu verketten. Verwenden Sie es dann in pairwise_distances_argmin und in pairwise_distances_argmin_min (Berechnen Sie die tatsächlichen Mindestabstände am Ende in letzterem).
Wenn wir das nicht können, greifen Sie auf den langsamen, aber präzisen zurück oder fügen Sie einen Schalter wie in # 12136 hinzu.
Wir können versuchen, es ein wenig zu optimieren, um den Leistungsabfall zu verringern, da ich damit einverstanden bin, dass dies nicht optimal erscheint. Ich habe ein paar Ideen dafür.
Eine andere Möglichkeit, BLAS weiterhin zu verwenden, besteht darin, axpy mit nrm2 kombinieren, dies ist jedoch
Idealerweise möchten wir, dass die euklidische Distanz in BLAS enthalten ist ...
Schließlich gibt es noch eine andere Lösung, die im Upcasting besteht. Dies erfolgt in # 11271 für float32. Der Vorteil ist, dass die Geschwindigkeit nur halb so hoch ist wie die aktuelle und die Präzision erhalten bleibt. Das Problem für float64 wird jedoch nicht gelöst. Vielleicht können wir einen Weg finden, etwas Ähnliches in cython für float64 zu tun. Ich weiß nicht genau wie, aber ich benutze 2 float64 Zahlen, um einen float128 zu simulieren. Ich kann es versuchen, um zu sehen, ob es etwas machbar ist.
Idealerweise möchten wir, dass die euklidische Distanz in BLAS enthalten ist ...
Würden die Bibliotheken das in Betracht ziehen? Wenn OpenBLAS es tut, wären wir schon in einer ziemlich guten Situation ...
Was sind die genauen Unterschiede zwischen uns und der BLAS? Erkennen Sie die CPU-Fähigkeiten und entscheiden Sie, welche Implementierung verwendet werden soll, oder so ähnlich? Oder einfach nur Versionen für unterschiedlichere Architekturen kompiliert haben?
Oder einfach mehr Zeit / Energie für das Schreiben effizienter Implementierungen?
Dies ist interessant: eine alternative Implementierung der schnellen instabilen Methode, die jedoch behauptet, viel schneller als sklearn zu sein:
https://github.com/droyed/eucl_dist
(löst dieses Problem überhaupt nicht, obwohl lol)
Diese Diskussion scheint verwandt zu sein https://github.com/scipy/scipy/issues/5657
Hier ist, was Julia tut: https://github.com/JuliaStats/Distances.jl/blob/master/README.md#precision -for-euclidean-and-sqeuclidean
Hiermit kann ein Präzisionsschwellenwert festgelegt werden, um eine Neuberechnung zu erzwingen.
Beantwortung meiner eigenen Frage: OpenBLAS bietet für jeden Prozessor (keine Architektur!) Eine handgeschriebene Assembly und Heutistik, um Kernel für verschiedene Problemgrößen auszuwählen. Ich denke also nicht, dass es darum geht, es in Openblas zu bringen, sondern jemanden zu finden, der all diese Kernel schreibt / optimiert ...
Danke für die zusätzlichen Gedanken!
In einer Teilantwort,
Wir möchten uns für diese Art der Berechnung auf eine hochoptimierte Bibliothek verlassen, wie wir es für die lineare Algebra mit BLAS-Bibliotheken wie OpenBLAS oder MKL tun.
Ja, ich hatte auch gehofft, dass wir in BLAS mehr davon machen können. Das letzte Mal, als ich nichts in der Standard-BLAS-API gesehen habe, sieht es nah genug aus (aber dann bin ich kein Experte für diese). BLIS bietet möglicherweise mehr Flexibilität, aber da wir es nicht standardmäßig verwenden, ist es von begrenztem Nutzen (obwohl numpy eines Tages https://github.com/numpy/numpy/issues/7372 sein könnte).
Julia macht Folgendes: Sie ermöglicht das Festlegen eines Präzisionsschwellenwerts, um eine Neuberechnung zu erzwingen.
Gut zu wissen!
Sollten wir ein separates Problem für die oben verlinkte schnellere ungefähre Berechnung eröffnen? Scheint interessant
Ihre Beschleunigung der CPU von x2-x4 könnte auf https://github.com/scikit-learn/scikit-learn/pull/10212 zurückzuführen sein .
Ich würde lieber ein Thema auf scipy eröffnen, wenn wir diese Frage genug studiert haben, um dort eine vernünftige Lösung zu finden (und sie dann möglicherweise zurück zu portieren), da ich der Meinung bin, dass euklidische Distanz etwas Grundlegendes ist, das für viele Menschen außerhalb von ML von Interesse sein sollte (und gleichzeitig wäre es hilfreich, die Meinung der Menschen dort zu haben, z. B. zu Genauigkeitsfragen).
Es ist bis zu 60x, richtig?
Dies ist interessant: eine alternative Implementierung der schnellen instabilen Methode, die jedoch behauptet, viel schneller als sklearn zu sein
Ich bin mir da nicht sicher. Sie messen %timeit pairwise_distances(a,b, 'sqeuclidean') , wobei scipy's verwendet wird. Sie sollten %timeit pairwise_distances(a,b, 'euclidean', metric_params={'squared': True}) und ihre Beschleunigung wäre nicht so gut :)
Wie viel früher in der Diskussion gezeigt, kann sklearn 35x schneller sein als scipy
Ja, die Benchmarks sind nur ~ 30% besser mit metric="euclidean" (anstelle von squeclidean ).
In [1]: from eucl_dist.cpu_dist import dist
... import numpy as np
In [4]: rng = np.random.RandomState(1)
... a = rng.rand(1000, 300)
...b = rng.rand(1000, 300)
In [7]: from sklearn.metrics.pairwise import pairwise_distances
In [8]: %timeit pairwise_distances(a, b, 'sqeuclidean')
214 ms ± 2.06 ms per loop (mean ± std. dev. of 7 runs, 1 loop each)
In [9]: %timeit pairwise_distances(a, b)
27.4 ms ± 2.48 ms per loop (mean ± std. dev. of 7 runs, 10 loops each)
In [10]: from eucl_dist.cpu_dist import dist
In [11]: %timeit dist(a, b, matmul='gemm', method='ext', precision='float32')
20.8 ms ± 330 µs per loop (mean ± std. dev. of 7 runs, 10 loops each)
In [12]: %timeit dist(a, b, matmul='gemm', method='ext', precision='float64')
20.4 ms ± 222 µs per loop (mean ± std. dev. of 7 runs, 10 loops each)
Würden die Bibliotheken das in Betracht ziehen? Wenn OpenBLAS es tut, wären wir schon in einer ziemlich guten Situation ...
Klingt nicht einfach. BLAS ist eine Reihe von Spezifikationen für lineare Algebra-Routinen, für die es mehrere Implementierungen gibt. Ich weiß nicht, wie offen sie dafür sind, neue Funktionen hinzuzufügen, die nicht in den ursprünglichen Spezifikationen enthalten sind. Dafür wäre blis vielleicht offener, aber wie gesagt, es ist vorerst nicht die Standardeinstellung.
Geöffnet unter https://github.com/scikit-learn/scikit-learn/issues/12600 für die Behandlung von sqeuclidean vs euclidean in pairwise_distances .
Ich brauche Klarheit darüber, was wir dafür wollen. Wollen wir, dass pairwise_distances nahe beieinander liegt - im Sinne von all_close - sowohl für 'euklidisch' als auch für 'sqeuklidisch'?
Es ist ein bisschen schwierig. Da x nahe bei y liegt, bedeutet dies nicht, dass x² nahe bei y² liegt. Beim Quadrieren geht die Präzision verloren.
Die oben verlinkte Julia-Problemumgehung ist sehr interessant und einfach zu implementieren. Ich vermute jedoch, dass es für 'sqeuclidean' nicht wie erwartet funktioniert. Ich vermute, dass Sie den Schwellenwert weit unten einstellen müssen, um die gewünschte Präzision zu erzielen.
Das Problem beim Festlegen eines sehr niedrigen Schwellenwerts besteht darin, dass viele Neuberechnungen und ein enormer Leistungsabfall erforderlich sind. Dies wird jedoch durch die Dimension des Datensatzes gemindert. Der gleiche Schwellenwert löst viel weniger Neuberechnungen in hohen Dimensionen aus (Entfernungen sind größer).
Vielleicht können wir 2 Implementierungen haben und je nach Dimension des Datensatzes wechseln. Die langsame, aber sichere für niedrigdimensionale (in diesem Fall gibt es ohnehin keinen großen Unterschied zwischen scipy und sklearn) und die schnelle + Schwelle für hochdimensionale.
Dies erfordert einige Benchmarks, um herauszufinden, wann gewechselt werden muss, und um den Schwellenwert festzulegen, aber dies kann ein Hoffnungsschimmer sein :)
Hier sind einige Benchmarks für den Geschwindigkeitsvergleich zwischen scipy und sklearn. Die Benchmarks vergleichen sklearn.metrics.pairwise.euclidean_distances(X,X) mit scipy.spatial.distance.cdist(X,X) für Xs aller Größen. Die Anzahl der Proben reicht von 2⁴ (16) bis 2¹³ (8192), und die Anzahl der Merkmale reicht von 2⁰ (1) bis 2¹³ (8192).
Der Wert in jeder Zelle ist die Beschleunigung von sklearn gegenüber scipy, dh unter 1 sklearn ist langsamer und über 1 sklearn ist schneller.
Der erste Benchmark verwendet die MKL-Implementierung von BLAS und einen einzelnen Kern.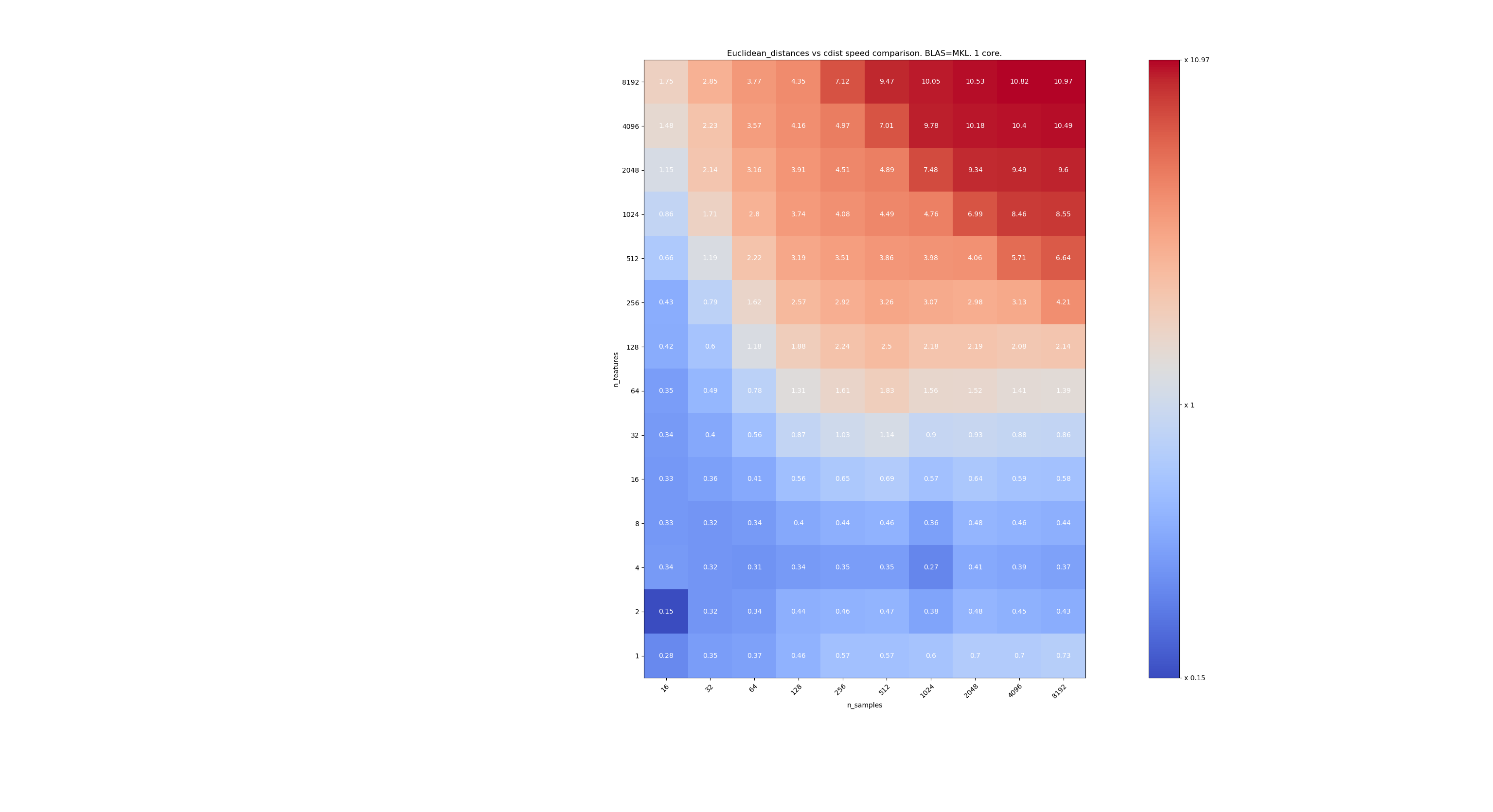
Der zweite verwendet die OpenBLAS-Implementierung von BLAS und einen einzelnen Kern. Es soll nur überprüft werden, ob sowohl MKL als auch OpenBLAS das gleiche Verhalten haben.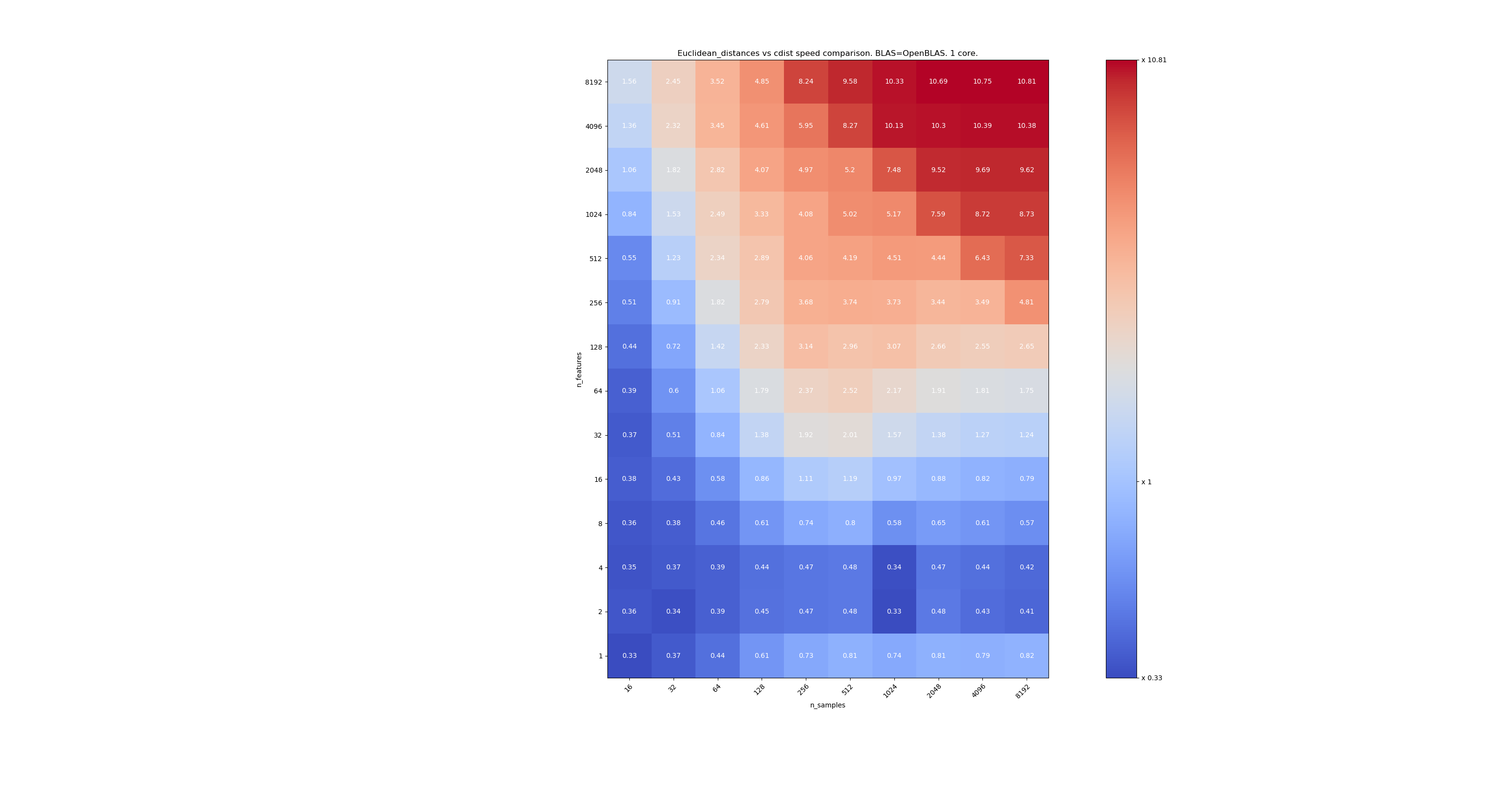
Der dritte verwendet die MKL-Implementierung von BLAS und 4 Kernen. Die Sache ist, dass euclidean_distances durch eine BLAS LEVEL 3-Funktion parallelisiert wird, aber cdist nur eine BLAS LEVEL 1-Funktion verwendet. Interessanterweise ändert es fast nichts an der Grenze.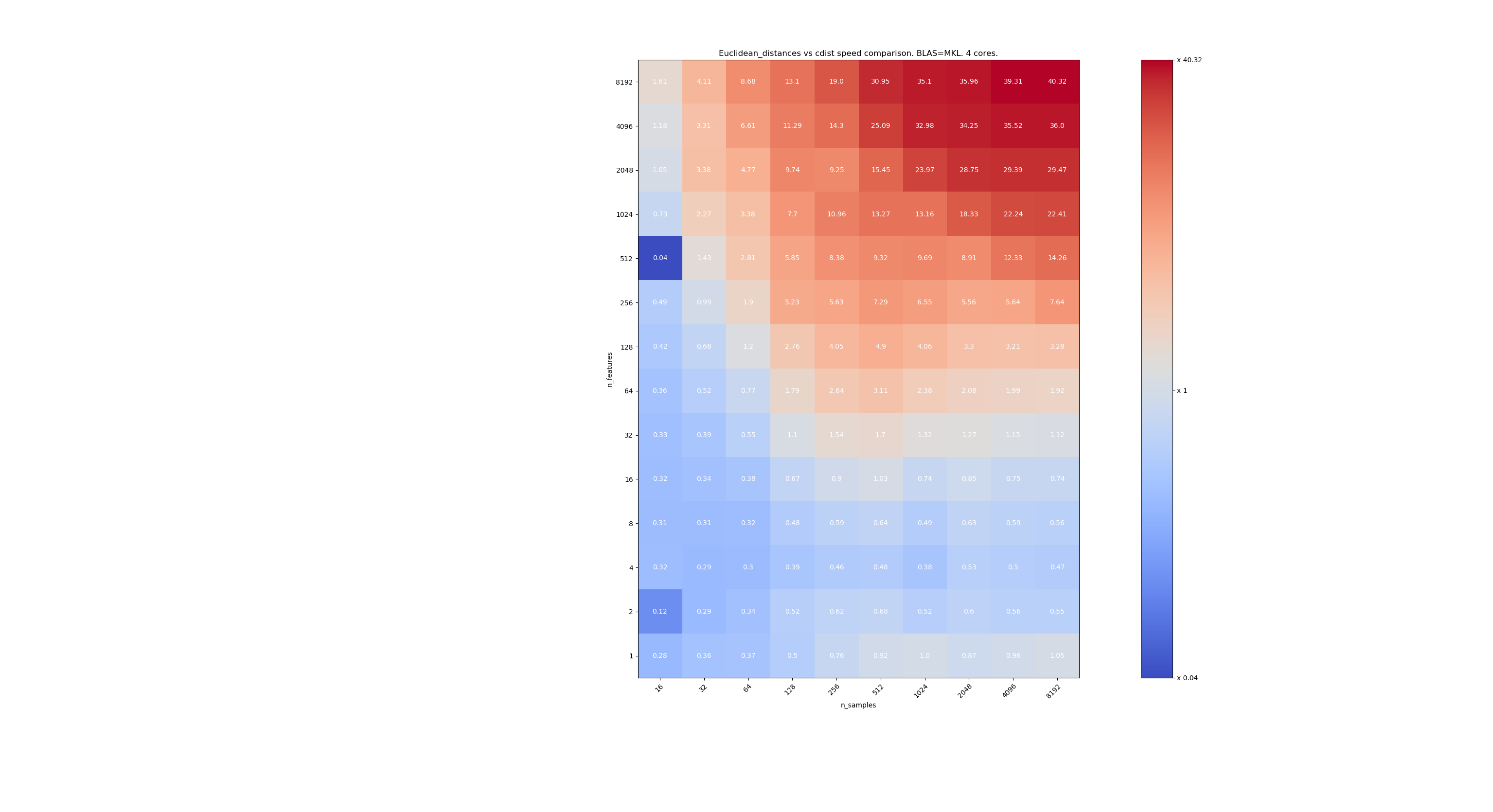
Wenn n_samples nicht zu niedrig ist (> 100), scheint die Grenze bei 32 Merkmalen zu liegen. Wir könnten uns entscheiden, cdist zu verwenden, wenn n_features <32 und euklidische_Distanzen, wenn n_features> 32 ist. Dies ist schneller und es gibt kein Präzisionsproblem. Dies hat auch den Vorteil, dass bei kleinen n_features die Julia-Schwelle zu vielen Neuberechnungen führt. Die Verwendung von cdist vermeidet dies.
Wenn n_features> 32 ist, können wir die Implementierung von euclidean_distances mit dem Julia-Schwellenwert aktualisieren. Das Hinzufügen des Schwellenwerts sollte euclidean_distances zu stark verlangsamen, da die Anzahl der Funktionen hoch genug ist, sodass nur wenige Neuberechnungen erforderlich sind.
@jeremiedbb super , danke für die analyse. Die Schlussfolgerung klingt für mich nach einem großartigen Weg nach vorne.
Oh, ich nehme an, das war alles für float64, oder? Was machen wir mit float32? immer ausgestoßen? Upcast für> 32 Funktionen?
Ich habe die Kommentare nicht sorgfältig durchgelesen (werde es bald tun), nur zu Ihrer Information, dass float64 seine Einschränkungen hat, siehe # 12128
@ qinhanmin2014 Ja, die Genauigkeit von float64 hat Einschränkungen, aber sie ist präzise genug, um zuverlässige fp32-Ergebnisse zu
Wie in den obigen Benchmarks zu sehen ist, ist selbst Multi-Core-BLAS im Allgemeinen nicht schneller. Dies scheint hauptsächlich für hochdimensionale Daten zu gelten (über 64 Dimensionen; davor ist der Nutzen IMHO normalerweise nicht die Mühe wert) - und da euklidische Abstände in dichten hochdimensionalen Daten nicht so zuverlässig sind, ist dieser Anwendungsfall IMHO nicht von höchster Bedeutung . Viele Benutzer haben weniger als 10 Dimensionen. In diesen Fällen scheint cdist normalerweise schneller zu sein?
Oh, ich nehme an, das war alles für float64, oder?
Eigentlich ist es sowohl für float32 als auch für float64 (ich meine sehr ähnlich). Ich schlage vor, immer cdist zu verwenden, wenn n_features <32 ist.
Die Frage ist, bei welchen Parametern ein Upcast auf fp64 tatsächlich billiger ist als die Verwendung von cdist von scipy.
Das Upcasting wird sich um den Faktor ~ 2 verlangsamen, also schätze ich um n_features = 64.
Viele Benutzer haben weniger als 10 Dimensionen.
Aber nicht jeder, deshalb müssen wir noch eine Lösung für hochdimensionale Daten finden.
Sehr schöne Analyse @jeremiedbb !
Für niedrigdimensionale Daten wäre es dann definitiv sinnvoll, cdist zu verwenden.
Auch FYI scipy des cdist upcasts float32 zu float64 https://github.com/scipy/scipy/issues/8771#issuecomment -384015674, ich bin nicht sicher , ob dies aufgrund Genauigkeit Fragen oder etwas anderes.
Insgesamt halte ich es für sinnvoll, den Parameter "algorithm" zu euclidean_distance hinzuzufügen, wie unter https://github.com/scikit-learn/scikit-learn/pull/12601#pullrequestreview -176076355 vorgeschlagen mit der Standardeinstellung "Keine", damit sie auch über eine globale Option wie unter https://github.com/scikit-learn/scikit-learn/pull/12136 festgelegt werden kann.
Es gibt auch einen interessanten Ansatz in Eigen3, um stabile Normen zu berechnen: https://eigen.tuxfamily.org/dox/StableNorm_8h_source.html (den ich noch nicht wirklich verstanden habe)
Gute Erklärung, verbesserte mein Verständnis
Wir haben beim Sprint keine Fortschritte gemacht und sollten es wahrscheinlich tun ... und
Ich kann remote beitreten, wenn Sie eine Zeit festlegen. Vielleicht am frühen Nachmittag?
Um die Situation zusammenzufassen,
Für Präzisionsprobleme bei euklidischen Entfernungsberechnungen
- Im niedrigdimensionalen Fall sollten wir, wie @jeremiedbb oben gezeigt hat, wahrscheinlich cdist verwenden
- im hochdimensionalen Fall und float32 könnten wir wählen zwischen,
- Chunking, Berechnung der Entfernung in 64-Bit und Verkettung
- In Fällen, in denen es um Präzision geht, auf cdist zurückzugreifen (wie ist eine offene Frage - z. B. nach Scipy zu greifen, kann nützlich sein https://github.com/scikit-learn/scikit-learn/issues/9354#issuecomment-438522881 )
Dann gibt es alle Probleme von Inkonsistenzen zwischen euklidisch, sqeuklidisch, minkowski usw.
In Bezug auf die Präzision unterhielten sich @jeremiedbb , @amueller und ich melkten Jeremie meistens nur wegen seines Fachwissens. Er ist der Meinung, dass wir uns nicht so sehr um die Instabilitätsprobleme in einem ML-Kontext in hohen Dimensionen in float64 kümmern müssen. Jeremie implizierte auch, dass es schwierig ist, einen effizienten Test dafür zu finden, ob gute Ergebnisse zurückgegeben wurden (vgl. # 12142).
Ich denke, wir sind zufrieden mit @rths vorherigem Kommentar mit dem Upcasting für float32. Da cdist auch auf float64 hochgespielt wird, könnten wir cdist erneut implementieren, um float32 zu verwenden (aber mit float64-Akkumulatoren?), Oder Chunking verwenden, wenn wir weniger kopieren in float32 mit niedrigem Dim möchten.
Möchte @Celelibi die PR in # 11271 ändern, oder sollte jemand anderes (einer von uns?) Eine vollständige Pull-Anfrage erstellen?
Und sobald dies behoben ist, sollten wir sqeuclidean und minkowski (p in {0,1}) dazu bringen, unsere Implementierungen zu verwenden. Wir haben keine Diskrepanz mit NearestNeighbors besprochen. Noch ein Sprint :)
Nach einer kurzen Diskussion im Sprint sind wir auf folgendem Weg gelandet:
im hochdimensionalen Fall (> 32 oder> 64 wählen Sie das Beste): Upcast von Chunks auf float64, wenn es float32 ist, und behalten Sie die 'schnelle' Methode bei. Für diese Art von Daten sind numerische Probleme auf float64 fast vernachlässigbar (ich werde Benchmarks dafür bereitstellen).
im niedrigdimensionalen Fall: Implementieren Sie die sichere Berechnung (anstatt wegen des Upcasts scipy cdist zu verwenden) in sklearn.
(Es ist verlockend, upcasting float32 auch in 0.20.3 zu werfen)
Hier sind einige Benchmarks für den Geschwindigkeitsvergleich zwischen scipy und sklearn.
[... schnipsen ...]
Das ist sehr interessant. Ich habe dieses Ergebnis eigentlich nicht erwartet. Ich habe Ihren Benchmark erneut durchgeführt und ein sehr ähnliches Ergebnis erzielt. Außer ich würde mich für eine niedrigere Entscheidungsgrenze einsetzen. Mein Benchmark würde 8 Funktionen vorschlagen.

Die Kosten für das Unrecht sind nicht symmetrisch. cdist ist nur für Berechnungen besser, die weniger als ein paar Sekunden dauern, und es wird sehr schnell langsam, wenn die Anzahl der Features zunimmt. Verwenden Sie daher im Zweifelsfall besser die BLAS-Implementierung.
Bearbeiten: Dieser Benchmark war für float64, aber ich finde auch, dass das Upcasting von float32-Matrizen auf float64 nur ein paar Prozent zur Gesamtzeit beiträgt und die Schlussfolgerung nicht ändert.
Mir ist aufgefallen, dass der Schwellenwert von der Maschine abhängt, auf der Sie die Benchmarks ausführen. Ich vermute, dass dies möglicherweise mit den AVX-Anweisungen zu tun hat. Ich stellte fest, dass die von mir veröffentlichten Benchmarks auf einem Computer ausgeführt wurden, der keine AVX2-Anweisungen hatte, sondern nur AVX. Und auf einem Computer mit AVX2 habe ich ähnliche Ergebnisse erzielt wie bei Ihnen.
Bei der Frage geht es jedoch nicht nur um die Leistung, sondern auch um die Präzision. Bei kleinen Abmessungen treten mit größerer Wahrscheinlichkeit Präzisionsprobleme auf. Vielleicht ist 16 ein guter Kompromiss. Was denken Sie ?
In Bezug auf diese Diskussion würde ich sagen, dass wir die Genauigkeit bewerten müssen, um eine fundierte Entscheidung zu treffen.
In Bezug auf Ihre PR sollte die Genauigkeit jedoch kein Problem mehr sein. Aber auf Kosten einer etwas teureren Berechnung. Daher sollte der Schwellenwert wahrscheinlich durch Benchmarking Ihrer PR festgelegt werden.
Die Genauigkeit des Benchmarking ist nicht so einfach. Weil die schwierigen Fälle nicht gleichmäßig verteilt werden.
Und es kann problematisch sein, wenn es in einem Eckfall unentdeckt passiert. Normalerweise möchten Sie eine garantierte numerische Genauigkeit innerhalb erschwinglicher CPU-Grenzen haben.
Wie an anderer Stelle erwähnt, sollte ein einzelnes Feature mit 10000000.01 und 10000000.00 ausreichen, um eine numerische Instabilität mit fp64 auszulösen, wenn die bekanntermaßen problematische Gleichung 10000 und 10001 mit fp32 verwendet wird. Versuchen Sie es mit 1024 Funktionen
>>> import sklearn.metrics.pairwise as sk, scipy.spatial.distance as sp
>>> X = [[10000.01] * 1024, [10000.00] * 1024]
>>> print(sk.euclidean_distances(X,X), "\n", sp.cdist(X,X))
[[ 0. 0.31895195]
[ 0.31895195 0. ]]
[[ 0. 0.32]
[ 0.32 0. ]]
(dies war unter Verwendung von 0.19.1) Der korrekte Abstand beträgt 0.32.
Wie Sie sehen können, verschlechtern sich die numerischen Instabilitäten mit der Anzahl der Features (es sei denn, Ihre Daten sind spärlich). Hier hat das Ergebnis mit FP64 eine Genauigkeit von weniger als zwei Stellen.
13410 behebt diesen speziellen Fall nicht. dh float64 + hohe Dimension.
Es behebt es jedoch für float32.
Wir haben jedoch beschlossen, Float64 + High Dim so zu belassen, wie es war, da die Genauigkeitsprobleme sehr unwahrscheinlich sind und nicht wirklich auf Anwendungsfälle für maschinelles Lernen zutreffen.
In Ihrem Beispiel haben X [0] und X [1] Normen gleich 320000.32 und 320000 und ihr Abstand beträgt 0,32, dh das 1e-6-fache ihrer Norm. Beim maschinellen Lernen sind die 16 signifikanten Ziffern (in float64) nicht alle relevant.
Wir haben jedoch beschlossen, Float64 + High Dim so zu belassen, wie es war, da die Genauigkeitsprobleme sehr unwahrscheinlich sind und nicht wirklich auf Anwendungsfälle für maschinelles Lernen zutreffen.
Ich wäre in diesem Fall moderater. Das Reduzieren der Dimensionalität ist ein üblicher erster Schritt in ML. Dafür kann MDS verwendet werden, und die euklidische Distanzmatrix wird stark genutzt.
Wenn jemand die Genauigkeit des float64-Gehäuses verbessern möchte, gibt es eine Möglichkeit, zwei Floats zu verwenden, um die Zwischenergebnisse darzustellen. Obwohl ich denke, dass es über den Rahmen des Scikit-Lernens hinausgeht.
ftp://ftp.math.ethz.ch/users/wpp/CELL/qd.pdf
Ich war nicht klar. Ich sage nicht, dass hochdimensionale Daten nicht für maschinelles Lernen gelten. Ich sage, dass die Art von Präzisionsproblemen, die in float64 auftreten, Punkte betrifft, deren Abstand 6 Größenordnungen kleiner ist als ihre Normen. Eine solche Präzision hat in einem realistischen Modell des maschinellen Lernens keine Bedeutung
Beim maschinellen Lernen sind die 16 signifikanten Ziffern (in float64) nicht alle relevant.
Ich bin überhaupt nicht davon überzeugt, dass dies allgemein so ist.
In diesem Beispiel haben wir 15 von 16 Stellen an Genauigkeit verloren. Ich würde zustimmen, wenn wir die Hälfte der Präzision verwenden würden, aber wir haben keine solche Beziehung. Der Verlust durch Downcasting von FP64 auf FP32 kann aufgrund der Messgenauigkeit häufig tolerierbar sein. GPUs für Endverbraucher sind mit FP32 viel schneller als beispielsweise mit FP64 (in einigen Fällen erlauben sie jetzt jedoch FP32-Daten und FP64-Akkumulatoren), und für die Inferenz neuronaler Netze wird möglicherweise sogar int8 angezeigt. Das gilt aber nicht überall.
Zum Beispiel in k-means Clustering besteht die Annahme , dass Cluster im Wesentlichen in ihren Mitteln unterscheiden (und dass wir nicht wissen , die Mittel vorher) und somit haben wir einen Verlust an Präzision hier. Wenn Sie viele Cluster haben, können einige ihrer Normen im Vergleich zu ihrer Trennung groß sein.
Darüber hinaus sind es nach den ersten anfänglichen Iterationen häufig kleine Entfernungsunterschiede, die dazu führen, dass ein Punkt zu einem anderen Cluster wechselt. Ein Genauigkeitsverlust kann hier die Ergebnisse beeinträchtigen und zu Instabilität führen.
Betrachten Sie nun k-means für Zeitreihenfragmente mit vielen Variablen.
Mit zunehmender Datengröße müssen wir davon ausgehen, dass die Abstände zum nächsten Nachbarn kleiner werden. Wenn Ihre Normen nicht 0 sind, sind sie möglicherweise kleiner als die Vektornormen und verursachen Probleme. Dies wird wahrscheinlich mit zunehmender Datensatzgröße schwerwiegender. Der Fluch der Dimensionalität besagt, dass die größten und die kleinsten Entfernungen immer ähnlicher werden; Um die korrekte Rangfolge der nächsten Nachbarn zu berechnen, benötigen wir möglicherweise eine gute Genauigkeit bei hochdimensionalen Daten. Im 20news-Datensatz liegt der kleinste Abstand ungleich Null bei etwa 0,02 (die Normen sind alle 1). Aber das sind nur 10.000 Instanzen und ziemlich unterschiedliche Inhalte. Nehmen wir nun an, der Datensatz befasste sich stattdessen mit nahezu doppelter Erkennung ...
Ich wäre mir nicht sicher, ob dies "unwahrscheinlich" in ML passiert ... natürlich wird es nicht jeden betreffen.
Wenn ich sage "Beim maschinellen Lernen sind die 16 signifikanten Stellen (in float64) nicht alle relevant.", Spreche ich nicht von der berechneten Entfernung, sondern von den Daten X.
Beim maschinellen Lernen stammen Ihre Daten aus einem Maß, und es gibt kein Maß, das auf die 9. Stelle genau ist (außer sehr wenigen in der Teilchenphysik).
Wie würden Sie in Ihrem Beispiel für 10000000.01 und 10000000.00 einem Abstand von 0,01 eine gewisse Bedeutung beimessen, wenn Ihre Unsicherheit über die Werte von X viel größer ist?
Für KMeans gibt es zunächst eine Möglichkeit, einen großen Teil der Präzisionsverluste zu überwinden. Wenn Sie nach dem nächstgelegenen Zentrum einer Beobachtung x suchen, müssen Sie der Entfernungsberechnung nicht die Norm x hinzufügen, wodurch die katastrophale Stornierung in den meisten Fällen vermieden wird.
Dann gruppieren sich km-Cluster basierend auf euklidischen Entfernungen. Sie wissen jedoch nicht, ob Ihre Daten genau so erfasst werden. Tatsächlich besteht eine Wahrscheinlichkeit von 0, dass Ihre Daten auf diese Weise geclustert werden. Kmeans gibt eine Schätzung darüber, wie Ihre Daten geclustert werden könnten, und Punkte, die an der Grenze von zwei Clustern liegen, können definitiv nicht mit Sicherheit als zu dem einen oder anderen gehörend angesehen werden. Wie interpretieren Sie einen Punkt im gleichen Abstand von 2 Clustern? Meins ist, dass entweder die 2 Cluster nur ein Cluster sein sollten oder KMeans nicht das beste Algo ist, um meine Daten zu clustern (oder sogar kmeans gibt mir eine ziemlich gute Vorstellung davon, wie meine Daten geclustert werden, aber ich weiß, dass Grenzen von Clustern nicht relevant sind).
Die Verwendung von nur "| b | ^ 2-2ab" hat keine katastrophale Stornierung - aber den gleichen Genauigkeitsverlust bei den Ziffern, die den Unterschied ausmachen. Die Ergebnisse sind die gleichen, als ob Sie anschließend jeder Entfernung die Norm a hinzugefügt hätten. Wenn die Abstände viel kleiner als die Norm von a sind, entsteht ein Genauigkeitsverlust, der vermieden werden kann, wenn die Berechnungen auf herkömmliche Weise ohne BLAS-Hacks durchgeführt werden.
Sie können das numerische Problem also NICHT auf diese Weise überwinden!
K-means ist ein Optimierungsproblem. Solche Hacks können also bedeuten, dass sklearn nur schlechtere Lösungen findet als andere Tools. Und wie bereits erwähnt, kann dies auch zu Instabilitäten führen. Im schlimmsten Fall könnte dies dazu führen, dass sklearn kmeans dieselben Zustände durchlaufen, bis max_iter ohne Verbesserung (unter der Annahme von tol = 0, wenn Sie ein lokales Optimum finden möchten), was theoretisch unmöglich wäre.
Bis k-means konvergiert hat, kann man nicht viel über Punkte mit dem "gleichen" Abstand zu zwei Clustern sagen. Bei der nächsten Iteration haben sich die Mittel möglicherweise bewegt und der Unterschied könnte viel größer und wichtiger werden!
Ich bin kein großer Fan von k-means, weil es bei verrauschten Daten nicht so gut funktioniert. Es gibt jedoch Variationen, die solche Fälle besser behandeln. Aber trotzdem, wenn Sie es verwenden, sollten Sie wahrscheinlich versuchen, die volle Qualität zu erhalten (weshalb ich auch immer tol=0 ) und es nicht schlechter als nötig machen. Es ist billig genug, um die richtigen Berechnungen durchzuführen (und wie bereits erwähnt, verschlimmern sich die Probleme mit der Datengröße. Bei kleinen Daten spielt die langsamere Laufzeit normalerweise keine Rolle, bei größeren Datenmengen wird die Genauigkeit wahrscheinlich wichtiger).
Je nach Anwendung kann die Differenz zwischen 10000000.01 und 10000000.00 kann Materie. Und wie ich bereits gezeigt habe, treten die Probleme früher auf, wenn Sie mehrere Funktionen verwenden. Mit fp32 von nur 10000 und 10001 mit einem einzigen Feature und 100 vs. 101 mit 100 Features denke ich:
Wie bereits erwähnt, kann der Mittelwert eine physikalische Bedeutung haben, die Sie nicht verlieren möchten. Wenn Sie Daten mit Temperaturen in Kelvin haben, möchten Sie diese nicht 0: 1 skalieren oder zentrieren. das würde deine Verhältnisskala ruinieren. Wenn Sie nun beispielsweise Zeitreihen der Temperatur eines Stahlprodukts beim Abkühlen vergleichen und herausfinden möchten, ob der Abkühlprozess die Zuverlässigkeit Ihres Stahlprodukts beeinträchtigt. Möglicherweise haben Sie Temperaturen von über 700 K, und die Zeitreihen enthalten möglicherweise Hunderte von Datenpunkten, wenn Sie den Abklingprozess analysieren möchten. Selbst mit nur 5 Stellen Eingabegenauigkeit (0,01 K) mit der Länge der Zeitreihe kann das numerische Problem auftreten. Möglicherweise erhalten Sie erneut nur 1-2 Ziffern im Ergebnis. Ich glaube nicht, dass Sie einfach ausschließen können, dass Präzision in ML jemals wichtig ist, wenn Sie diese katastrophale Wirkung haben. Es ist etwas anderes, wenn Sie garantieren könnten, immer 10 von 16 Stellen Genauigkeit zu erhalten. Hier können Sie das nicht tun, im schlimmsten Fall haben Sie möglicherweise 0 Ziffern (deshalb ist es katastrophal).
Beim maschinellen Lernen stammen Ihre Daten aus einem Maß, und es gibt kein Maß, das auf die 9. Stelle genau ist (außer sehr wenigen in der Teilchenphysik).
Die Rohwerte aus der realen Welt haben selten diese Genauigkeit, das ist richtig. Aber ML ist nicht auf diese Art von Eingabe beschränkt. Möglicherweise möchten Sie ML auf mathematische Probleme anwenden, z. B. auf MDS in der Grafik eines würfelförmigen Puzzles eines Rubiks oder auf die Zusammenfassung der erfolgreichen Strategien, die Ihr Schwarm von RL-Agenten gefunden hat, die Pacman spielen.
Selbst wenn die ursprüngliche Informationsquelle die reale Welt ist, kann es zu einer Zwischenverarbeitung kommen, bei der die meisten Ziffern für den Clustering-Algorithmus relevant sind. Wie das Ergebnis eines Gradientenabfalls auf eine Funktion, deren Parameter in der realen Welt statistisch erfasst werden.
Ich frage mich eigentlich, warum wir das immer noch diskutieren. Ich denke, wir sind uns alle einig, dass Scikit-Learn sein Bestes in Bezug auf die Kompromissgenauigkeit gegenüber der Rechenzeit geben sollte. Und wer mit dem aktuellen Status nicht zufrieden ist, sollte eine Pull-Anfrage einreichen.
Die Verwendung von nur "| b | ^ 2-2ab" hat keine katastrophale Stornierung - aber den gleichen Genauigkeitsverlust bei den Ziffern, die den Unterschied ausmachen. Die Ergebnisse sind die gleichen, als ob Sie anschließend jeder Entfernung die Norm a hinzugefügt hätten. Wenn die Abstände viel kleiner als die Norm von a sind, entsteht ein Genauigkeitsverlust, der vermieden werden kann, wenn die Berechnungen auf herkömmliche Weise ohne BLAS-Hacks durchgeführt werden.
Sie können das numerische Problem also NICHT auf diese Weise überwinden!
Es gibt einen Genauigkeitsverlust, der jedoch keine katastrophale Stornierung verursachen kann (zumindest wenn a und b nahe beieinander liegen), und Sie können zeigen, dass der relative Fehler in der Entfernung (die keine Entfernung ist) gering bleibt.
Bei KMeans, bei denen Sie nur das nächstgelegene Zentrum finden möchten, haben Sie genügend Präzision, um die Bestellung richtig zu halten. Wenn Sie am Ende die Trägheit wünschen, können Sie einfach die Abstände jedes Punkts zu seinem Clusterzentrum mit der genauen Formel berechnen.
Außerdem ist KMeans kein konvexes Optimierungsproblem. Selbst wenn Sie es bis zur Konvergenz mit tol = 0 laufen lassen, landen Sie in lokalen Minima, die weit von den globalen Minima entfernt sein können (selbst bei der Initialisierung von kmeans ++). Daher würde ich lieber viele Kilometer mit unterschiedlichen Init und einer relativ geringen Anzahl von Iterationen laufen. Sie haben bessere Chancen, in einem besseren lokalen Minimum zu landen. Dann können Sie den besten bis zur Konvergenz erneut ausführen.
Der relative Fehler im Vergleich zur tatsächlichen Entfernung kann beliebig groß sein und daher falsche nächste Nachbarn verursachen. Betrachten Sie den Fall, in dem | a | ² = | b | ² = 1 ist, beispielsweise für tf-idf. Angenommen, die Vektoren sind sehr nahe. Dann ist ab auch nahe bei 1, und zu diesem Zeitpunkt haben Sie bereits viel von Ihrer Präzision verloren.
Wie ich oben geschrieben habe, ist der Fehler vorhanden, auch wenn Sie keine katastrophale Stornierung haben. Betrachten Sie 8 Stellen Genauigkeit. Die tatsächliche Entfernung kann 0,000012345678 betragen und kann mit FP32 und der regulären euklidischen Entfernung mit acht Ziffern berechnet werden. Mit dieser Gleichung berechnen Sie jedoch stattdessen den Wert ab = 0,99998765432, der mit FP32 bestenfalls auf ungefähr 0,99998765 abgeschnitten wird, sodass Sie in diesem Beispiel unnötig drei Stellen Genauigkeit verloren haben. Der Verlust ist so groß wie im Katastrophenfall. Wenn die Abstände viel kleiner als die Normen sind, kann Ihre Präzision bei diesem Ansatz beliebig schlecht werden.
Ja, kmeans ist nicht konvex. Aber dann möchten Sie zumindest ein lokales Optimum finden und nicht stecken bleiben (oder sogar schwingen, weil sich die resultierenden Fehler unregelmäßig verhalten), weil Ihre Präzision zu niedrig ist. So haben Sie zumindest die Möglichkeit , die globale in gut erzogenen Fällen und mit mehreren Versuchen zu finden.
Ich schätze diese Diskussion, aber was wir wirklich brauchen, ist eine Lösung, die nicht schlechter ist als das, was wir getan haben, bevor wir aufgehört haben, Dinge auf float64 zu übertragen. In diesem Sinne war die Upcasting -Lösung von @Celelibi ausreichend. Die Verwendung der exakten Lösung in geringen Dimensionen ist eine zusätzliche Verbesserung gegenüber dem, was wir früher getan haben.
Haben Sie in Bezug auf eine zukünftige Version mehr Vertrauen, um effizient zu erkennen, wann wir die genaue Berechnung in hohen Dimensionen in Betracht ziehen könnten?
Ich habe einen Benchmark durchgeführt, um die durchschnittliche Genauigkeit des float64-Falls mit Zufallszahlen zu bewerten. Ich vergleiche 3 Algorithmen: neumaier_sum((x-y)**2) , numpy.sum((x-y)**2) und X2 - 2*X.dot(Y.T) + Y2.T . Das genaue zu vergleichende Ergebnis wurde mit mpmath mit einer Genauigkeit von 256 Bit erhalten.
X und Y haben 100 Samples und eine variable Anzahl von Features und sind mit Zufallszahlen zwischen -2 und 2 gefüllt.
Auf dem folgenden GIF befindet sich ein Bild pro Feature-Nummer (zwischen 1 und 200). Auf jedem Bild repräsentiert jeder Punkt den relativen Fehler des quadratischen euklidischen Abstands zwischen einem der 10000 Vektorpaare von X und Y . Der relative Fehler wird zur besseren Lesbarkeit mit 2 ^ 53 multipliziert, was in etwa der ULP-Einheit entspricht.
Die obigen Kurven sind die ungefähre Verteilung (unter Verwendung einer Kernel-Dichteschätzung).

Beachten Sie, dass die Diagramme zur besseren Lesbarkeit bei 6 ULP geschnitten wurden. Es zeigt den Durchschnittsfall, nicht den schlimmsten Fall. Der Fehler der erweiterten Formel kann ziemlich groß werden.
Meine Analyse dieses Ergebnisses ist, dass der relative Fehler der erweiterten Formel im Durchschnitt mit wenigen Merkmalen sehr groß sein kann, aber schnell dem der Differenz und der Zahlensumme ähnlich wird. Der Schwellenwert liegt zwischen 5 und 10 Merkmalen.
Ich versuche derzeit auch, eine Obergrenze für den Fehler der erweiterten Formel sowie pathologische Beispiele zu finden.
Ich denke, @ kno10 ist besorgt, dass wir oft an Fällen interessiert sind, in denen
Punkte sind nicht zufällig verteilt, sondern liegen nahe beieinander oder haben eine Einheit
Norm.
In der Tat, aber ich musste überzeugt sein, dass es in der Praxis keine vollständige BS ist. ^^
Um den obigen Kommentar zu vervollständigen: Der relative Fehler der Formel x²+y²-2ab scheint unbegrenzt zu sein. Wenn meine Analyse nicht falsch ist, kann der relative Fehler bis zu 2^(52*2) betragen, wenn x und y nahe beieinander liegen. Zumindest theoretisch. In der Praxis ist der schlimmste Fall, den ich gefunden habe, ein relativer Fehler von 2^52+1 .
>>> a, b = (0xfffffec4d6282+1) * 2.0**(511-52), 0xfffffec4d6282 * 2.0**(511-52)
>>> a, b
(6.703903473040778e+153, 6.7039034730407766e+153)
>>> exactdiff = (a-b)**2
>>> exactdiff
2.2158278651204453e+276
>>> computeddiff = a**2 + b**2 - 2*a*b
>>> computeddiff
-9.9792015476736e+291
>>> abs((computeddiff - exactdiff) / exactdiff)
4503599627370497.0
>>> bin(int(abs((computeddiff - exactdiff) / exactdiff)))
'0b10000000000000000000000000000000000000000000000000001'
Das Umdrehen des Ergebnisses des Ergebnisses würde es tatsächlich der Wahrheit näher bringen. Dies ist das dramatischste Beispiel, das ich finden konnte, aber das Ändern des Exponenten in den Werten von a und b ändert den relativen Fehler nicht.
>>> a, b = (0xfffffec4d6282+1) * 2.0**(-52), 0xfffffec4d6282 * 2.0**(-52)
>>> a, b
(0.9999999266202912, 0.999999926620291)
>>> exactdiff = (a-b)**2
>>> computeddiff = a**2 + b**2 - 2*a*b
>>> abs((computeddiff - exactdiff) / exactdiff)
4503599627370497.0
Ich denke, ein Histogrammplot in ULPs wäre sinnvoller als die obige Animation mit der Fehlerverteilung innerhalb des ULP. 0 ULP-Fehler und 1 ULP-Fehler sind also "so gut wie es nur geht". 2 ULP ist aufgrund des sqrt wahrscheinlich unvermeidbar. Ich nehme an, dass es sich lohnt, größere Fehler zu untersuchen.
Die Verwendung von (computed - exact) / exact ist sinnvoll, solange genau genau groß ist. Sobald wir jedoch numerische Herausforderungen für den genauen Wert erhalten, wird dies ziemlich instabil. In solchen Fällen kann es sich lohnen, stattdessen (computed-exact)/norm zu verwenden, dh die Genauigkeit unserer Entfernungsberechnungen im Vergleich zu den Eingabedaten und nicht im Vergleich zu den abgeleiteten Entfernungen zu betrachten.
Wenn wir zwei eindimensionale Werte haben, die sich nur um 1 ULP unterscheiden, kann der Fehler von 2 ULP sehr groß erscheinen. Wir sind jedoch bereits bei der Auflösung der Eingabedaten, sodass die Ergebnisse ziemlich instabil sind.
Beachten Sie, dass bei mehreren Dimensionen die Eingabedaten möglicherweise eine höhere Auflösung aufweisen.
Betrachten Sie Eingabedaten vom Typ (1, 1e-16) vs. (1, 2e-16) . Wenn wir beispielsweise ein konstantes Attribut in den Eingabedaten haben, beispielsweise ein weißes Pixel in MNIST.
Mit der differenzbasierten Gleichung wird dies in Ordnung sein, aber die Punktversion gerät in Schwierigkeiten, nicht wahr? Aus diesem Grund reichen eindimensionale Experimente möglicherweise nicht aus, um dies zu untersuchen.
Ich denke, ein Histogrammplot in ULPs wäre sinnvoller als die obige Animation mit der Fehlerverteilung innerhalb des ULP.
Ich bin mir nicht sicher, wie Sie es dargestellt hätten. Es würde ein Histogramm pro Anzahl von Merkmalen und pro Algorithmus geben. Neben einem 3D-Plot oder einer Animation kann ich nicht viel tun.
Die Verwendung von
(computed - exact) / exactist sinnvoll, solange genau genau groß ist. Sobald wir jedoch numerische Herausforderungen für den genauen Wert erhalten, wird dies ziemlich instabil.
Ich bin mir nicht sicher, was Sie in diesem Zusammenhang unter instabil verstehen. Der genaue Wert sollte mit allem berechnet werden, was erforderlich ist, um ihn genau zu machen.
(Apropos, ich hätte den relativen Fehler auch in meinem Plot mit willkürlicher Genauigkeit berechnen sollen, anstatt ihn mit dem genau gerundeten Ergebnis zu vergleichen. Ich habe meinen Plot aktualisiert, die seltsamen Wellen sind verschwunden.)
In solchen Fällen kann es sich lohnen, stattdessen
(computed-exact)/normzu verwenden, dh die Genauigkeit unserer Entfernungsberechnungen im Vergleich zu den Eingabedaten und nicht im Vergleich zu den abgeleiteten Entfernungen zu betrachten.
Wenn ich Ihre Idee richtig verstehe, möchten Sie den absoluten Fehler lieber mit der Größe der Eingabedaten vergleichen. Verwendung der Vektornormen als aggregiertes Maß für die Größe der Eingaben. Während der relative Standardfehler ihn mit der Größe des exakten Ergebnisses vergleicht.
Ich denke, mit dieser Metrik versuchen Sie zu erfassen, wie fehlerhaft ein Algorithmus ist. Aber es scheint aus einigen Gründen nicht besonders nützlich zu sein.
- Es sagt nicht wirklich aus, wie viele Ziffern des Ergebnisses ungenau sind.
- Tatsächlich hätten die meisten Algorithmen eine Punktzahl von weniger als 1e-15. Sogar die erweiterte Formel (punktbasierter Algorithmus) hätte eine Punktzahl, die durch etwa 5 ULP (Eingabe) begrenzt ist (grobe Schätzung, ich habe nicht den vollständigen Beweis geschrieben).
- Und da beide Metriken nur eine neu skalierte Version des absoluten Fehlers
computed - exact, würden sie die Algorithmen in derselben Reihenfolge einordnen, wenn sie an denselben Eingaben ausgewertet werden.
Es ist also dasselbe wie der übliche relative Fehler, nur mit einer weniger nützlichen Wertinterpretation (IMO).
Betrachten Sie Eingabedaten vom Typ
(1, 1e-16)vs.(1, 2e-16). Wenn wir beispielsweise ein konstantes Attribut in den Eingabedaten haben, beispielsweise ein weißes Pixel in MNIST.
Mit der differenzbasierten Gleichung wird dies in Ordnung sein, aber die Punktversion gerät in Schwierigkeiten, nicht wahr? Aus diesem Grund reichen eindimensionale Experimente möglicherweise nicht aus, um dies zu untersuchen.
Der punktbasierte Algorithmus hätte einen relativen Fehler von 1 , was bedeutet, dass der Fehler so groß ist wie das genaue Ergebnis und daher keine Ziffer des Ergebnisses korrekt ist. Und Ihre Metrik hätte einen Wert von 1e-16 was bedeutet, dass relativ zur Skala der Vektornorm nur die 16. Ziffer deaktiviert ist.
Ich bin mir nicht sicher, was Sie mit diesem Beispiel zeigen wollen.
Wenn wir uns immer noch Sorgen über die Genauigkeit von euklidischen Abständen mit float64 machen, ist es wahrscheinlich besser, diese Diskussion in einer neuen Ausgabe zusammenzufassen, da es hier 100 Kommentare gibt.
Hilfreichster Kommentar
Die Rohwerte aus der realen Welt haben selten diese Genauigkeit, das ist richtig. Aber ML ist nicht auf diese Art von Eingabe beschränkt. Möglicherweise möchten Sie ML auf mathematische Probleme anwenden, z. B. auf MDS in der Grafik eines würfelförmigen Puzzles eines Rubiks oder auf die Zusammenfassung der erfolgreichen Strategien, die Ihr Schwarm von RL-Agenten gefunden hat, die Pacman spielen.
Selbst wenn die ursprüngliche Informationsquelle die reale Welt ist, kann es zu einer Zwischenverarbeitung kommen, bei der die meisten Ziffern für den Clustering-Algorithmus relevant sind. Wie das Ergebnis eines Gradientenabfalls auf eine Funktion, deren Parameter in der realen Welt statistisch erfasst werden.
Ich frage mich eigentlich, warum wir das immer noch diskutieren. Ich denke, wir sind uns alle einig, dass Scikit-Learn sein Bestes in Bezug auf die Kompromissgenauigkeit gegenüber der Rechenzeit geben sollte. Und wer mit dem aktuellen Status nicht zufrieden ist, sollte eine Pull-Anfrage einreichen.