Scikit-learn: الدقة العددية للمسافات الإقليدية ذات العوامة 32
وصف
لقد لاحظت أن وظيفة sklearn.metrics.pairwise.pairwise_distances تتفق مع np.linalg.norm عند استخدام مصفوفات np.float64 ، لكنني لا أوافق عند استخدام مصفوفات np.float32. انظر مقتطف الرمز أدناه.
خطوات / كود الاستنساخ
import numpy as np
import scipy
import sklearn.metrics.pairwise
# create 64-bit vectors a and b that are very similar to each other
a_64 = np.array([61.221637725830078125, 71.60662841796875, -65.7512664794921875], dtype=np.float64)
b_64 = np.array([61.221637725830078125, 71.60894012451171875, -65.72847747802734375], dtype=np.float64)
# create 32-bit versions of a and b
a_32 = a_64.astype(np.float32)
b_32 = b_64.astype(np.float32)
# compute the distance from a to b using numpy, for both 64-bit and 32-bit
dist_64_np = np.array([np.linalg.norm(a_64 - b_64)], dtype=np.float64)
dist_32_np = np.array([np.linalg.norm(a_32 - b_32)], dtype=np.float32)
# compute the distance from a to b using sklearn, for both 64-bit and 32-bit
dist_64_sklearn = sklearn.metrics.pairwise.pairwise_distances([a_64], [b_64])
dist_32_sklearn = sklearn.metrics.pairwise.pairwise_distances([a_32], [b_32])
# note that the 64-bit sklearn results agree exactly with numpy, but the 32-bit results disagree
np.set_printoptions(precision=200)
print(dist_64_np)
print(dist_32_np)
print(dist_64_sklearn)
print(dist_32_sklearn)
نتائج متوقعة
أتوقع أن النتائج من sklearn.metrics.pairwise.pairwise_distances ستتفق مع np.linalg.norm لكل من 64 بت و 32 بت. بمعنى آخر ، أتوقع الناتج التالي:
[ 0.0229059506440019884643266578905240749008953571319580078125]
[ 0.02290595136582851409912109375]
[[ 0.0229059506440019884643266578905240749008953571319580078125]]
[[ 0.02290595136582851409912109375]]
النتائج الفعلية
ينتج عن مقتطف الشفرة أعلاه الإخراج التالي بالنسبة لي:
[ 0.0229059506440019884643266578905240749008953571319580078125]
[ 0.02290595136582851409912109375]
[[ 0.0229059506440019884643266578905240749008953571319580078125]]
[[ 0.03125]]
إصدارات
Darwin-16.6.0-x86_64-i386-64bit
('Python', '2.7.11 | 64-bit | (default, Jun 11 2016, 03:41:56) \n[GCC 4.2.1 Compatible Apple LLVM 6.0 (clang-600.0.57)]')
('NumPy', '1.11.3')
('SciPy', '0.19.0')
('Scikit-Learn', '0.18.1')
ال 102 كومينتر
نفس النتائج مع Python 3.5:
Darwin-15.6.0-x86_64-i386-64bit
Python 3.5.1 (v3.5.1:37a07cee5969, Dec 5 2015, 21:12:44)
[GCC 4.2.1 (Apple Inc. build 5666) (dot 3)]
NumPy 1.11.0
SciPy 0.18.1
Scikit-Learn 0.17.1
يحدث فقط مع مسافة إقليدية ويمكن إعادة إنتاجه باستخدام sklearn.metrics.pairwise.euclidean_distances :
import scipy
import sklearn.metrics.pairwise
# create 64-bit vectors a and b that are very similar to each other
a_64 = np.array([61.221637725830078125, 71.60662841796875, -65.7512664794921875], dtype=np.float64)
b_64 = np.array([61.221637725830078125, 71.60894012451171875, -65.72847747802734375], dtype=np.float64)
# create 32-bit versions of a and b
a_32 = a_64.astype(np.float32)
b_32 = b_64.astype(np.float32)
# compute the distance from a to b using sklearn, for both 64-bit and 32-bit
dist_64_sklearn = sklearn.metrics.pairwise.euclidean_distances([a_64], [b_64])
dist_32_sklearn = sklearn.metrics.pairwise.euclidean_distances([a_32], [b_32])
np.set_printoptions(precision=200)
print(dist_64_sklearn)
print(dist_32_sklearn)
لم أستطع تعقب الخطأ أكثر.
آمل أن يكون هذا يمكن أن تساعد.
قد يستخدم numpy تراكمًا عالي الدقة. نعم ، يبدو مثل هذا
يستحق الإصلاح.
في 19 يوليو 2017 ، الساعة 12:05 صباحًا ، كتب "nvauquie" [email protected] :
نفس النتائج مع Python 3.5:
داروين 15.6.0-x86_64-i386-64bit
Python 3.5.1 (v3.5.1: 37a07cee5969 ، 5 كانون الأول (ديسمبر) 2015 ، 21:12:44)
[GCC 4.2.1 (Apple Inc. build 5666) (نقطة 3)]
NumPy 1.11.0
SciPy 0.18.1.0 تحديث
Scikit-Learn 0.17.1.1 تحديثيحدث فقط مع مسافة إقليدية ويمكن إعادة إنتاجه باستخدام
مباشرة sklearn.metrics.pairwise.euclidean_distances:استيراد scipy
استيراد sklearn.metrics.pairwiseإنشاء متجهات 64 بت a و b متشابهة جدًا مع بعضها البعض
a_64 = np.array ([61.221637725830078125 ، 71.60662841796875 ، -65.7512664794921875] ، dtype = np.float64)
b_64 = np.array ([61.221637725830078125 ، 71.60894012451171875 ، -65.72847747802734375] ، dtype = np.float64)إنشاء إصدارات 32 بت من a و b
a_32 = a_64.astype (np.float32)
b_32 = b_64.astype (np.float32)احسب المسافة من a إلى b باستخدام sklearn ، لكل من 64 بت و 32 بت
dist_64_sklearn = sklearn.metrics.pairwise.euclidean_distances ([a_64]، [b_64])
dist_32_sklearn = sklearn.metrics.pairwise.euclidean_distances ([a_32]، [b_32])np.set_printoptions (الدقة = 200)
طباعة (dist_64_sklearn)
طباعة (dist_32_sklearn)لم أستطع تعقب الخطأ أكثر.
آمل أن يكون هذا يمكن أن تساعد.-
أنت تتلقى هذا لأنك مشترك في هذا الموضوع.
قم بالرد على هذا البريد الإلكتروني مباشرة ، وقم بعرضه على GitHub
https://github.com/scikit-learn/scikit-learn/issues/9354#issuecomment-316074315 ،
أو كتم الخيط
https://github.com/notifications/unsubscribe-auth/AAEz65yy8Aq2FcsDAcWHT8qkkdXF_MfPks5sPLu_gaJpZM4OXbpZ
.
أود العمل على هذا إن أمكن
أذهب خلفها!
لذا أعتقد أن المشكلة تكمن في حقيقة أننا نستخدم sqrt(dot(x, x) - 2 * dot(x, y) + dot(y, y)) لحساب المسافة الإقليدية
لأنني إذا حاولت - (-2 * np.dot(X, Y.T) + (X * X).sum(axis=1) + (Y * Y).sum(axis=1) أحصل على الإجابة 0 لـ np.float32 ، بينما أحصل على الإجابة الصحيحة لـ np.float 64.
jnothman ما رأيك يجب أن أفعل بعد ذلك؟ كما هو مذكور في تعليقي أعلاه ، ربما تكون المشكلة هي حساب المسافة الإقليدية باستخدام sqrt(dot(x, x) - 2 * dot(x, y) + dot(y, y))
إذن أنت تقول أن النقطة تعرض نتيجة أقل دقة من حاصل الضرب ثم المجموع؟
لا ، ما أحاول قوله هو أن النقطة تعرض نتيجة أكثر دقة من حاصل الضرب ثم المجموع
يعطي -2 * np.dot(X, Y.T) + (X * X).sum(axis=1) + (Y * Y).sum(axis=1) الناتج [[0.]]
بينما يعطي np.sqrt(((X-Y) * (X-Y)).sum(axis=1)) الناتج [ 0.02290595]
ليس من الواضح ما تفعله ، ويرجع ذلك جزئيًا إلى أنك لا تنشر مقتطفًا منفردًا بالكامل.
بالنظر بسرعة إلى آخر مشاركة لك ، فإن الشيئين اللذين تحاول مقارنتهما [[0.]] و [0.022...] ليس لهما نفس الأبعاد (ربما مشكلة نسخ ولصق ولكن مرة أخرى يصعب معرفة ذلك لأننا لا الحصول على مقتطف كامل).
طيب آسف بلدي سيئة
import numpy as np
import scipy
from sklearn.metrics.pairwise import check_pairwise_arrays, row_norms
from sklearn.utils.extmath import safe_sparse_dot
# create 64-bit vectors a and b that are very similar to each other
a_64 = np.array([61.221637725830078125, 71.60662841796875, -65.7512664794921875], dtype=np.float64)
b_64 = np.array([61.221637725830078125, 71.60894012451171875, -65.72847747802734375], dtype=np.float64)
# create 32-bit versions of a and b
X = a_64.astype(np.float32)
Y = b_64.astype(np.float32)
X, Y = check_pairwise_arrays(X, Y)
XX = row_norms(X, squared=True)[:, np.newaxis]
YY = row_norms(Y, squared=True)[np.newaxis, :]
#Euclidean distance computed using product-then-sum
distances = safe_sparse_dot(X, Y.T, dense_output=True)
distances *= -2
distances += XX
distances += YY
print(np.sqrt(distances))
#Euclidean distance computed using (X-Y)^2
print(np.sqrt(row_norms(X-Y, squared=True)[:, np.newaxis]))
انتاج
[[ 0.03125]]
[[ 0.02290595136582851409912109375]]
الطريقة الأولى هي كيفية حسابها بواسطة دالة المسافة الإقليدية.
ولتوضيح ما قصدته أعلاه أيضًا ، كان حقيقة أن منتج sum-then-product لديه دقة أقل حتى عندما نستخدم وظائف numpy للقيام بذلك
نعم ، يمكنني تكرار هذا. أرى أن إجراء عملية الطرح في البداية
يسمح بالحفاظ على دقة الاختلاف. عمل النقطة
المنتج ثم الطرح (أو النفي والجمع) ، كما نفعل حاليًا ،
يفقد هذه الدقة نظرًا لأن الأرقام الأكثر أهمية أكبر بكثير من
اوجه الاختلاف.
يعد التنفيذ الحالي أكثر كفاءة في استخدام الذاكرة لعدد كبير من ملفات
المميزات. لكني أفترض أن المسافة الإقليدية أصبحت غير ذات صلة بشكل متزايد
بأبعاد عالية ، لذلك يهيمن على الذاكرة عدد المخرجات
القيم.
لذلك أصوت لاعتماد التطبيق الأكثر استقرارًا عدديًا على
د - تنفيذ فعال بشكل مقارب لدينا حاليًا. رأي،
ogrisel؟ agramfort؟
وهذا بالطبع مصدر قلق أكثر لأننا سمحنا مؤخرًا بـ float32s
أن تكون أكثر شيوعًا عبر المقدرين.
لذلك في هذا المثال ، يعمل product-then-sum بشكل جيد تمامًا مع np.float64 ، لذلك قد يكون الحل المحتمل هو تحويل الإدخال إلى float64 ثم حساب النتيجة وإرجاع النتيجة المحولة مرة أخرى إلى float32. أعتقد أن هذا سيكون أكثر كفاءة ، لكن لست متأكدًا مما إذا كان هذا سيعمل بشكل جيد في بعض الأمثلة الأخرى.
لن يكون التحويل إلى float64 أكثر كفاءة في استخدام الذاكرة من
الطرح.
أوه نعم ، أنت على حق في ذلك ، لكنني أعتقد أن استخدام float64 ثم إجراء منتج ثم مجموع سيكون أكثر كفاءة من الناحية الحسابية إن لم يكن من الحكمة في الذاكرة.
والسبب في استخدام المنتج ثم المجموع هو زيادة الكفاءة الحسابية وليس كفاءة الذاكرة.
بالتأكيد ، لكنني لا أعتقد أن هناك أي سبب لافتراض أنه كذلك في الواقع
أكثر كفاءة من الناحية الحسابية إلا عن طريق عدم الاضطرار إلى إدراك
مجموعة وسيطة. بافتراض أننا نحد من الذاكرة العاملة المطلقة (على سبيل المثال
chunking) ، فلماذا أخذ حاصل الضرب النقطي ومضاعفة القواعد وطرحها
تكون أكثر كفاءة من الطرح والتربيع؟
توفير معايير؟
حسنًا ، لقد أنشأت برنامج نصي بلغة Python لمقارنة الوقت الذي يستغرقه الطرح ثم التربيع والتحويل إلى float64 ثم المنتج ثم الجمع ، واتضح أنه إذا اخترنا X و Y كمتجهات كبيرة جدًا ، فستكون النتائج مختلفة جدًا . jnothman أيضًا ،
هذا هو النص الذي كتبته ، إذا كان هناك أي مشكلة فيرجى إبلاغي بذلك
import numpy as np
import scipy
from sklearn.metrics.pairwise import check_pairwise_arrays, row_norms
from sklearn.utils.extmath import safe_sparse_dot
from timeit import default_timer as timer
for i in range(9):
X = np.random.rand(1,3 * (10**i)).astype(np.float32)
Y = np.random.rand(1,3 * (10**i)).astype(np.float32)
X, Y = check_pairwise_arrays(X, Y)
XX = row_norms(X, squared=True)[:, np.newaxis]
YY = row_norms(Y, squared=True)[np.newaxis, :]
#Euclidean distance computed using product-then-sum
distances = safe_sparse_dot(X, Y.T, dense_output=True)
distances *= -2
distances += XX
distances += YY
ans1 = np.sqrt(distances)
start = timer()
ans2 = np.sqrt(row_norms(X-Y, squared=True)[:, np.newaxis])
end = timer()
if ans1 != ans2:
print(end-start)
start = timer()
X = X.astype(np.float64)
Y = Y.astype(np.float64)
X, Y = check_pairwise_arrays(X, Y)
XX = row_norms(X, squared=True)[:, np.newaxis]
YY = row_norms(Y, squared=True)[np.newaxis, :]
distances = safe_sparse_dot(X, Y.T, dense_output=True)
distances *= -2
distances += XX
distances += YY
distances = np.sqrt(distances)
end = timer()
print(end-start)
print('')
if abs(ans2 - distances) > 1e-3:
# np.set_printoptions(precision=200)
print(ans2)
print(np.sqrt(distances))
print(X, Y)
break
يجدر اختبار كيفية قياسها مع عدد العينات ، وليس فقط
عدد الميزات ... قد يكون لأخذ المعايير فائدة في حساب البعض
مرة واحدة لكل عينة ، وليس مرة واحدة لكل زوج من العينات
في 20 أكتوبر 2017 ، الساعة 2:39 صباحًا ، تلقيت "أسيد رحمن ناصر" [email protected]
كتب:
حسنًا ، لقد أنشأت نصًا بلغة Python لمقارنة الوقت المستغرق
الطرح ثم التربيع والتحويل إلى float64 ثم الضرب ثم الجمع
واتضح أننا إذا اخترنا X و Y كمتجهات كبيرة جدًا ، فإن 2
النتائج مختلفة جدا. أيضا jnothman https://github.com/jnothman
كنت على حق في الطرح ثم التربيع أسرع.
هذا هو النص الذي كتبته ، إذا كان هناك أي مشكلة فيرجى إبلاغي بذلكاستيراد numpy كـ np
استيراد scipy
من sklearn.metrics.pairwise الاستيراد check_pairwise_arrays ، row_norms
من sklearn.utils.extmath استيراد safe_sparse_dot
من وقت استيراد default_timer كمؤقتلأني في النطاق (9):
X = np.random.rand (1،3 * (10 i)). astype (np.float32)Y = np.random.rand (1،3 * (10 i)). astype (np.float32)X ، Y = check_pairwise_arrays (X ، Y)
XX = row_norms (X، تربيع = صحيح) [:، np.newaxis]
YY = row_norms (Y، squared = True) [np.newaxis ،:]# المسافة الإقليدية المحسوبة باستخدام حاصل الضرب ثم المجموع
المسافات = safe_sparse_dot (X ، YT ، dense_output = True)
المسافات * = -2
المسافات + = XX
المسافات + = YYans1 = np.sqrt (مسافات)
بدء = مؤقت ()
ans2 = np.sqrt (row_norms (XY، squared = True) [:، np.newaxis])
النهاية = المؤقت ()
إذا كان ans1! = ans2:
طباعة (بداية نهاية)start = timer() X = X.astype(np.float64) Y = Y.astype(np.float64) X, Y = check_pairwise_arrays(X, Y) XX = row_norms(X, squared=True)[:, np.newaxis] YY = row_norms(Y, squared=True)[np.newaxis, :] distances = safe_sparse_dot(X, Y.T, dense_output=True) distances *= -2 distances += XX distances += YY distances = np.sqrt(distances) end = timer() print(end-start) print('') if abs(ans2 - distances) > 1e-3: # np.set_printoptions(precision=200) print(ans2) print(np.sqrt(distances)) print(X, Y) break-
أنت تتلقى هذا لأنه تم ذكرك.
قم بالرد على هذا البريد الإلكتروني مباشرة ، وقم بعرضه على GitHub
https://github.com/scikit-learn/scikit-learn/issues/9354#issuecomment-337948154 ،
أو كتم الخيط
https://github.com/notifications/unsubscribe-auth/AAEz6z5o2Ao_7V5-Lflb4HosMrHCeOrVks5st209gaJpZM4OXbpZ
.
على أي حال ، هل ترغب في إرسال PR ،ragnerok؟
نعم بالتأكيد ، ماذا تريد مني أن أفعل؟
توفر تطبيقًا أكثر استقرارًا ، وهو أيضًا اختبار قد يفشل في إطار
التنفيذ الحالي ، ومن الناحية المثالية معيارًا يوضح أننا لا نخسر
الكثير من التغيير ، في الحالات المعقولة.
أردت أن أسأل عما إذا كان من الممكن إيجاد مسافة بين كل زوج من الصفوف باستخدام Vectorisation. لا أستطيع التفكير في كيفية القيام بذلك متجهًا.
تقصد الفرق (وليس المسافة) بين أزواج من الصفوف؟ بالتأكيد يمكنك القيام بذلك إذا كنت تعمل مع مصفوفات غير مرتبة. إذا كانت لديك مصفوفات ذات أشكال (n_samples1، n_features) و (n_samples2، n_features) ، فأنت تحتاج فقط إلى إعادة تشكيلها على (n_samples1، 1، n_features) و (1، n_samples2، n_features) وقم بعملية الطرح:
>>> X = np.random.randint(10, size=(10, 5))
>>> Y = np.random.randint(10, size=(11, 5))
X.reshape(-1, 1, X.shape[1]) - Y.reshape(1, -1, X.shape[1])
نعم شكرًا الذي ساعدك حقًا 😄
أردت أيضًا أن أسأل عما إذا كنت أقدم تطبيقًا أكثر استقرارًا لن أستخدم X_norm_squared و Y_norm_squared. فهل أقوم بإزالتها من الحجج أيضًا أم يجب أن أحذر من كونها غير مجدية؟
أعتقد أنه سيتم إهمالهم ، لكن قد نحتاج إلى التأكد أولاً من ذلك
لا توجد حالة يجب أن نحتفظ فيها بهذا الإصدار.
سنكون حذرين للغاية في تغيير هذا. انها تستخدم على نطاق واسع و
تنفيذ طويل الأمد. يجب أن نتأكد من عدم إبطاء أي مهمة
حالات. قد نحتاج إلى إجراء العملية في أجزاء لتجنب الذاكرة العالية
الاستخدام (والذي ربما يكون أكثر تعقيدًا من حقيقة أن هذا يسمى
ضمن الوظائف التي تقسم لتقليل تقاعد ذاكرة الإخراج من
مسافات زوجية).
أود حقًا أن أسمع من المطورين الأساسيين الآخرين الذين يعرفون شيئًا عن الحوسبة
التكاليف والدقة العددية ... ogrisel ، @ lesteve ،rth ...
في 5 تشرين الثاني (نوفمبر) 2017 الساعة 5:27 صباحًا ، "أسيد رحمن ناصر" [email protected]
كتب:
أردت أيضًا أن أسأل عما إذا كنت أقدم تطبيقًا أكثر استقرارًا لن أكون كذلك
باستخدام X_norm_squared و Y_norm_squared. فهل أقوم بإزالتها من ملف
الحجج أيضًا أم يجب أن أحذر من أنها ليست ذات فائدة؟-
أنت تتلقى هذا لأنه تم ذكرك.
قم بالرد على هذا البريد الإلكتروني مباشرة ، وقم بعرضه على GitHub
https://github.com/scikit-learn/scikit-learn/issues/9354#issuecomment-341919282 ،
أو كتم الخيط
https://github.com/notifications/unsubscribe-auth/AAEz63izdpQGDEuW32m8Aob6rrsvV6q-ks5szKyHgaJpZM4OXbpZ
.
ولكن سيكون من الأسهل المناقشة بدقة إذا قمت بفتح العلاقات العامة
حسنًا ، سأفتح العلاقات العامة بعد ذلك ، مع تنفيذ أساسي جدًا لهذه الوظيفة
الأسئلة هي ما الذي يجب عمله حيال هذا الإصدار 0.20. هل يمكن أن يكون هناك بعض التحسينات البسيطة / المؤقتة (حدث على حساب تكلفة استخدام الذاكرة ، على سبيل المثال) التي يمكن أخذها في الاعتبار؟
الحل والتحليل المقترحان في # 11271 قيمتان للغاية بالتأكيد ، ولكن قد يتطلب الأمر مزيدًا من المناقشة للتأكد من أن هذا هو الحل الأمثل. على وجه الخصوص ، أنا قلق بشأن حقيقة أن لدينا الآن بعض المناقشات المعلقة حول الذاكرة العاملة العالمية المثلى في https://github.com/scikit-learn/scikit-learn/issues/11506 اعتمادًا على نوع وحدة المعالجة المركزية وما إلى ذلك أثناء هذا سيضيف مستوى آخر من التقسيم والتعقيد في الكل سيكون الحصول على القليل من التحكم في المنظمة البحرية الدولية. لكن ربما أنا فقط أبحث عن رأي ثان.
ما رأيك ينبغي القيام به حول هذه المشكلة من أجل إطلاق سراحjnothmanamuellerogrisel؟
الاستقرار يتفوق على الكفاءة. يجب إصلاح مشكلات الاستقرار حتى عندما
الكفاءة لا تزال بحاجة إلى تعديلات.
كان تركيز work_memory على صنع أشياء مثل الصور الظلية مع عينة كبيرة
أحجام العمل. لقد حسن أيضًا الكفاءة ، ولكن يمكن تعديل ذلك إلى أسفل
خط.
أعتقد بشدة أننا يجب أن نحاول الحصول على إصلاح للمسافات الإقليدية باستخدام
float32 in. لقد كسرناه في 0.19 بافتراض أننا نستطيع تحقيق ذلك
تعمل euclidean_distances على 32 بت بطريقة ساذجة.
أوافق على أننا بحاجة إلى إصلاح. ما يهمني هنا ليس الكفاءة ولكن التعقيد الإضافي في قاعدة الكود.
بالرجوع إلى الوراء ، يبدو أن تطبيق scipy الإقليدي عبارة عن 10 أسطر من كود C و 32 بت ، قم ببساطة بنقلها إلى 64 بت. أتفهم أنه ليس الأسرع ولكن من السهل من الناحية المفاهيمية المتابعة والفهم. في scikit-Learn ، نستخدم الحيلة لجعل العمليات الحسابية أسرع في BLAS ، ثم هناك تحسينات محتملة بسبب https://github.com/scikit-learn/scikit-learn/pull/10212 والآن الحل المقسم المحتمل للإقليدية المسافة 32 بت.
أنا أبحث فقط عن مدخلات حول ما يجب أن يكون عليه الاتجاه العام حول هذا الموضوع (على سبيل المثال ، حاول توجيه بعض منه إلى scipy ، إلخ).
لا يبدو أن scipy قلقة من نسخ البيانات ...
انتقل إلى 0.21 بعد PR.
إزالة مانع؟
sqrt(dot(x, x) - 2 * dot(x, y) + dot(y, y))
غير مستقر عدديًا ، إذا كانت النقطة (x ، x) والنقطة (y ، y) ذات حجم مماثل للنقطة (x ، y) بسبب ما يعرف بالإلغاء الكارثي .
هذا لا يؤثر فقط على دقة FP32 ، ولكنه بالطبع أكثر وضوحًا ، وسوف يفشل قبل ذلك بكثير.
فيما يلي حالة اختبار بسيطة توضح مدى سوء ذلك حتى مع الدقة المزدوجة:
import numpy
from sklearn.metrics.pairwise import euclidean_distances
a = numpy.array([[100000001, 100000000]])
b = numpy.array([[100000000, 100000001]])
print "skelarn:", euclidean_distances(a, b)[0,0]
print "correct:", numpy.sqrt(numpy.sum((a-b)**2))
a = numpy.array([[10001, 10000]], numpy.float32)
b = numpy.array([[10000, 10001]], numpy.float32)
print "skelarn:", euclidean_distances(a, b)[0,0]
print "correct:", numpy.sqrt(numpy.sum((a-b)**2))
يحسب sklearn مسافة 0 هنا في المرتين ، بدلاً من الجذر التربيعي (2).
يمكن الاطلاع على مناقشة القضايا العددية للتباين والتغاير - وهذا ينتقل بشكل تافه إلى هذا النهج لتسريع المسافة الإقليدية - هنا:
إريك شوبرت ومايكل جيرتز.
الحساب المتوازي المستقر عدديًا للتباين (المشترك).
في: وقائع المؤتمر الدولي الثلاثين لإدارة قواعد البيانات العلمية والإحصائية (SSDBM) ، بولزانو بوزن ، إيطاليا. 2018 ، 10: 1-10: 12
في الواقع ، يمكن إزالة الإحداثي y من حالة الاختبار هذه ، ثم تصبح المسافة الصحيحة 1. لقد قدمت طلب سحب يؤدي إلى هذه المشكلة الرقمية:
XA = np.array([[10000]], np.float32)
XB = np.array([[10001]], np.float32)
assert_equal(euclidean_distances(XA, XB)[0,0], 1)
لا أعتقد أن ورقي المذكور أعلاه يوفر حلاً لهذه المشكلة - فقط احسب المسافة الإقليدية كـ sqrt (sum (power ())) وهي تمريرة واحدة ولها دقة معقولة. الخسارة في استخدام المربعات بالفعل ، أي النقطة (x ، x) نفسها تفقد الدقة بالفعل.
amueller نظرًا لأن المشكلة قد تكون أكثر
شكرا على هذا المثال البسيط جدا.
السبب في تنفيذها بهذه الطريقة هو أنها أسرع بكثير. انظر أدناه:
x = np.random.random_sample((1000, 1000))
%timeit euclidean_distances(x,x)
20.6 ms ± 452 µs per loop (mean ± std. dev. of 7 runs, 10 loops each)
%timeit cdist(x,x)
695 ms ± 4.06 ms per loop (mean ± std. dev. of 7 runs, 1 loop each)
على الرغم من أن عدد العمليات من نفس الترتيب في كلتا الطريقتين (1.5x أكثر في الطريقة الثانية) ، فإن التسريع يأتي من إمكانية استخدام مكتبات BLAS المحسنة جيدًا لمضاعفة مصفوفة المصفوفة.
سيكون هذا تباطؤًا كبيرًا للعديد من المقدرين في scikit-Learn.
نعم، ولكن فقط 3-4 أرقام من الدقة مع FP32، و7-8 الأرقام مع FP64 يفعل سبب عدم الدقة كبيرة، أليس كذلك؟ على وجه الخصوص ، لأن مثل هذه الأخطاء تميل إلى تضخيم ...
حسنًا ، أنا لا أقول أنه جيد الآن. :)
أنا أقول إننا بحاجة إلى إيجاد حل بينهما.
هناك PR (# 11271) الذي يقترح الإلقاء على float64 للقيام بالحسابات. لا يصلح In مشكلة float64 ولكنه يعطي دقة أفضل لـ float32.
هل لديك مثال على أن استخدام المقدّر الذي يستخدم مسافات إقليدية يعطي نتائج خاطئة بسبب فقدان الدقة؟
بالتأكيد ما زلت أعتقد أن هذه مشكلة كبيرة ويجب أن تكون مانعًا لـ 0.21. لقد كانت مشكلة تم تقديمها لـ 32 بت في 0.19 ، وليس من الجيد تركها. أتمنى أن نكون قد حللناها في وقت سابق في 0.20 ، وسأكون بخير ، أو حتى متحمسًا ، لرؤية # 11271 مدمجًا في غضون ذلك. المشكلات الوحيدة في تلك العلاقات العامة التي أعرفها عن تحسين كفاءة الذاكرة المحيطية ، وهي حفرة أرنب عميقة.
لقد كان لدينا هذا الإصدار "السريع" لفترة طويلة ، ولكن دائمًا في float64. أعرف ، @ kno10 ، أن هناك مشكلات تتعلق بالدقة. هل لديك إرشاد جيد وسريع بالنسبة لنا لمعرفة متى قد تكون هذه مشكلة واستخدام حل أبطأ ولكن أكيد؟
نعم ، ولكن فقط 3-4 أرقام من الدقة مع FP32 و7-8 أرقام مع FP64 تسبب عدم دقة كبيرة ، أليس كذلك
شكرا لتوضيح هذه المشكلة بمثال بسيط جدا!
لا أعتقد أن المشكلة منتشرة على نطاق واسع كما تقترح ، ومع ذلك - فهي تؤثر في الغالب على العينات التي تكون المسافة المتبادلة صغيرة فيما يتعلق بمعاييرها.
يوضح الشكل أدناه هذا ، بالنسبة لأزواج العينات العشوائية 2e6 ، حيث تكون كل عينة 1D في الفترة [-100 ، 100]. يتم رسم الخطأ النسبي بين تطبيق scikit-Learn و scipy كدالة للمسافة بين العينات ، والتي تم تطبيعها بواسطة معايير L2 الخاصة بهم ، أي ،
d_norm(A, B) = d(A, B) / sqrt(‖A‖₂*‖B‖₂)
(لست متأكدًا من أنها المعلمات الصحيحة ، ولكن فقط للحصول على نتائج ثابتة إلى حد ما لمقياس البيانات) ،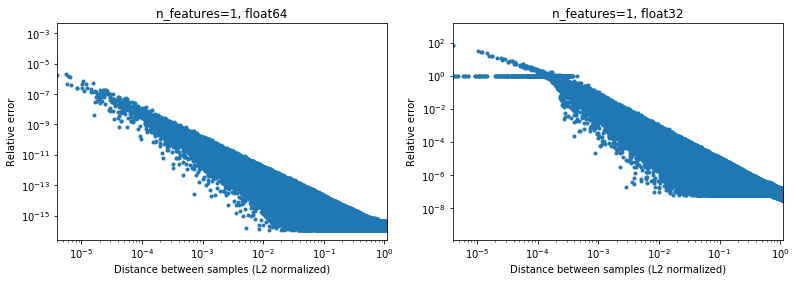
على سبيل المثال،
- إذا أخذ المرء
[10000]و[10001]فإن المسافة المقيسة L2 هي 1e-4 والخطأ النسبي في حساب المسافة سيكون 1e-8 في 64 بت ، و> 1 في 32 بت (أو 1e -8 و> 1 في القيمة المطلقة على التوالي). في 32 بت هذه الحالة رهيبة بالفعل. - من ناحية أخرى ، بالنسبة إلى
[1]و[10001]، سيكون الخطأ النسبي ~ 1e-7 في 32 بت ، أو أقصى دقة ممكنة.
السؤال هو كم مرة ستحدث الحالة 1. في الممارسة العملية في تطبيقات ML.
من المثير للاهتمام ، إذا انتقلنا إلى 2D ، مرة أخرى بتوزيع عشوائي منتظم ، سيكون من الصعب العثور على نقاط قريبة جدًا ،
بالطبع ، في الواقع ، لن يتم أخذ عينات من بياناتنا بشكل موحد ، ولكن بالنسبة لأي توزيع بسبب لعنة الأبعاد ، فإن المسافة بين أي نقطتين سوف تتقارب ببطء مع قيم متشابهة جدًا (تختلف عن 0) مع زيادة البعد. في حين أنها مشكلة عامة في ML ، إلا أنها قد تخفف إلى حد ما مشكلة الدقة هذه ، حتى بالنسبة للأبعاد المنخفضة نسبيًا. أسفل نتائج n_features=5 ،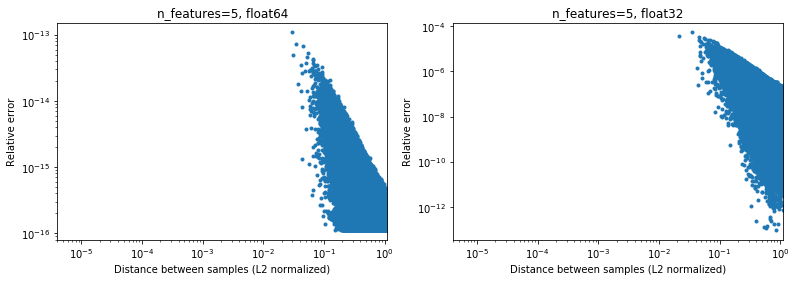 .
.
لذلك بالنسبة للبيانات المركزية ، على الأقل في 64 بت ، قد لا تكون مشكلة كبيرة في الممارسة (بافتراض وجود أكثر من ميزتين). قد يكون التسريع الحسابي بمقدار 50 ضعفًا (كما هو موضح أعلاه) يستحق ذلك (في 64 بت). بالطبع يمكن للمرء دائمًا إضافة 1e6 إلى بعض البيانات التي تم تسويتها في [-1 ، 1] والقول إن النتائج ليست دقيقة ، لكنني سأجادل بأن الأمر نفسه ينطبق على عدد من الخوارزميات الرقمية ، والعمل مع البيانات المعبر عنها في السادس رقم كبير يبحث فقط عن مشكلة.
(يمكن العثور على رمز الأرقام أعلاه هنا ).
أي نهج سريع باستخدام إصدار النقطة (x ، x) + النقطة (y ، y) -2 نقطة (x ، y) من المحتمل أن يكون له نفس المشكلة لكل ما يمكنني قوله ، ولكن من الأفضل أن تسأل بعض الخبراء الحقيقيين في الأرقام لهذا. دقة بيانات
لا يمكنني معرفة مقدار النفقات العامة التي تحصل عليها من تحويل float32 إلى float64 سريعًا لاستخدام هذا الأسلوب. على الأقل بالنسبة لـ float32 ، حسب فهمي ، يجب أن يكون إجراء جميع الحسابات وتخزين المنتجات النقطية على أنها float64 جيدًا.
IMHO ، مكاسب الأداء (التي ليست أسية ، مجرد عامل ثابت) لا تستحق الخسارة في الدقة (والتي يمكن أن تعضك بشكل غير متوقع) والطريقة الصحيحة هي عدم استخدام هذه الحيلة الإشكالية. ومع ذلك ، قد يكون من الممكن زيادة تحسين الكود لإجراء العمليات الحسابية "التقليدية" ، على سبيل المثال لاستخدام AVX. لأن مجموع ((س ص) * 2) صعب التنفيذ في AVX.على الأقل ، أقترح إعادة تسمية الطريقة إلى approximate_euclidean_distances ، بسبب الدقة المنخفضة في بعض الأحيان (التي تزداد سوءًا كلما اقتربت القيمتان ، والتي * قد تكون جيدة في البداية ثم تبدأ في الاهتمام عند التقارب إلى حد ما) ، حتى يكون المستخدمون على دراية بهذه المشكلة.
rth شكرا على الرسوم التوضيحية. ولكن ماذا لو كنت تحاول تحسين ، على سبيل المثال ، x نحو بعض الأمثل. على الأرجح لن يكون المستوى الأمثل عند الصفر (إذا كان دائمًا مركز البيانات الخاص بك ، فستكون الحياة رائعة) ، وفي النهاية قد يكون للدلتا التي تحسبها من أجل التدرجات وما إلى ذلك بعض الاختلافات الصغيرة جدًا.
وبالمثل ، في التجميع ، لن يكون لجميع المجموعات مراكز قريبة من الصفر ، ولكن على وجه الخصوص مع العديد من العناقيد ، يكون مركز x مع بضعة أرقام ممكنًا تمامًا.
بشكل عام ، ومع ذلك ، أوافق على أن هذه المشكلة تحتاج إلى إصلاح. على أي حال ، نحتاج إلى توثيق قضايا الدقة في التنفيذ الحالي في أقرب وقت ممكن.
بشكل عام على الرغم من أنني لا أعتقد أن هذه المناقشة يجب أن تحدث في scikit-Learn. تُستخدم المسافة الإقليدية في مجالات مختلفة من الحوسبة العلمية ، وتعد القائمة البريدية أو القضايا الخاصة بـ IMO مكانًا أفضل لمناقشتها: يتمتع هذا المجتمع أيضًا بخبرة أكبر في مثل هذه القضايا الدقيقة العددية. في الواقع ، ما لدينا هنا هو خوارزمية سريعة ولكنها تقريبية إلى حد ما. قد نضطر إلى تنفيذ بعض الحلول البديلة للإصلاحات على المدى القصير ، ولكن على المدى الطويل سيكون من الجيد معرفة أن هذا سيتم المساهمة فيه هناك.
بالنسبة إلى 32 بت ، قد يكون https://github.com/scikit-learn/scikit-learn/pull/11271 حلاً بالفعل ، فأنا لست حريصًا جدًا على مستويات متعددة من التقسيم عبر المكتبة لأن ذلك يزيد من تعقيد الكود ، وتريد التأكد من عدم وجود طريقة أفضل للتغلب عليها.
شكرا لردكم @ kno10! (تعليقاتي أعلاه لا تأخذها في الاعتبار حتى الآن) سأرد لاحقًا.
نعم ، قد يكون التقارب مع نقطة ما خارج الأصل مشكلة.
IMHO ، مكاسب الأداء (التي ليست أسية ، مجرد عامل ثابت) لا تستحق الخسارة في الدقة (والتي يمكن أن تعضك بشكل غير متوقع) والطريقة الصحيحة هي عدم استخدام هذه الحيلة الإشكالية.
حسنًا ، سيكون للإبطاء> 10x لحساباتهم في 64 بت تأثير حقيقي جدًا على المستخدمين.
ومع ذلك ، قد يكون من الممكن زيادة تحسين الكود لإجراء العمليات الحسابية "التقليدية" ، على سبيل المثال لاستخدام AVX. لأن مجموع ((س ص) ** 2) صعب التنفيذ في AVX.
حاولت تنفيذًا سريعًا ساذجًا باستخدام numba (والذي يجب أن يستخدم SSE) ،
@numba.jit(nopython=True, fastmath=True)
def pairwise_distance_naive(A, B):
n_samples_a, n_features_a = A.shape
n_samples_b, n_features_b = B.shape
assert n_features_a == n_features_b
distance = np.empty((n_samples_a, n_samples_b), dtype=A.dtype)
for i in range(n_samples_a):
for j in range(n_samples_b):
psum = 0.0
for k in range(n_features_a):
psum += (A[i, k] - B[j, k])**2
distance[i, j] = math.sqrt(psum)
return distance
الحصول على سرعة مماثلة لـ scipy cdist حتى الآن (لكنني لست خبيرًا في numba) ، ولست متأكدًا أيضًا من تأثير fastmath .
باستخدام إصدار النقطة (x، x) + dot (y، y) -2 * dot (x، y)
للإشارة إلى المستقبل فقط ، ما نقوم به حاليًا هو ما يلي تقريبًا (نظرًا لوجود بُعد لا يظهر في التدوين أعلاه) ،
def quadratic_pairwise_distance(A, B):
A2 = np.einsum('ij,ij->i', A, A)
B2 = np.einsum('ij,ij->i', B, B)
return np.sqrt(A2[:, None] + B2[None, :] - 2*np.dot(A, B.T))
حيث يستخدم كل من einsum و dot الآن BLAS. أتساءل ، بصرف النظر عن استخدام BLAS ، فهذا أيضًا يؤدي في الواقع نفس عدد العمليات الحسابية مثل الإصدار الأول أعلاه.
أتساءل ، بصرف النظر عن استخدام BLAS ، فهذا أيضًا يؤدي في الواقع نفس عدد العمليات الحسابية مثل الإصدار الأول أعلاه.
لا ، يتم تنفيذ ((x - y) * 2.sum ())* n_samples_x * n_samples_y * n_features * (1 استبدال + 1 إضافة + 1 مضاعفة)
بينما يؤدي xx + yy -2x.y
n_samples_x * n_samples_y * n_features * (إضافة واحدة + مضاعفة واحدة) .
هناك نسبة 2/3 لعدد العمليات بين الإصدارين.
بعد المناقشة أعلاه ،
- صنع علاقات عامة للسماح اختياريًا بحوسبة المسافات الإقليدية بالضبط https://github.com/scikit-learn/scikit-learn/pull/12136
- بعض الأعمال قيد التقدم لمعرفة ما إذا كان بإمكاننا اكتشاف النقاط الإشكالية والتخفيف منها في https://github.com/scikit-learn/scikit-learn/pull/12142
بالنسبة إلى 32 بت ، ما زلنا بحاجة إلى دمج https://github.com/scikit-learn/scikit-learn/pull/11271 في شكل ما على الرغم من IMO ، فإن العلاقات العامة المذكورة أعلاه متعامدة إلى حد ما معها.
لمعلوماتك: عند إصلاح بعض المشكلات في OPTICS ، وتحديث الاختبار لاستخدام النتائج المرجعية من ELKI ، تفشل هذه مع metric="euclidean" ولكنها تنجح مع metric="minkowski" . الاختلافات العددية كبيرة بما يكفي للتسبب في أمر معالجة مختلف (لا يكفي تقليل العتبة فقط).
أنا حقًا لم ألاحظ هذا ، لكنني مندهش من عدم وجود حل حالي. يبدو أن هذا حساب شائع جدًا ويبدو أننا نعيد اختراع العجلة. هل حاول أي شخص الوصول إلى مجتمع الحوسبة العلمية الأوسع؟
ليس بعد ، لكنني أوافق على أنه ينبغي لنا ذلك. الشيء الوحيد الذي وجدته حول هذا في scipy هو https://github.com/scipy/scipy/pull/2815 والمشكلات المرتبطة.
أشعر أن jeremiedbb قد يكون لديه فكرة؟
لسوء الحظ لم تكن مرضية حتى الآن :(
نود الاعتماد على مكتبة مُحسَّنة للغاية لهذا النوع من الحسابات ، كما نفعل مع الجبر الخطي مع مكتبات BLAS مثل OpenBLAS أو MKL. لكن المسافة الإقليدية ليست جزءًا منها. خدعة النقطة هي محاولة للقيام بذلك بالاعتماد على روتين مضاعف مصفوفة مصفوفة المستوى 3 من BLAS. لكن هذا ليس دقيقًا ولا توجد طريقة لجعله أكثر دقة باستخدام نفس الطريقة. علينا خفض توقعاتنا سواء من حيث السرعة أو من حيث الدقة.
أعتقد في بعض الحالات ، الدقة الكاملة ليست إلزامية والحفاظ على الطريقة السريعة أمر جيد. هذا عندما يتم استخدام المسافات لمهام "العثور على الأقرب". تظهر مشكلات الدقة في الطريقة السريعة عندما تكون المسافات بين النقاط صغيرة مقارنة بمعيارها (في النسبة ~ <1e-4 لـ float 32 و ~ <1e-8 لـ float64). أولاً ، لكي يحدث هذا الموقف ، يجب أن تكون مجموعة البيانات كثيفة جدًا. ثم إذا كان لديك خطأ في الطلب ، فأنت بحاجة إلى الحصول على أقرب نقطتين في نفس المسافة تقريبًا. علاوة على ذلك ، في هذه الحالة ، من وجهة نظر ML ، سيؤدي كلاهما إلى نوبات جيدة على قدم المساواة تقريبًا.
في الحالة المذكورة أعلاه ، هناك شيء يمكننا القيام به لتقليل تكرار هذه الطلبات الخاطئة (وصولاً إلى 0؟). في حالة أرجمين المسافة الزوجية. يمكننا نقل الترتيب الخاطئ إلى نقاط ليست الأقرب. استخدام حقيقة أن أحد القواعد ليس ضروريًا للعثور على الأرجمين ، انظر التعليق . لها ميزتان. إنه أكثر قوة (حتى الآن لم أجد طلبًا خاطئًا حتى الآن) وهو أسرع لأنه يتجنب بعض الحسابات.
عيب واحد ، لا يزال في نفس الموقف ، إذا أردنا في النهاية المسافات الفعلية لأقرب النقاط ، فلا يمكن استخدام المسافات المحسوبة بالطريقة المذكورة أعلاه. تم حسابها جزئيًا فقط وليست دقيقة على أي حال. نحتاج إلى إعادة حساب المسافات من كل نقطة إلى أقرب نقطة لها. لكن هذا سريع لأن لكل نقطة مسافة واحدة فقط يجب حسابها.
أتساءل أن ما وصفته أعلاه يغطي جميع حالات استخدام euclidean_distances في sklearn. لكنني أقترح القيام بذلك في أي مكان يمكن تطبيقه فيه. للقيام بذلك ، يمكننا إضافة مُعامل جديد إلى euclidean_distances لحساب الجزء الضروري فقط من أجل ربطه بـ argmin. ثم استخدمه في pairwise_distances_argmin وفي pairwise_distances_argmin_min (إعادة حساب الحد الأدنى للمسافات الفعلية في النهاية في الأخير).
عندما لا نستطيع فعل ذلك ، عد إلى الطريقة البطيئة والدقيقة ، أو أضف مفتاحًا كما في # 12136.
يمكننا محاولة تحسينه قليلاً لتقليل انخفاض الأداء لأنني أوافق على أن هذا لا يبدو مثاليًا. لدي بعض الأفكار لذلك.
هناك إمكانية أخرى للاستمرار في استخدام BLAS وهي الجمع بين axpy nrm2 لكن هذا أبعد ما يكون عن المستوى الأمثل. كلاهما من وظائف BLAS من المستوى 1 ، وهو يتضمن نسخة. سيكون هذا فقط أسرع في البعد> 100.
من الناحية المثالية ، نود تضمين المسافة الإقليدية في BLAS ...
أخيرًا ، هناك حل آخر ، يتمثل في التنبيه. يتم ذلك في # 11271 لـ float32. الميزة هي أن السرعة هي نصف السرعة الحالية ويتم الحفاظ على الدقة. ومع ذلك ، فإنه لا يحل مشكلة float64. ربما يمكننا إيجاد طريقة لعمل شيء مماثل في cython لـ float64. لا أعرف بالضبط كيف ولكن باستخدام رقمين float64 لمحاكاة float128. يمكنني تجربة ذلك لمعرفة ما إذا كان ممكنًا إلى حد ما.
من الناحية المثالية ، نود تضمين المسافة الإقليدية في BLAS ...
هل هذا شيء قد تنظر فيه المكتبات؟ إذا قام OpenBLAS بذلك ، فسنكون في وضع جيد جدًا بالفعل ...
أيضًا ، ما هي الاختلافات الدقيقة بين قيامنا بذلك وبين قيام BLAS بذلك؟ الكشف عن إمكانيات وحدة المعالجة المركزية وتحديد التطبيق الذي يجب استخدامه ، أو شيء من هذا القبيل؟ أو مجرد الحصول على إصدارات مجمعة لمزيد من البنى المتنوعة؟
أو قضاء المزيد من الوقت / الطاقة في كتابة تطبيقات فعالة؟
هذا مثير للاهتمام: تطبيق بديل للطريقة السريعة غير المستقرة ولكن يدعي أنه أسرع بكثير من sklearn:
https://github.com/droyed/eucl_dist
(لا يحل هذه المشكلة على الإطلاق على الرغم من لول)
يبدو أن هذه المناقشة مرتبطة https://github.com/scipy/scipy/issues/5657
إليك ما تفعله جوليا: https://github.com/JuliaStats/Distances.jl/blob/master/README.md#precision-for-euclidean-and-sqeuclidean
يسمح بتعيين حد الدقة لفرض إعادة الحساب.
الإجابة على سؤالي الخاص: يحتوي OpenBLAS على ما يشبه التجميع المكتوب يدويًا لكل معالج (وليس الهندسة المعمارية!) والإرشادات لاختيار نواة لأحجام مشكلة مختلفة. لذلك لا أعتقد أن الأمر يتعلق بإدخاله في openblas بقدر ما يتعلق بالعثور على شخص ما لكتابة / تحسين كل تلك النوى ...
شكرا على الأفكار الإضافية!
في رد جزئي ،
نود الاعتماد على مكتبة مُحسَّنة للغاية لهذا النوع من الحسابات ، كما نفعل مع الجبر الخطي مع مكتبات BLAS مثل OpenBLAS أو MKL.
نعم ، كنت آمل أيضًا أن نتمكن من فعل المزيد من هذا في BLAS. في المرة الأخيرة التي بحثت فيها عن أي شيء في BLAS API القياسي يبدو قريبًا بدرجة كافية (ولكن بعد ذلك لست خبيرًا في ذلك). قد يوفر BLIS مزيدًا من المرونة ولكن نظرًا لأننا لا نستخدمه افتراضيًا ، فهو ذو استخدام محدود نوعًا ما (على الرغم من أن numpy قد يكون يومًا ما https://github.com/numpy/numpy/issues/7372)
إليك ما تفعله جوليا: فهي تسمح بتعيين حد الدقة لفرض إعادة الحساب.
جيد المعرفه!
هل يجب أن نفتح مشكلة منفصلة للحساب التقريبي الأسرع المرتبط أعلاه؟ يبدو مثيرا للإهتمام
قد يكون تسريعهم على وحدة المعالجة المركزية x2-x4 بسبب https://github.com/scikit-learn/scikit-learn/pull/10212 .
أفضل فتح قضية على scipy بمجرد أن ندرس هذا السؤال بما يكفي للتوصل إلى حل معقول هناك (ومن ثم ربما دعمه) لأنني أشعر أن المسافة الإقليدية هي شيء أساسي بما يكفي ويجب أن يكون محل اهتمام العديد من الأشخاص خارج ML (وفي نفس الوقت يكون الحصول على رأي الناس هناك على سبيل المثال حول قضايا الدقة سيكون مفيدًا).
يصل إلى 60x ، أليس كذلك؟
هذا مثير للاهتمام: تطبيق بديل للطريقة السريعة غير المستقرة ولكن يزعم أنه أسرع بكثير من sklearn
همهمة غير متأكد من ذلك. إنهم يقيسون %timeit pairwise_distances(a,b, 'sqeuclidean') ، والذي يستخدم مقياس scipy. يجب أن يفعلوا %timeit pairwise_distances(a,b, 'euclidean', metric_params={'squared': True}) ولن يكون تسريعهم جيدًا :)
كما هو موضح سابقًا في المناقشة ، يمكن أن يكون sklearn أسرع بـ 35 مرة من scipy
نعم ، فهي أفضل بنسبة 30٪ فقط مع metric="euclidean" (بدلاً من squeclidean ) ،
In [1]: from eucl_dist.cpu_dist import dist
... import numpy as np
In [4]: rng = np.random.RandomState(1)
... a = rng.rand(1000, 300)
...b = rng.rand(1000, 300)
In [7]: from sklearn.metrics.pairwise import pairwise_distances
In [8]: %timeit pairwise_distances(a, b, 'sqeuclidean')
214 ms ± 2.06 ms per loop (mean ± std. dev. of 7 runs, 1 loop each)
In [9]: %timeit pairwise_distances(a, b)
27.4 ms ± 2.48 ms per loop (mean ± std. dev. of 7 runs, 10 loops each)
In [10]: from eucl_dist.cpu_dist import dist
In [11]: %timeit dist(a, b, matmul='gemm', method='ext', precision='float32')
20.8 ms ± 330 µs per loop (mean ± std. dev. of 7 runs, 10 loops each)
In [12]: %timeit dist(a, b, matmul='gemm', method='ext', precision='float64')
20.4 ms ± 222 µs per loop (mean ± std. dev. of 7 runs, 10 loops each)
هل هذا شيء قد تنظر فيه المكتبات؟ إذا قام OpenBLAS بذلك ، فسنكون في وضع جيد جدًا بالفعل ...
لا يبدو صريحًا. BLAS عبارة عن مجموعة من المواصفات لإجراءات الجبر الخطي وهناك عدة تطبيقات لها. لا أعرف مدى انفتاحهم على إضافة ميزات جديدة ليست في المواصفات الأصلية. لذلك ربما يكون blis أكثر انفتاحًا ولكن كما قيل من قبل ، فهو ليس الوضع الافتراضي في الوقت الحالي.
تم فتح https://github.com/scikit-learn/scikit-learn/issues/12600 على sqeuclidean مقابل euclidean في pairwise_distances .
أحتاج إلى بعض الوضوح حول ما نريده لهذا الغرض. هل نريد أن يكون pairwise_distances قريبًا - بمعنى all_close - لكلٍّ من "الإقليدية" و "sqeuclidean"؟
إنها صعبة بعض الشيء. لأن x قريبة من y لا يعني ذلك أن x² قريبة من y². تفقد الدقة أثناء التربيع.
حل جوليا المرتبط أعلاه مثير جدًا للاهتمام وهو نوع من السهولة في التنفيذ. ومع ذلك ، أظن أنه لا يعمل كما هو متوقع لـ 'sqeuclidean'. أظن أنه يجب عليك تعيين طريقة العتبة أدناه للحصول على الدقة المطلوبة.
تكمن المشكلة في تحديد عتبة منخفضة جدًا في أنها تؤدي إلى الكثير من عمليات إعادة الحساب وانخفاض كبير في الأداء. ومع ذلك ، يتم تخفيف هذا من خلال أبعاد مجموعة البيانات. ستؤدي نفس العتبة إلى عمليات إعادة حساب أقل في الأبعاد العالية (المسافات أكبر).
ربما يمكن أن يكون لدينا تطبيقان والتبديل حسب أبعاد مجموعة البيانات. النوع البطيء والآمن للأبعاد المنخفضة (لا يوجد فرق كبير بين scipy و sklearn في هذه الحالة على أي حال) و fast + threshold واحد للأبعاد العالية.
سيحتاج هذا إلى بعض المعايير للعثور على وقت التبديل وتعيين العتبة ولكن قد يكون هذا بصيص أمل :)
فيما يلي بعض المعايير لمقارنة السرعة بين scipy و sklearn. تقارن المقاييس بين sklearn.metrics.pairwise.euclidean_distances(X,X) scipy.spatial.distance.cdist(X,X) لـ Xs من جميع الأحجام. ينتقل عدد العينات من 2⁴ (16) إلى 2¹³ (8192) ، ويتراوح عدد الميزات من 2⁰ (1) إلى 2¹³ (8192).
القيمة في كل خلية هي تسريع sklearn مقابل scipy ، أي أن أقل من 1 sklearn أبطأ وأكثر من 1 sklearn أسرع.
المعيار الأول هو استخدام تطبيق MKL لـ BLAS ونواة واحدة.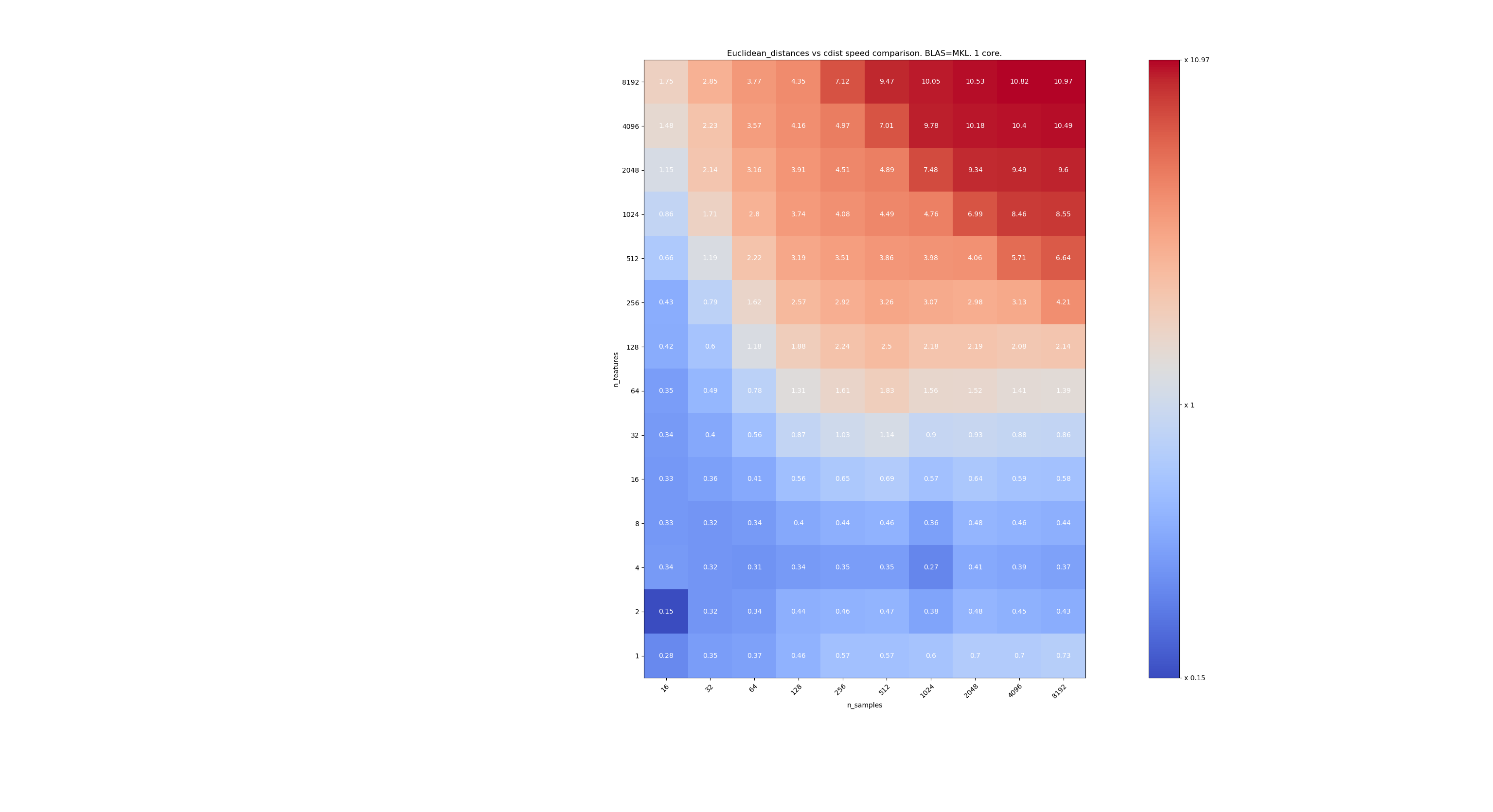
الثاني يستخدم تطبيق OpenBLAS لـ BLAS ونواة واحدة. إنه فقط للتحقق من أن كلا من MKL و OpenBLAS لهما نفس السلوك.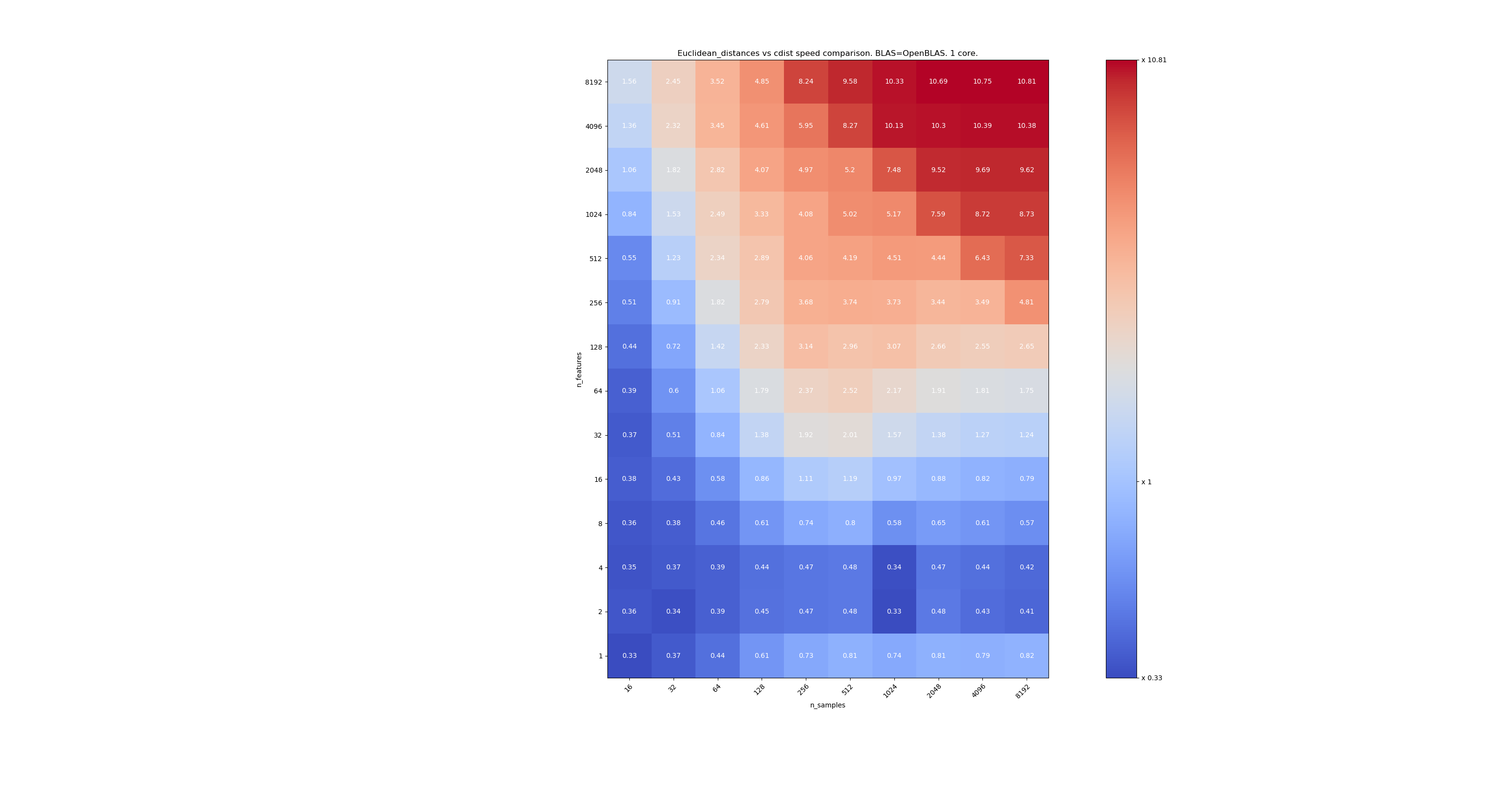
والثالث يستخدم تنفيذ MKL لـ BLAS و 4 مراكز. الشيء هو أن euclidean_distances متوازي من خلال وظيفة BLAS LEVEL 3 ولكن cdist يستخدم فقط وظيفة BLAS LEVEL 1. ومن المثير للاهتمام أنه لا يغير الحدود تقريبًا.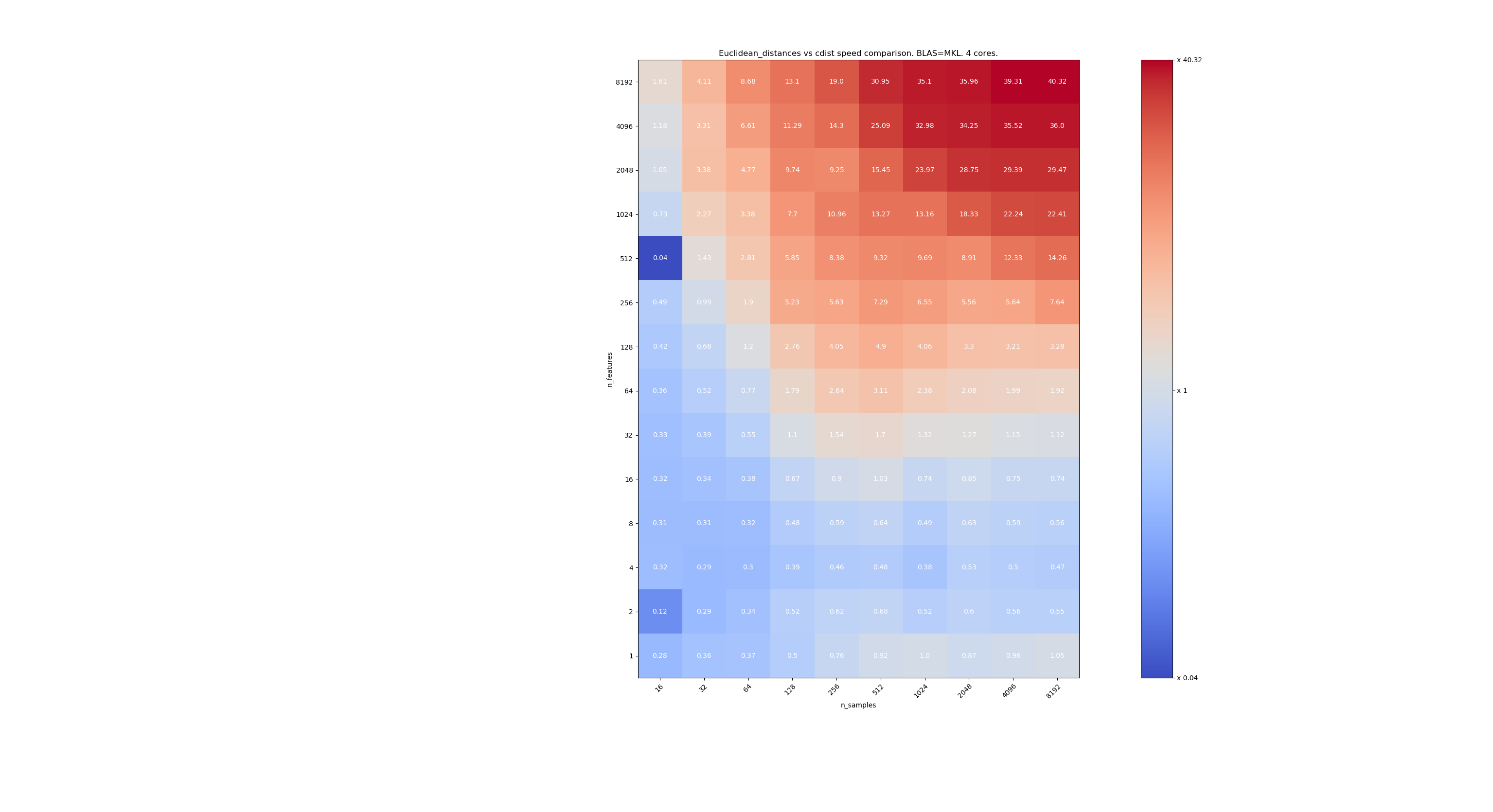
عندما لا تكون n_samples منخفضة جدًا (> 100) ، يبدو أن الحد الأقصى حول 32 ميزة. يمكننا أن نقرر استخدام cdist عندما n_features <32 و euclidean_distances عند n_features> 32. هذا أسرع ولا توجد مشكلة في الدقة. هذا أيضًا له ميزة أنه عندما تكون n_features صغيرة ، فإن عتبة جوليا تؤدي إلى الكثير من عمليات إعادة الحساب. باستخدام cdist يتجنب ذلك.
عندما n_features> 32 ، يمكننا الاحتفاظ بالتنفيذ euclidean_distances ، محدثًا بحد جوليا. لا ينبغي أن تؤدي إضافة العتبة إلى إبطاء euclidean_distances كثيرًا لأن عدد الميزات مرتفع بما يكفي بحيث لا يلزم سوى عدد قليل من عمليات إعادة الحساب.
jeremiedbb رائع ، شكرًا لك على التحليل. الاستنتاج يبدو وكأنه وسيلة رائعة للمضي قدما بالنسبة لي.
أوه ، أفترض أن كل هذا كان لـ float64 ، أليس كذلك؟ ماذا نفعل مع float32؟ صاخب دائما؟ upcast ل> 32 ميزة؟
لم أقرأ التعليقات بعناية (قريباً) ، فقط لمعلوماتك أن float64 له قيود ، انظر # 12128
@ qinhanmin2014 نعم ، دقة float64 لها حدود ، لكنها دقيقة بما يكفي لإنتاج نتائج fp32 موثوقة لكل ما يمكنني قوله. السؤال هو في أي معلمات تكون upcast إلى fp64 أرخص من استخدام cdist من scipy.
كما هو موضح في المعايير أعلاه ، حتى BLAS متعدد النواة ليس أسرع بشكل عام. يبدو أن هذا ينطبق في الغالب على البيانات عالية الأبعاد (أكثر من 64 بعدًا ؛ قبل ذلك ، لا تستحق الفائدة عادةً جهد IMHO) - وبما أن المسافات الإقليدية ليست موثوقة في البيانات عالية الأبعاد الكثيفة ، فإن حالة الاستخدام IMHO ليست ذات أهمية قصوى . سيكون للعديد من المستخدمين أقل من 10 أبعاد. في هذه الحالات ، يبدو أن cdist عادة ما يكون أسرع؟
أوه ، أفترض أن كل هذا كان لـ float64 ، أليس كذلك؟
في الواقع ، إنه لكل من float32 و float64 (أعني متشابهين جدًا). أقترح دائمًا استخدام cdist عند n_features <32.
السؤال هو في أي معلمات تكون upcast إلى fp64 أرخص من استخدام cdist من scipy.
سوف يتباطأ البث المنبثق بمعامل ~ 2 لذا أعتقد أن n_features = 64.
سيكون للعديد من المستخدمين أقل من 10 أبعاد.
ولكن ليس الجميع ، لذلك ما زلنا بحاجة إلى إيجاد حل للبيانات عالية الأبعاد.
تحليل جميل جدا jeremiedbb !
بالنسبة للبيانات ذات الأبعاد المنخفضة ، سيكون من المنطقي بالتأكيد استخدام cdist بعد ذلك.
أيضًا ، لمعلوماتك scipy cdist upcasts float32 إلى float64 https://github.com/scipy/scipy/issues/8771#issuecomment -384015674 ، لست متأكدًا مما إذا كان هذا بسبب مشكلات الدقة أو أي شيء آخر.
بشكل عام ، أعتقد أنه من المنطقي إضافة معلمة "الخوارزمية" إلى euclidean_distance كما هو مقترح في https://github.com/scikit-learn/scikit-learn/pull/12601#pullrequestreview -176076355 ، ربما باستخدام الإعداد الافتراضي على "بلا" بحيث يمكن أيضًا تعيينه عبر خيار عام كما هو الحال في https://github.com/scikit-learn/scikit-learn/pull/12136.
هناك أيضًا نهج مثير للاهتمام في Eigen3 لحساب المعايير الثابتة: https://eigen.tuxfamily.org/dox/StableNorm_8h_source.html (لم أتذمر حقًا بعد)
شرح جيد ، تحسين فهمي
لم نحرز أي تقدم في هذا السباق وربما ينبغي علينا ذلك ... و rth ليس موجودًا اليوم.
يمكنني الانضمام عن بعد إذا حددت موعدًا. ربما في بداية بعد الظهر؟
لتلخيص الموقف ،
لقضايا الدقة في حسابات المسافة الإقليدية ،
- في الحالة ذات الأبعاد المنخفضة ، كما أوضح jeremiedbb أعلاه ، ربما يجب علينا استخدام cdist
- في الحالة عالية الأبعاد و float32 ، يمكننا الاختيار بين ،
- التقسيم وحساب المسافة في 64 بت والتسلسل
- الرجوع إلى cdist في الحالات التي تكون فيها الدقة مشكلة (كيف يكون السؤال مفتوحًا - قد يكون الوصول إلى scipy على سبيل المثال مفيدًا https://github.com/scikit-learn/scikit-learn/issues/9354#issuecomment-438522881 )
ثم هناك كل قضايا التناقضات بين الإقليدية ، الإقليدية ، المينكوفسكي ، إلخ.
فيما يتعلق بالدقة ، أجرينا محادثة سريعة مع jeremiedbb و
لذلك أعتقد أننا سعداء بتعليقrth السابق مع التنبيه إلى upcasting لـ float32. نظرًا لأن cdist تنبثق أيضًا إلى float64 ، فيمكننا إعادة تنفيذ cdist لأخذ float32 (ولكن مع المراكم float64؟) ، أو يمكننا استخدام التقسيم ، إذا كنا نريد نسخًا أقل في float منخفض التعتيم.
هل تريد Celelibi تغيير العلاقات العامة في # 11271 ، أم يجب على شخص آخر (أحدنا؟) تقديم طلب سحب كامل؟
وبمجرد أن يتم إصلاح هذا ، أعتقد أننا يجب أن نجعل sqeuclidean و minkowski (p in {0،1}) يستخدمان تطبيقاتنا. لم نناقش التناقض مع أقرب الجيران. سباق آخر :)
بعد مناقشة سريعة في السباق ، انتهى الأمر بالطريقة التالية:
في حالة ذات أبعاد عالية (> 32 أو> 64 اختر الأفضل): مقلوب من القطع إلى float64 عندما يكون float32 مع الاحتفاظ بالطريقة "السريعة". بالنسبة لهذا النوع من البيانات ، فإن المشكلات العددية ، في float64 ، تكاد لا تذكر (سأقدم معايير لذلك)
في حالة الأبعاد المنخفضة: قم بتنفيذ الحساب الآمن (بدلاً من استخدام scipy cdist بسبب الانكسار) في sklearn.
(من المغري رمي float32 في 0.20.3 أيضًا)
فيما يلي بعض المعايير لمقارنة السرعة بين scipy و sklearn.
[... قص ...]
هذا ممتع جدا. لم أكن أتوقع هذه النتيجة في الواقع. أعدت إجراء المقياس الخاص بك ووجدت نتيجة مشابهة جدًا. إلا أنني أود أن أدافع عن حد أدنى للقرار. يقترح مقياس الأداء الخاص بي 8 ميزات.

تكلفة الخطأ ليست متماثلة. cdist أفضل فقط للحسابات التي تدوم أقل من بضع ثوانٍ ، كما أنه يصبح بطيئًا بسرعة كبيرة عندما يزداد عدد الميزات. لذا ، من الأفضل استخدام تطبيق BLAS عند الشك.
تحرير: هذا المعيار كان لـ float64 ، لكنني أجد أيضًا أن مصفوفات float32 المنبثقة لتطفو 64 تضيف بالكاد نسبة مئوية قليلة إلى إجمالي الوقت ولا تغير الاستنتاج.
لقد لاحظت أن العتبة تعتمد على الجهاز الذي تقوم بتشغيل المعايير عليه. أظن أنه قد يكون له علاقة بتعليمات AVX. أدركت أن المعايير التي نشرتها تم تشغيلها على جهاز لم يكن يحتوي على تعليمات AVX2 ، فقط AVX. وعلى جهاز به AVX2 ، حصلت على نتائج مماثلة لما لديك.
لكن السؤال لا يتعلق بالأداء فحسب ، بل يتعلق أيضًا بالدقة ومن المرجح أن يكون هناك مشكلات تتعلق بالدقة عندما يكون البعد صغيرًا. ربما 16 هو حل وسط جيد. ما رأيك ؟
فيما يتعلق بهذه المناقشة ، أود أن أقول إننا بحاجة إلى قياس الدقة لاتخاذ قرار مستنير.
ومع ذلك ، فيما يتعلق بعلاقاتك العامة ، لا ينبغي أن تكون الدقة مشكلة بعد الآن. ولكن على حساب تكلفة أعلى قليلاً. لذلك من المحتمل أن يتم تحديد الحد الأدنى من خلال قياس العلاقات العامة الخاصة بك.
دقة القياس المعياري ليست بهذه السهولة. لأن الحالات الصعبة لن يتم توزيعها بشكل موحد.
وقد يكون الأمر إشكاليًا إذا حدث دون اكتشافه في قضية ركنية. عادة ، سوف ترغب في الحصول على دقة رقمية مضمونة ضمن حدود وحدة المعالجة المركزية بأسعار معقولة.
ولكن كما هو مذكور في مكان آخر ، يجب أن تكون ميزة واحدة مع 10000000.01 و 10000000.00 كافية لإحداث عدم استقرار رقمي مع fp64 عند استخدام المعادلة ذات المشكلات المعروفة ، 10000 و 10001 مع fp32. مع ميزات 1024 ، جرب
>>> import sklearn.metrics.pairwise as sk, scipy.spatial.distance as sp
>>> X = [[10000.01] * 1024, [10000.00] * 1024]
>>> print(sk.euclidean_distances(X,X), "\n", sp.cdist(X,X))
[[ 0. 0.31895195]
[ 0.31895195 0. ]]
[[ 0. 0.32]
[ 0.32 0. ]]
(تم استخدام 0.19.1) المسافة الصحيحة هي 0.32.
كما ترى ، يميل عدم الاستقرار الرقمي إلى التفاقم مع عدد الميزات (ما لم تكن بياناتك متفرقة). هنا ، تكون النتيجة أقل من رقمين من الدقة مع FP64.
13410 لا يصلح هذه الحالة بالتحديد. أي float64 + بعد عالي.
ومع ذلك ، يتم إصلاحه لـ float32.
لكننا قررنا أنه بالنسبة لـ float64 + عالي التعتيم ، فإننا نحافظ عليه كما كان ، لأن مشكلات الدقة من غير المرجح أن تحدث ولا تنطبق حقًا على حالات استخدام التعلم الآلي.
في المثال الخاص بك ، X [0] و X [1] لها معايير تساوي 320000.32 و 320000 ومسافتهما 0.32 ، أي 1e-6 أضعاف القاعدة. في التعلم الآلي ، لا تكون جميع الأرقام الستة عشر المهمة (في float64) ذات صلة.
لكننا قررنا أنه بالنسبة لـ float64 + عالي التعتيم ، فإننا نحافظ عليه كما كان ، لأن مشكلات الدقة من غير المرجح أن تحدث ولا تنطبق حقًا على حالات استخدام التعلم الآلي.
سأكون أكثر اعتدالًا في هذا. يعد تقليل الأبعاد خطوة أولى معتادة في ML. يمكن استخدام MDS لذلك ، وهو يستخدم بشكل مكثف مصفوفة المسافة الإقليدية.
إذا أراد شخص ما إلقاء نظرة على تحسين دقة حالة float64 ، فهناك طريقة لاستخدام عوامين لتمثيل النتائج الوسيطة. على الرغم من أنني أعتقد أنه بدأ في الخروج عن نطاق تعلم scikit.
ftp://ftp.math.ethz.ch/users/wpp/CELL/qd.pdf
لم أكن واضحا. أنا لا أقول أن البيانات عالية الأبعاد لا تنطبق على التعلم الآلي. أنا أقول إن هذا النوع من مشكلات الدقة التي تحدث في float64 يتضمن نقاطًا تكون المسافة فيها 6 أوامر من حيث الحجم أصغر من معاييرها. إن وجود مثل هذه الدقة ليس له معنى في نموذج التعلم الآلي الواقعي
في التعلم الآلي ، لا تكون جميع الأرقام الستة عشر المهمة (في float64) ذات صلة.
لست مقتنعًا على الإطلاق بأن هذا صحيح بشكل عام.
في هذا المثال ، فقدنا 15 من 16 رقمًا في الدقة. أوافق على ما إذا كنا سنستخدم نصف الدقة ، لكن ليس لدينا مثل هذه العلاقة. غالبًا ما يكون الخسارة الناتجة عن خفض FP64 إلى FP32 مقبولة بسبب دقة القياس. وتكون وحدات معالجة الرسومات على مستوى المستهلك أسرع بكثير مع FP32 مقارنة مع FP64 ، على سبيل المثال (في بعض الحالات ، تسمح ببيانات FP32 ومراكمات FP64 الآن ، على الرغم من ذلك) ، وبالنسبة لاستدلال الشبكات العصبية ، قد ترى int8 الآن. لكن هذا لا ينطبق في كل مكان.
على سبيل المثال في وسائل ك المجموعات، هناك افتراض أن مجموعات تختلف إلى حد كبير في وسائلها (وأننا لا نعرف الوسائل مسبقا)، وبالتالي لدينا خسارة في الدقة هنا. إذا كان لديك العديد من المجموعات ، يمكن أن تكون بعض معاييرها كبيرة مقارنة بفصلها.
علاوة على ذلك ، بعد التكرارات الأولية الأولى ، غالبًا ما تؤدي الاختلافات الصغيرة في المسافة إلى تحويل نقطة واحدة إلى مجموعة أخرى. يمكن أن يؤثر فقدان الدقة هنا على النتائج وقد يتسبب في عدم الاستقرار.
فكر الآن في الوسائل k على أجزاء السلاسل الزمنية مع العديد من المتغيرات.
مع زيادة أحجام البيانات ، يجب أن نفترض أن المسافات إلى أقرب الجار تصبح أصغر ، وما لم تكن معاييرك 0 ، فستكون في النهاية أصغر من معايير المتجه وتسبب مشاكل. لذلك من المحتمل أن يصبح هذا أكثر خطورة مع زيادة أحجام مجموعات البيانات. تقول لعنة الأبعاد أن المسافات الأكبر والأصغر تتشابه أكثر فأكثر ؛ لذلك من أجل حساب الترتيب الصحيح لأقرب الجيران ، قد نحتاج إلى دقة جيدة في البيانات عالية الأبعاد. في مجموعة بيانات 20news ، تكون أصغر مسافة غير صفرية حوالي 0.02 (المعايير كلها 1). ولكن هذا مجرد 10 آلاف حالة ومحتويات متنوعة إلى حد ما. افترض الآن أن مجموعة البيانات كانت تدور حول الاكتشاف شبه المكرر بدلاً من ذلك ...
لن أكون متأكدًا من حدوث هذا "غير المحتمل" في ML ... بالطبع لن يؤثر على الجميع.
عندما أقول "في التعلم الآلي ، فإن 16 رقمًا مهمًا (في float64) ليست كلها ذات صلة." ، لا أتحدث عن المسافة المحسوبة ، فأنا أتحدث عن البيانات X.
في التعلم الآلي ، تأتي بياناتك من مقياس ، ولا يوجد مقياس دقيق للرقم التاسع (باستثناء القليل جدًا في فيزياء الجسيمات).
إذن ، في مثالك 10000000.01 و 10000000.00 ، كيف يمكنك إعطاء بعض الأهمية لمسافة 0.01 عندما يكون عدم اليقين بشأن قيم X أكبر بكثير؟
بالنسبة لـ KMeans ، أولاً هناك طريقة للتغلب على جزء كبير من فقدان الدقة. عندما تبحث عن أقرب مركز للمراقبة x ، لا تحتاج إلى إضافة معيار x إلى حساب المسافة الذي يتجنب الإلغاء الكارثي في معظم الحالات.
ثم ، مجموعات kmeans على أساس المسافات الإقليدية. لكنك لا تعرف ما إذا كانت هذه هي الطريقة الدقيقة لجمع بياناتك. في الواقع ، هناك احتمال صفر أن تكون بياناتك مجمعة بهذه الطريقة. يقدم برنامج Kmeans تقديراً لكيفية تجميع بياناتك ، ولا يمكن بالتأكيد اعتبار النقاط التي تقع في حدود مجموعتين من المجموعات التي تنتمي على وجه اليقين إلى واحدة أو أخرى. ما هو تفسيرك لنقطة على نفس المسافة بين مجموعتين؟ المنجم هو إما أن تكون المجموعتان مجموعة واحدة فقط أو أن KMeans ليس أفضل مجموعة لتجميع بياناتي (أو حتى كمينز تعطيني فكرة جيدة إلى حد ما عن كيفية تجميع بياناتي ولكني أعلم أن حدود المجموعات ليست ذات صلة).
لا يؤدي استخدام "| b | ^ 2-2ab" فقط إلى إلغاء كارثي - ولكن الخسارة نفسها في الدقة في الأرقام هي التي تحدث فرقًا. النتائج هي نفسها كما لو أضفت معيار a لكل مسافة بعد ذلك ؛ إذا كانت المسافات أصغر بكثير من المعيار a ، فستحصل على خسارة في الدقة يمكن تجنبها عن طريق إجراء الحسابات بالطريقة التقليدية دون اختراق BLAS.
لذلك في الواقع لا يمكنك التغلب على المشكلة العددية بهذه الطريقة!
K- يعني مشكلة التحسين. لذلك قد تعني هذه الاختراقات أن sklearn لا تجد سوى حلول أسوأ من الأدوات الأخرى. وكما أشرنا من قبل ، يمكن أن يتسبب هذا أيضًا في عدم الاستقرار. في أسوأ الحالات ، يمكن أن يتسبب ذلك في أن تتكرر كيلومترات sklearn خلال نفس الحالات حتى max_iter بدون أي تحسن (بافتراض أن tol = 0 ، إذا كنت تريد العثور على أفضل محلي) ، وهو ما قد تقوله النظرية أنه مستحيل.
حتى تتقارب الوسائل k ، لا يمكنك قول الكثير عن النقاط التي لها نفس المسافة إلى مجموعتين. في التكرار التالي ، ربما تحركت الوسائل ويمكن أن يصبح الفرق أكبر بكثير ويصبح مهمًا!
أنا لست من أشد المعجبين بوسائل k لأنها لا تعمل بشكل جيد على البيانات الصاخبة. ولكن هناك اختلافات تتعامل مع مثل هذه الحالات بشكل أفضل. ولكن مع ذلك ، إذا كنت تستخدمها ، فمن المحتمل أن تحاول الحصول على الجودة الكاملة (وهذا هو السبب في أنني أستخدم دائمًا tol=0 ) ولا أجعل الأمر أسوأ من اللازم. إنها رخيصة بما يكفي لإجراء العمليات الحسابية الصحيحة (وكما ذكرنا ، تزداد المشكلات سوءًا مع حجم البيانات - لذلك بالنسبة للبيانات الصغيرة ، لا يهم وقت التشغيل الأبطأ عادةً ، بالنسبة لمجموعات البيانات الأكبر ، تصبح الدقة أكثر أهمية على الأرجح).
اعتمادا على التطبيق، والفرق بين 10000000.01 و 10000000.00 يمكن أن يهم. وكما أوضحت من قبل ، إذا كنت تستخدم ميزات متعددة ، فستظهر المشاكل في وقت سابق. مع fp32 بحجم صغير يصل إلى 10000 و 10001 بميزة واحدة و 100 مقابل 101 مع 100 ميزة أعتقد:
كما ذكرنا ، قد يكون للمتوسط معنى مادي لا تريد أن تفقده. إذا كانت لديك بيانات بدرجات حرارة بمقياس كلفن ، فأنت لا تريد قياسها بنسبة 0: 1 أو توسيطها ؛ من شأنه أن يفسد مقياس النسبة الخاص بك. الآن إذا كنت تريد مقارنة ، على سبيل المثال ، السلاسل الزمنية لدرجة حرارة بعض منتجات الصلب أثناء تبريدها ، ومعرفة ما إذا كانت عملية التبريد تؤثر على موثوقية منتج الصلب الخاص بك. قد يكون لديك درجات حرارة تزيد عن 700 كلفن ، وقد تحتوي السلسلة الزمنية على مئات من نقاط البيانات إذا كنت تريد تحليل عملية التهدئة. حتى مع وجود 5 أرقام فقط من دقة الإدخال (0.01 ك) مع طول السلسلة الزمنية ، يمكن أن تحدث مشكلة رقمية. قد ينتهي بك الأمر مرة أخرى مع رقم واحد إلى رقمين فقط في النتيجة. لا أعتقد أنه يمكنك فقط استبعاد أن الدقة مهمة في ML إذا كان لديك هذا النوع الكارثي من التأثير. إنه أمر مختلف إذا كان بإمكانك ضمان الحصول دائمًا على 10 من 16 رقمًا بدقة. هنا لا يمكنك فعل ذلك ، قد يكون لديك 0 أرقام في أسوأ الحالات (وهذا هو السبب في أنها كارثية).
في التعلم الآلي ، تأتي بياناتك من مقياس ، ولا يوجد مقياس دقيق للرقم التاسع (باستثناء القليل جدًا في فيزياء الجسيمات).
نادراً ما تتمتع القيم الأولية من العالم الحقيقي بهذا النوع من الدقة ، هذا صحيح. لكن ML لا يقتصر على هذا النوع من المدخلات. قد يرغب المرء في تطبيق ML على المشاكل الرياضية ، مثل تطبيق MDS على الرسم البياني لألغاز تشبه مكعب روبيك أو تجميع الاستراتيجيات الناجحة التي وجدها سرب وكلاء RL الذين يلعبون pacman.
حتى إذا كان المصدر الأولي للمعلومات هو العالم الحقيقي ، فقد يكون هناك بعض المعالجة المتوسطة التي تجعل معظم الأرقام ذات صلة بخوارزمية التجميع. مثل نتيجة نزول متدرج على دالة يتم أخذ عينات معلماتها إحصائيًا في العالم الحقيقي.
أنا في الواقع أتساءل لماذا ما زلنا نناقش هذا. أعتقد أننا نتفق جميعًا على أن scikit-Learn يجب أن يبذل قصارى جهده في دقة المقايضة مقابل وقت الحساب. ومن غير راضٍ عن الوضع الحالي ، يجب عليه تقديم طلب سحب.
لا يؤدي استخدام "| b | ^ 2-2ab" فقط إلى إلغاء كارثي - ولكن الخسارة نفسها في الدقة في الأرقام هي التي تحدث فرقًا. النتائج هي نفسها كما لو أضفت معيار a لكل مسافة بعد ذلك ؛ إذا كانت المسافات أصغر بكثير من المعيار a ، فستحصل على خسارة في الدقة يمكن تجنبها عن طريق إجراء الحسابات بالطريقة التقليدية دون اختراق BLAS.
لذلك في الواقع لا يمكنك التغلب على المشكلة العددية بهذه الطريقة!
هناك فقدان في الدقة ، لكن لا يمكن أن يتسبب ذلك في إلغاء كارثي (على الأقل عندما يكون a و b قريبين) ، ويمكنك إظهار أن الخطأ النسبي على المسافة (وهو ليس مسافة) يبقى صغيرًا.
في حالة KMeans حيث تكون مهتمًا فقط بالعثور على أقرب مركز ، لديك دقة كافية للحفاظ على الترتيب الصحيح. إذا كنت تريد القصور الذاتي في النهاية ، فيمكنك فقط حساب مسافات كل نقطة إلى مركز الكتلة الخاص بها باستخدام الصيغة الدقيقة.
بالإضافة إلى ذلك ، لا تعد KMeans مشكلة تحسين محدبة ، لذلك حتى إذا سمحت لها بالعمل مع tol = 0 حتى التقارب ، ينتهي بك الأمر في حد أدنى محلي يمكن أن يكون بعيدًا عن الحدود الدنيا العالمية (حتى مع تهيئة kmeans ++). لذلك أفضل تشغيل kmeans عدة مرات باستخدام init مختلفة وعدد صغير من التكرارات. سيكون لديك فرصة أفضل لتنتهي في منطقة دنيا محلية أفضل. ثم يمكنك إعادة تشغيل أفضل واحد حتى التقارب.
يمكن أن يكون الخطأ النسبي مقارنة بالمسافة الحقيقية كبيرًا بشكل تعسفي ، وبالتالي يتسبب في أقرب جيران خاطئين. ضع في اعتبارك الحالة حيث | a | ² = | b | ² = 1 ، على سبيل المثال في tf-idf. افترض أن المتجهات قريبة جدًا. ثم ab قريب أيضًا من 1 ، وفي هذه المرحلة فقدت بالفعل الكثير من دقتك.
كما كتبت أعلاه ، الخطأ موجود ، حتى لو لم يكن لديك إلغاء كارثي. ضع في اعتبارك 8 أرقام من الدقة. قد تكون المسافة الحقيقية 0.000012345678 ويمكن حسابها بثمانية أرقام باستخدام FP32 والمسافة الإقليدية العادية. ولكن باستخدام هذه المعادلة ، يمكنك حساب القيمة ab = 0.99998765432 بدلاً من ذلك ، والتي سيتم اقتطاعها باستخدام FP32 إلى ما يقرب من 0.99998765 في أحسن الأحوال ، لذا فقد فقدت ثلاثة أرقام من الدقة دون داعٍ في هذا المثال. الخسارة كبيرة كما في الحالة الكارثية. إذا كانت المسافات أصغر بكثير من المعايير ، يمكن أن تصبح الدقة سيئة بشكل تعسفي مع هذا النهج.
نعم ، كمينز ليست محدبة. ولكن بعد ذلك سترغب على الأقل في العثور على المستوى الأمثل فرصة للعثور على الحالة العالمية في حالات حسنة التصرف ومع محاولات متعددة.
أقدر هذه المناقشة ، لكن ما نحتاجه حقًا هو حل ليس أسوأ مما كنا نفعله قبل أن نتوقف عن تنبيه الأشياء إلى float64. في هذا المعنى، وكان الحلCelelibi الصورة upcasting كافية. يعد استخدام الحل الدقيق بأبعاد منخفضة تحسينًا إضافيًا لما اعتدنا القيام به.
فيما يتعلق بالإصدار المستقبلي ، هل تشعر بمزيد من الثقة للكشف بكفاءة عن الوقت الذي يمكننا فيه التفكير في الحساب الدقيق بأبعاد عالية؟
لقد أجريت معيارًا لتقييم متوسط دقة حالة float64 بأرقام عشوائية. أقارن ثلاث خوارزميات: neumaier_sum((x-y)**2) ، numpy.sum((x-y)**2) و X2 - 2*X.dot(Y.T) + Y2.T . تم الحصول على النتيجة الدقيقة للمقارنة باستخدام mpmath بدقة 256 بت.
X و Y على 100 عينة وعدد متغير من الميزات ويتم ملؤها بأرقام عشوائية بين -2 و 2.
في الصورة المتحركة التالية ، توجد صورة واحدة لكل عدد من المعالم (بين 1 و 200). في كل صورة ، تمثل كل نقطة الخطأ النسبي للمسافة الإقليدية التربيعية بين زوج من المتجهات 10000 X و Y . يتم ضرب الخطأ النسبي في 2 ^ 53 لسهولة القراءة ، وهو ما يتوافق تقريبًا مع وحدة ULP.
المنحنيات أعلاه هي التوزيع التقريبي (باستخدام تقدير كثافة النواة).

لاحظ أنه تم قطع الرسوم البيانية عند 6 ULP لسهولة القراءة. إنه يظهر الحالة المتوسطة ، وليس الحالة الأسوأ. يمكن أن ينمو خطأ الصيغة الموسعة بشكل كبير.
تحليلي لهذه النتيجة هو أنه في المتوسط ، يمكن أن يكون الخطأ النسبي للصيغة الموسعة كبيرًا جدًا مع عدد قليل من الميزات ، ولكنه سرعان ما يصبح مشابهًا لذلك الخاص بالفرق والمبلغ الإجمالي. الحد الأدنى بين 5 و 10 ميزات.
أحاول حاليًا أيضًا العثور على حد أعلى لخطأ الصيغة الموسعة بالإضافة إلى الأمثلة المرضية.
أعتقد أن اهتمام @ kno10 هو أننا غالبًا ما نهتم بالحالات التي يكون فيها
لا يتم توزيع النقاط بشكل عشوائي ، ولكنها قريبة من بعضها البعض أو لها وحدة
معيار.
في الواقع ، لكنني كنت بحاجة إلى أن أكون مقتنعًا أنه من الناحية العملية ، ليس هذا هو درجة البكالوريوس الكاملة. ^ ^
لإكمال التعليق أعلاه: يبدو أن الخطأ النسبي للصيغة x²+y²-2ab غير محدود. ما لم يكن تحليلي خاطئًا ، عندما يقترب x و y من بعضهما البعض ، يمكن أن يصل الخطأ النسبي إلى 2^(52*2) . على الأقل من الناحية النظرية. من الناحية العملية ، فإن أسوأ حالة وجدتها هي خطأ نسبي قدره 2^52+1 .
>>> a, b = (0xfffffec4d6282+1) * 2.0**(511-52), 0xfffffec4d6282 * 2.0**(511-52)
>>> a, b
(6.703903473040778e+153, 6.7039034730407766e+153)
>>> exactdiff = (a-b)**2
>>> exactdiff
2.2158278651204453e+276
>>> computeddiff = a**2 + b**2 - 2*a*b
>>> computeddiff
-9.9792015476736e+291
>>> abs((computeddiff - exactdiff) / exactdiff)
4503599627370497.0
>>> bin(int(abs((computeddiff - exactdiff) / exactdiff)))
'0b10000000000000000000000000000000000000000000000000001'
إن قلب علامة النتيجة سيجعلها في الواقع أقرب إلى الحقيقة. هذا هو المثال الأكثر دراماتيكية الذي يمكن أن أجده ، لكن تغيير الأس في قيم a و b لا يغير الخطأ النسبي.
>>> a, b = (0xfffffec4d6282+1) * 2.0**(-52), 0xfffffec4d6282 * 2.0**(-52)
>>> a, b
(0.9999999266202912, 0.999999926620291)
>>> exactdiff = (a-b)**2
>>> computeddiff = a**2 + b**2 - 2*a*b
>>> abs((computeddiff - exactdiff) / exactdiff)
4503599627370497.0
أعتقد أن رسم الرسم البياني في ULPs سيكون أكثر منطقية من الرسوم المتحركة أعلاه مع توزيع الخطأ داخل ULP. لذا فإن خطأ 0 ULP و 1 خطأ ULP "جيد كما يحصل". 2 من المحتمل أن تكون ULP أمرًا لا مفر منه بسبب الجذر التربيعي. أفترض أن أي أخطاء أكبر تستحق التحقيق.
يعد استخدام (computed - exact) / exact أمرًا معقولاً طالما أن القيمة الدقيقة كبيرة. ولكن بمجرد أن نواجه تحديات عددية للقيمة الدقيقة ، يصبح هذا غير مستقر تمامًا. في مثل هذه الحالات ، قد يكون من المفيد استخدام (computed-exact)/norm بدلاً من ذلك ، أي النظر إلى دقة حسابات المسافة لدينا مقارنةً ببيانات الإدخال ، وليس مقارنة بالمسافات المشتقة.
إذا كان لدينا قيمتان أحاديتا البعد لا تختلفان إلا بمقدار 1 ULP ، وقد يبدو خطأ 2 ULP ضخمًا ؛ لكننا في حل بيانات الإدخال بالفعل ، وبالتالي فإن النتائج غير مستقرة تمامًا.
لاحظ أنه مع أبعاد متعددة ، قد نحصل على دقة أعلى في بيانات الإدخال.
ضع في اعتبارك بيانات الإدخال من النوع (1, 1e-16) مقابل (1, 2e-16) . على سبيل المثال ، إذا كانت لدينا سمة ثابتة في بيانات الإدخال ، على سبيل المثال ، بكسل أبيض في MNIST.
مع المعادلة المبنية على الفروق ، سيكون هذا جيدًا ، لكن النسخة النقطية تواجه مشكلة ، أليس كذلك؟ هذا هو السبب في أن التجارب أحادية البعد قد لا تكون كافية لدراسة هذا.
أعتقد أن رسم الرسم البياني في ULPs سيكون أكثر منطقية من الرسوم المتحركة أعلاه مع توزيع الخطأ داخل ULP.
لست متأكدًا من أنني أرى كيف كنت ستمثلها. سيكون هناك مدرج تكراري واحد لكل عدد من الميزات ولكل خوارزمية. لا يمكنني فعل الكثير بجانب الرسم ثلاثي الأبعاد أو الرسوم المتحركة.
يعد استخدام
(computed - exact) / exactأمرًا معقولاً طالما أن القيمة الدقيقة كبيرة. ولكن بمجرد أن نواجه تحديات عددية للقيمة الدقيقة ، يصبح هذا غير مستقر تمامًا.
لست متأكدًا مما تقصده بعدم الاستقرار في هذا السياق. يجب حساب القيمة الدقيقة بكل ما يلزم لجعلها دقيقة.
(بالحديث عن ذلك ، كان يجب أن أحسب الخطأ النسبي بدقة تعسفية أيضًا في مؤامرة بياني ، بدلاً من المقارنة بالنتيجة المقربة بالضبط. لقد قمت بتحديث مؤامرة ، واختفت الموجات الغريبة.)
في مثل هذه الحالات ، قد يكون من المفيد استخدام
(computed-exact)/normبدلاً من ذلك ، أي النظر إلى دقة حسابات المسافة لدينا مقارنةً ببيانات الإدخال ، وليس مقارنة بالمسافات المشتقة.
إذا فهمت فكرتك بشكل صحيح ، تفضل مقارنة الخطأ المطلق بحجم بيانات الإدخال. استخدام معايير المتجه كمقياس مجمع لحجم المدخلات. في حين أن الخطأ النسبي القياسي يقارنه بحجم النتيجة الدقيقة.
أعتقد أنه باستخدام هذا المقياس ، ستحاول معرفة مقدار الخلل في الخوارزمية. لكنها في الواقع لا تبدو مفيدة بشكل خاص لعدة أسباب.
- لا يشير حقًا إلى عدد أرقام النتيجة غير الدقيقة.
- في الواقع ، سيكون لمعظم الخوارزميات درجة أقل من 1e-15. حتى الصيغة الموسعة (الخوارزمية القائمة على النقاط) سيكون لها درجة يحدها شيء مثل 5 ULP (مدخلات) (تقدير تقريبي ، لم أكتب الدليل الكامل).
- ونظرًا لأن كلا المقياسين ما هي إلا نسخة معدلة من الخطأ المطلق
computed - exact، فسيقومان بترتيب الخوارزميات بالترتيب نفسه عند تقييمها على نفس المدخلات.
لذلك فهو نفس الخطأ النسبي المعتاد ، فقط مع تفسير القيمة الأقل فائدة (IMO).
ضع في اعتبارك بيانات الإدخال من النوع
(1, 1e-16)مقابل(1, 2e-16). على سبيل المثال ، إذا كانت لدينا سمة ثابتة في بيانات الإدخال ، على سبيل المثال ، بكسل أبيض في MNIST.
مع المعادلة المبنية على الفروق ، سيكون هذا جيدًا ، لكن النسخة النقطية تواجه مشكلة ، أليس كذلك؟ هذا هو السبب في أن التجارب أحادية البعد قد لا تكون كافية لدراسة هذا.
سيكون للخوارزمية القائمة على النقاط خطأ نسبي 1 ، مما يعني أن الخطأ كبير مثل النتيجة الدقيقة ، وبالتالي ، لا يوجد رقم صحيح في النتيجة. وسيكون للمقياس قيمة 1e-16 مما يعني أنه بالنسبة لمقياس معيار المتجه ، فإن الرقم 16 فقط هو خارج.
لست متأكدًا مما تحاول إظهاره بهذا المثال.
إذا كنا لا نزال نشعر بالقلق إزاء دقة euclidean_distances مع float64 ، فمن الأفضل على الأرجح تلخيص هذه المناقشة في عدد جديد حيث يوجد 100 تعليق هنا.
التعليق الأكثر فائدة
نادراً ما تتمتع القيم الأولية من العالم الحقيقي بهذا النوع من الدقة ، هذا صحيح. لكن ML لا يقتصر على هذا النوع من المدخلات. قد يرغب المرء في تطبيق ML على المشاكل الرياضية ، مثل تطبيق MDS على الرسم البياني لألغاز تشبه مكعب روبيك أو تجميع الاستراتيجيات الناجحة التي وجدها سرب وكلاء RL الذين يلعبون pacman.
حتى إذا كان المصدر الأولي للمعلومات هو العالم الحقيقي ، فقد يكون هناك بعض المعالجة المتوسطة التي تجعل معظم الأرقام ذات صلة بخوارزمية التجميع. مثل نتيجة نزول متدرج على دالة يتم أخذ عينات معلماتها إحصائيًا في العالم الحقيقي.
أنا في الواقع أتساءل لماذا ما زلنا نناقش هذا. أعتقد أننا نتفق جميعًا على أن scikit-Learn يجب أن يبذل قصارى جهده في دقة المقايضة مقابل وقت الحساب. ومن غير راضٍ عن الوضع الحالي ، يجب عليه تقديم طلب سحب.