Scikit-learn: float32的euclidean_distances的数值精度
描述
我注意到sklearn.metrics.pairwise.pairwise_distances函数在使用np.float64数组时与np.linalg.norm一致,但是在使用np.float32数组时则不同意。 请参见下面的代码段。
复制步骤/代码
import numpy as np
import scipy
import sklearn.metrics.pairwise
# create 64-bit vectors a and b that are very similar to each other
a_64 = np.array([61.221637725830078125, 71.60662841796875, -65.7512664794921875], dtype=np.float64)
b_64 = np.array([61.221637725830078125, 71.60894012451171875, -65.72847747802734375], dtype=np.float64)
# create 32-bit versions of a and b
a_32 = a_64.astype(np.float32)
b_32 = b_64.astype(np.float32)
# compute the distance from a to b using numpy, for both 64-bit and 32-bit
dist_64_np = np.array([np.linalg.norm(a_64 - b_64)], dtype=np.float64)
dist_32_np = np.array([np.linalg.norm(a_32 - b_32)], dtype=np.float32)
# compute the distance from a to b using sklearn, for both 64-bit and 32-bit
dist_64_sklearn = sklearn.metrics.pairwise.pairwise_distances([a_64], [b_64])
dist_32_sklearn = sklearn.metrics.pairwise.pairwise_distances([a_32], [b_32])
# note that the 64-bit sklearn results agree exactly with numpy, but the 32-bit results disagree
np.set_printoptions(precision=200)
print(dist_64_np)
print(dist_32_np)
print(dist_64_sklearn)
print(dist_32_sklearn)
预期成绩
我希望sklearn.metrics.pairwise.pairwise_distances的结果对于64位和32位都与np.linalg.norm一致。 换句话说,我期望以下输出:
[ 0.0229059506440019884643266578905240749008953571319580078125]
[ 0.02290595136582851409912109375]
[[ 0.0229059506440019884643266578905240749008953571319580078125]]
[[ 0.02290595136582851409912109375]]
实际结果
上面的代码段为我产生了以下输出:
[ 0.0229059506440019884643266578905240749008953571319580078125]
[ 0.02290595136582851409912109375]
[[ 0.0229059506440019884643266578905240749008953571319580078125]]
[[ 0.03125]]
版本号
Darwin-16.6.0-x86_64-i386-64bit
('Python', '2.7.11 | 64-bit | (default, Jun 11 2016, 03:41:56) \n[GCC 4.2.1 Compatible Apple LLVM 6.0 (clang-600.0.57)]')
('NumPy', '1.11.3')
('SciPy', '0.19.0')
('Scikit-Learn', '0.18.1')
所有102条评论
与python 3.5相同的结果:
Darwin-15.6.0-x86_64-i386-64bit
Python 3.5.1 (v3.5.1:37a07cee5969, Dec 5 2015, 21:12:44)
[GCC 4.2.1 (Apple Inc. build 5666) (dot 3)]
NumPy 1.11.0
SciPy 0.18.1
Scikit-Learn 0.17.1
它仅在欧几里德距离处发生,并且可以直接使用sklearn.metrics.pairwise.euclidean_distances复制:
import scipy
import sklearn.metrics.pairwise
# create 64-bit vectors a and b that are very similar to each other
a_64 = np.array([61.221637725830078125, 71.60662841796875, -65.7512664794921875], dtype=np.float64)
b_64 = np.array([61.221637725830078125, 71.60894012451171875, -65.72847747802734375], dtype=np.float64)
# create 32-bit versions of a and b
a_32 = a_64.astype(np.float32)
b_32 = b_64.astype(np.float32)
# compute the distance from a to b using sklearn, for both 64-bit and 32-bit
dist_64_sklearn = sklearn.metrics.pairwise.euclidean_distances([a_64], [b_64])
dist_32_sklearn = sklearn.metrics.pairwise.euclidean_distances([a_32], [b_32])
np.set_printoptions(precision=200)
print(dist_64_sklearn)
print(dist_32_sklearn)
我无法进一步追踪错误。
希望对您有所帮助。
numpy可能使用精度更高的累加器。 是的,看起来像这样
值得修复。
在2017年7月19日上午12:05,“ nvauquie” [email protected]写道:
与python 3.5相同的结果:
达尔文15.6.0-x86_64-i386-64bit
Python 3.5.1(v3.5.1:37a07cee5969,2015年12月5日,21:12:44)
[GCC 4.2.1(Apple Inc.版本5666)(第3点)]
NumPy 1.11.0
科学0.10.1
Scikit-Learn 0.17.1它仅在欧几里德距离处发生,并且可以使用
直接sklearn.metrics.pairwise.euclidean_distances:导入密码
导入sklearn.metrics.pairwise创建彼此非常相似的64位向量a和b
a_64 = np.array([61.221637725830078125,71.60662841796875,-65.7512664794921875],dtype = np.float64)
b_64 = np.array([61.221637725830030025,71.60894012451171875,-65.72847747802734375],dtype = np.float64)创建a和b的32位版本
a_32 = a_64.astype(np.float32)
b_32 = b_64.astype(np.float32)使用sklearn计算64位和32位从a到b的距离
dist_64_sklearn = sklearn.metrics.pairwise.euclidean_distances([a_64],[b_64])
dist_32_sklearn = sklearn.metrics.pairwise.euclidean_distances([a_32],[b_32])np.set_printoptions(精度= 200)
打印(dist_64_sklearn)
打印(dist_32_sklearn)我无法进一步追踪错误。
希望对您有所帮助。-
您收到此消息是因为您已订阅此线程。
直接回复此电子邮件,在GitHub上查看
https://github.com/scikit-learn/scikit-learn/issues/9354#issuecomment-316074315 ,
或使线程静音
https://github.com/notifications/unsubscribe-auth/AAEz65yy8Aq2FcsDAcWHT8qkkdXF_MfPks5sPLu_gaJpZM4OXbpZ
。
如果可能的话,我想做这个
去吧!
所以我认为问题在于我们正在使用sqrt(dot(x, x) - 2 * dot(x, y) + dot(y, y))计算欧几里德距离
因为如果我尝试- (-2 * np.dot(X, Y.T) + (X * X).sum(axis=1) + (Y * Y).sum(axis=1)我会得到np.float32的答案0,而我会得到np.float 64的正确答案。
@jnothman您认为我应该怎么办? 正如我在上面的评论中提到的,问题可能是使用sqrt(dot(x, x) - 2 * dot(x, y) + dot(y, y))计算欧几里得距离
因此,您是说点返回的结果不如乘积之和精确?
不,我要说的是,点返回的结果比乘积之和更精确
-2 * np.dot(X, Y.T) + (X * X).sum(axis=1) + (Y * Y).sum(axis=1)给出输出[[0.]]
而np.sqrt(((X-Y) * (X-Y)).sum(axis=1))给出输出[ 0.02290595]
目前尚不清楚您在做什么,部分原因是您没有发布完全独立的代码段。
快速查看您的上一个帖子,您尝试比较[[0.]]和[0.022...]件事没有相同的尺寸(也许是复制和粘贴问题,但又一次很难知道,因为我们没有有完整的摘要)。
好吧,不好意思
import numpy as np
import scipy
from sklearn.metrics.pairwise import check_pairwise_arrays, row_norms
from sklearn.utils.extmath import safe_sparse_dot
# create 64-bit vectors a and b that are very similar to each other
a_64 = np.array([61.221637725830078125, 71.60662841796875, -65.7512664794921875], dtype=np.float64)
b_64 = np.array([61.221637725830078125, 71.60894012451171875, -65.72847747802734375], dtype=np.float64)
# create 32-bit versions of a and b
X = a_64.astype(np.float32)
Y = b_64.astype(np.float32)
X, Y = check_pairwise_arrays(X, Y)
XX = row_norms(X, squared=True)[:, np.newaxis]
YY = row_norms(Y, squared=True)[np.newaxis, :]
#Euclidean distance computed using product-then-sum
distances = safe_sparse_dot(X, Y.T, dense_output=True)
distances *= -2
distances += XX
distances += YY
print(np.sqrt(distances))
#Euclidean distance computed using (X-Y)^2
print(np.sqrt(row_norms(X-Y, squared=True)[:, np.newaxis]))
输出值
[[ 0.03125]]
[[ 0.02290595136582851409912109375]]
第一种方法是如何通过欧氏距离函数来计算它。
同样要澄清我的意思是,即使我们使用numpy函数进行求和,乘积的精度也较低
是的,我可以复制。 我看到最初做减法
允许保持差异的精度。 做点
乘积,然后减去(或取反),就像我们目前所做的那样,
由于最重要的数字远大于
差异。
当前的实现对于大量的内存使用效率更高
特征。 但是我想欧几里德距离变得越来越无关紧要
在高尺寸中,因此内存由输出数量决定
价值观。
因此,我投票赞成采用比
我们目前拥有d渐近有效的实现。 一个观点,
@ogrisel? @agramfort?
这当然是一个值得关注的问题,因为我们最近允许使用float32
在估算器中更常见。
因此,对于此示例,product-then-sum对于np.float64来说效果很好,因此可能的解决方案是将输入转换为float64,然后计算结果,然后将转换后的结果返回给float32。 我想这会更有效,但不确定是否可以在其他示例中使用。
转换为float64不会比使用内存效率更高
减法。
哦,是的,对此您感到很抱歉,但是我认为使用float64然后执行product-then-sum运算,如果不是内存明智的话,将在计算上更加高效。
而使用乘积之和的原因是为了提高计算效率,而不是存储效率。
可以,但是我不相信有任何理由可以假设它确实是
除了不必实现
中间数组。 假设我们限制绝对工作记忆(例如
分块),为什么要取点积,加倍和减去范数
比减法和平方更有效?
提供基准?
好的,所以我创建了一个python脚本来比较减法-然后-平方和转换为float64然后乘积-然后-求和所花费的时间,事实证明,如果我们选择X和Y作为非常大的向量,那么2个结果就非常不同。 还有@jnothman,您是正确的减法,然后平方更快。
这是我写的脚本,如果有任何问题,请告诉我
import numpy as np
import scipy
from sklearn.metrics.pairwise import check_pairwise_arrays, row_norms
from sklearn.utils.extmath import safe_sparse_dot
from timeit import default_timer as timer
for i in range(9):
X = np.random.rand(1,3 * (10**i)).astype(np.float32)
Y = np.random.rand(1,3 * (10**i)).astype(np.float32)
X, Y = check_pairwise_arrays(X, Y)
XX = row_norms(X, squared=True)[:, np.newaxis]
YY = row_norms(Y, squared=True)[np.newaxis, :]
#Euclidean distance computed using product-then-sum
distances = safe_sparse_dot(X, Y.T, dense_output=True)
distances *= -2
distances += XX
distances += YY
ans1 = np.sqrt(distances)
start = timer()
ans2 = np.sqrt(row_norms(X-Y, squared=True)[:, np.newaxis])
end = timer()
if ans1 != ans2:
print(end-start)
start = timer()
X = X.astype(np.float64)
Y = Y.astype(np.float64)
X, Y = check_pairwise_arrays(X, Y)
XX = row_norms(X, squared=True)[:, np.newaxis]
YY = row_norms(Y, squared=True)[np.newaxis, :]
distances = safe_sparse_dot(X, Y.T, dense_output=True)
distances *= -2
distances += XX
distances += YY
distances = np.sqrt(distances)
end = timer()
print(end-start)
print('')
if abs(ans2 - distances) > 1e-3:
# np.set_printoptions(precision=200)
print(ans2)
print(np.sqrt(distances))
print(X, Y)
break
值得测试它如何随样本数量缩放,而不仅仅是
功能的数量...采用规范可能会受益于计算一些
每个样本一次,而不是每对样本一次
2017年10月20日上午2:39,“ Osaid Rehman Nasir”通知@ github.com
写道:
好的,所以我创建了一个python脚本来比较
减去然后平方并转换为float64然后乘积然后求和
事实证明,如果我们选择X和Y作为非常大的向量,则2
结果有很大的不同。 也是@jnothman https://github.com/jnothman
您是正确的减法,然后平方更快。
这是我写的脚本,如果有任何问题,请告诉我将numpy导入为np
导入密码
从sklearn.metrics.pairwise导入check_pairwise_arrays,row_norms
从sklearn.utils.extmath导入safe_sparse_dot
从timeit导入default_timer作为计时器对于我在范围(9)中:
X = np.random.rand(1,3 *(10 i))。astype(np.float32)Y = np.random.rand(1,3 *(10 i))。astype(np.float32)X,Y = check_pairwise_arrays(X,Y)
XX = row_norms(X,squared = True)[:, np.newaxis]
YY = row_norms(Y,squared = True)[np.newaxis,:]#使用乘积之和求和的欧几里得距离
距离= safe_sparse_dot(X,YT,density_output = True)
距离* = -2
距离+ = XX
距离+ = YYans1 = np.sqrt(距离)
开始= timer()
ans2 = np.sqrt(row_norms(XY,squared = True)[:, np.newaxis])
结束= timer()
如果ans1!= ans2:
打印(结束开始)start = timer() X = X.astype(np.float64) Y = Y.astype(np.float64) X, Y = check_pairwise_arrays(X, Y) XX = row_norms(X, squared=True)[:, np.newaxis] YY = row_norms(Y, squared=True)[np.newaxis, :] distances = safe_sparse_dot(X, Y.T, dense_output=True) distances *= -2 distances += XX distances += YY distances = np.sqrt(distances) end = timer() print(end-start) print('') if abs(ans2 - distances) > 1e-3: # np.set_printoptions(precision=200) print(ans2) print(np.sqrt(distances)) print(X, Y) break-
您收到此邮件是因为有人提到您。
直接回复此电子邮件,在GitHub上查看
https://github.com/scikit-learn/scikit-learn/issues/9354#issuecomment-337948154 ,
或使线程静音
https://github.com/notifications/unsubscribe-auth/AAEz6z5o2Ao_7V5-Lflb4HosMrHCeOrVks5st209gaJpZM4OXbpZ
。
无论如何,您想提交PR,@ ragnerok吗?
是的,您要我做什么?
提供更稳定的实施,以及在
当前的实施方式,理想情况下是表明我们没有失败的基准
在合理的情况下,变化不大。
我想问问是否可以通过向量化找到每对行之间的距离。 我无法考虑如何将其向量化。
您是说成对的行之间的差异(不是距离)? 当然,如果您使用的是numpy数组,则可以这样做。 如果您有形状为(n_samples1,n_features)和(n_samples2,n_features)的数组,则只需将其重塑为(n_samples1,1,n_features)和(1,n_samples2,n_features)并进行减法:
>>> X = np.random.randint(10, size=(10, 5))
>>> Y = np.random.randint(10, size=(11, 5))
X.reshape(-1, 1, X.shape[1]) - Y.reshape(1, -1, X.shape[1])
是的,这真的有帮助😄
我还想问我是否提供了一个更稳定的实现,我将不会使用X_norm_squared和Y_norm_squared。 那么我是否也将它们从参数中删除,还是应该警告它没有任何用处?
我认为它们将被弃用,但我们可能需要首先确保
在任何情况下都不应保留该版本。
我们将在更改此设置时非常小心。 这是一个广泛使用的
长期实施。 我们应该确保不要放慢任何重要的动作
案件。 我们可能需要分块进行操作以避免高内存
用法(这可能由于称为
在功能块内,以最大程度地减少来自
配对距离)。
我真的很想听听其他了解计算的核心开发人员
成本和数值精度... @ ogrisel , @ lesteve ,@ rth ...
2017年11月5日上午5:27,“ Osaid Rehman Nasir”通知@ github.com
写道:
我还想问一下我是否会提供更稳定的实施方案
使用X_norm_squared和Y_norm_squared。 所以我要从
争论还是应该警告它没有任何用处?-
您收到此邮件是因为有人提到您。
直接回复此电子邮件,在GitHub上查看
https://github.com/scikit-learn/scikit-learn/issues/9354#issuecomment-341919282 ,
或使线程静音
https://github.com/notifications/unsubscribe-auth/AAEz63izdpQGDEuW32m8Aob6rrsvV6q-ks5szKyHgaJpZM4OXbpZ
。
但是,如果您打开PR,则更容易进行精确地讨论
好吧,我将打开PR,并使用此功能的非常基本的实现
问题是,对于0.20版本,应该对此做些什么。 是否可以考虑进行一些简单/临时的改进(事件以例如内存使用为代价)?
#11271中提出的解决方案和分析绝对是非常有价值的,但是可能需要更多讨论才能确保这是最佳解决方案。 特别是,我担心以下事实:根据CPU类型等,我们现在在
对于发布@jnothman @amueller @ogrisel,您认为应该如何处理此问题?
稳定性胜过效率。 即使在以下情况下也应解决稳定性问题
效率仍然需要调整。
working_memory的重点是制作带有大样本的轮廓之类的东西
大小工作。 它还提高了效率,但是可以对其进行调整
线。
我坚信我们应该尝试使用以下方法修复euclidean_distances
float32 in。我们假设可以
euclidean_distances以天真的方式在32位上工作。
我同意我们需要修复。 我这里关心的不是效率,而是代码库中增加的复杂性。
退一步,scipy的欧几里得实现似乎是10行C代码,对于32位,只需将它们转换为64位即可。 我知道这不是最快的方法,但从概念上讲很容易理解和理解。 在scikit-learn中,我们使用该技巧使BLAS中的计算速度更快,然后在https://github.com/scikit-learn/scikit-learn/pull/10212中进行了可能的改进,现在提供了可能的欧几里得分块解决方案距离(32位)。
我只是在寻找有关此主题的总体方向的输入(例如,尝试将其上游传播至其他内容)。
scipy似乎并不关心复制数据...
PR后移至0.21。
移除阻滞剂?
sqrt(dot(x, x) - 2 * dot(x, y) + dot(y, y))
如果dot(x,x)和dot(y,y)的大小与dot(x,y)相似,则它在数值上不稳定,这是由于所谓的灾难性抵消。
这不仅影响FP32精度,而且当然会更加突出,并且会更早失效。
这是一个简单的测试用例,它显示了即使双精度也有多糟糕:
import numpy
from sklearn.metrics.pairwise import euclidean_distances
a = numpy.array([[100000001, 100000000]])
b = numpy.array([[100000000, 100000001]])
print "skelarn:", euclidean_distances(a, b)[0,0]
print "correct:", numpy.sqrt(numpy.sum((a-b)**2))
a = numpy.array([[10001, 10000]], numpy.float32)
b = numpy.array([[10000, 10001]], numpy.float32)
print "skelarn:", euclidean_distances(a, b)[0,0]
print "correct:", numpy.sqrt(numpy.sum((a-b)**2))
sklearn两次都计算距离为0,而不是sqrt(2)。
有关方差和协方差的数值问题的讨论-并轻松地延续到这种加速欧几里德距离的方法-可以在这里找到:
埃里希·舒伯特(Erich Schubert)和迈克尔·格茨(Michael Gertz)。
(共)方差的数值稳定并行计算。
在:第30届科学和统计数据库管理国际会议(SSDBM)会议录,意大利博尔扎诺-博岑。 2018,10:1–10:12
实际上,可以从该测试用例中删除y坐标,然后正确的距离就变成1。我发出了一个拉动请求,触发了此数字问题:
XA = np.array([[10000]], np.float32)
XB = np.array([[10001]], np.float32)
assert_equal(euclidean_distances(XA, XB)[0,0], 1)
我认为上面提到的我的论文没有提供解决此问题的方法-只需将欧几里德距离计算为sqrt(sum(power()))即可,它是单次通过且具有合理的精度。 损失在于已经使用了平方,即点(x,x)本身已经失去了精度。
@amueller,因为问题可能比预期的要严重,我建议重新添加阻止程序标签...
感谢这个非常简单的示例。
之所以以这种方式实现,是因为它速度更快。 见下文:
x = np.random.random_sample((1000, 1000))
%timeit euclidean_distances(x,x)
20.6 ms ± 452 µs per loop (mean ± std. dev. of 7 runs, 10 loops each)
%timeit cdist(x,x)
695 ms ± 4.06 ms per loop (mean ± std. dev. of 7 runs, 1 loop each)
尽管两种方法中的运算数量是相同的(第二种运算的数量是1.5倍),但是加速的原因是可以使用经过优化的BLAS库进行矩阵矩阵乘法。
对于scikit-learn中的多个估计器而言,这将是一个巨大的放缓。
是的,但只是3-4位精度FP32带,并与FP64 7-8位不造成实质性的不精确性,不是吗? 特别是,由于此类错误往往会放大...
好吧,我并不是说现在还好。 :)
我是说,我们需要在两者之间找到解决方案。
有一个PR(#11271)建议将其浮在float64上进行计算。 In不能解决float64的问题,但可以为float32提供更好的精度。
您是否有一个示例,该示例使用使用euclidean_distances的估计器会由于精度损失而产生错误的结果?
我当然仍然认为这很重要,应该成为0.21的阻止者。 这是在0.19中针对32位引入的一个问题,这不是一个好的事情。 我希望我们早些时候以0.20的价格解决它,我很高兴,甚至热衷于看到#11271在过渡期间合并。 我知道该PR中的唯一问题是内存效率的环绕优化,这是一个很深的难题。
我们已经有很长时间了,但是总是在float64中。 我知道@ kno10 ,它存在精度问题。 如果这可能是一个问题,您是否有一个很好的快速启发方法可以帮助我们解决问题,并使用速度较慢但解决方案更简单的解决方案?
是的,但是FP32的精度只有3-4位数,而FP64的精度只有7-8位数会引起很大的不准确性,不是吗?
感谢您使用非常简单的示例说明此问题!
但是,我认为这个问题并不像您建议的那么广泛-它主要影响相对于标准而言相互距离较小的样本。
下图说明了2e6个随机样本对的情况,其中每个1D样本的间隔为[-100,100]。 将scikit-learn和scipy实现之间的相对误差绘制为样本之间距离的函数,并通过样本的L2范数进行归一化,即
d_norm(A, B) = d(A, B) / sqrt(‖A‖₂*‖B‖₂)
(不确定是否是正确的参数化,只是为了获得与数据范围有些许差异的结果),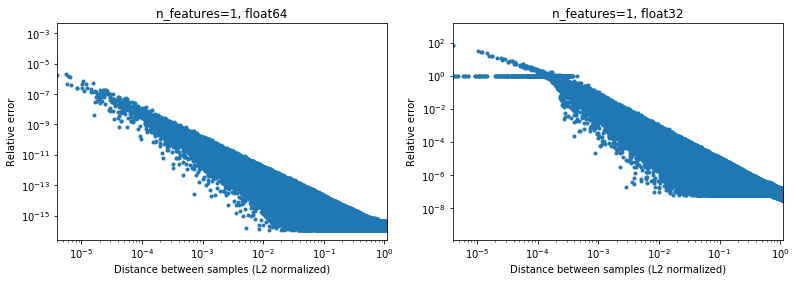
例如,
- 如果使用
[10000]和[10001]则L2归一化距离为1e-4,距离计算的相对误差为64位1e-8,而32位> 1(或1e)绝对值分别为-8和> 1)。 如果是32位,这种情况确实非常糟糕。 - 另一方面,对于
[1]和[10001],相对误差将是32位的〜1e-7或最大可能的精度。
问题是案例1.在ML应用程序中实际发生的频率是多少。
有趣的是,如果我们再次使用均匀随机分布的2D模式,将很难找到非常接近的点,
当然,实际上,我们的数据不会被统一采样,但是对于任何分布,由于维数的诅咒,随着维数的增加,任意两点之间的距离将缓慢收敛到非常相似的值(不同于0)。 尽管这是一个常见的ML问题,但即使在相对较低的尺寸下,它也可以在某种程度上缓解此准确性问题。 在n_features=5的结果下方,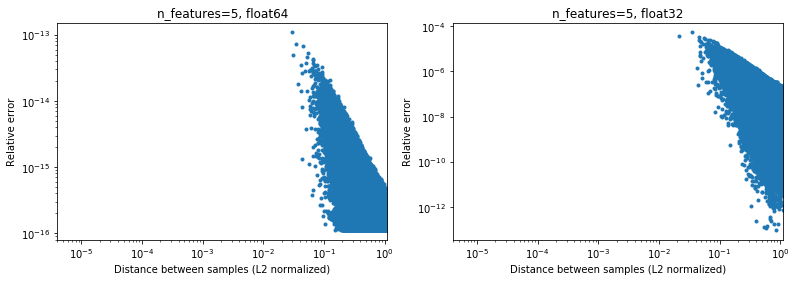 。
。
因此,对于至少在64位中居中的数据,在实践中可能并不是什么大问题(假设有2个以上的功能)。 50倍的计算速度(如上所示)可能是值得的(64位)。 当然,可以总是将1e6加到在[-1,1]中归一化的某些数据上,并说结果不准确,但是我认为,这同样适用于许多数值算法,并且使用在6th中表示的数据有效数字只是在寻找麻烦。
(以上代码可在此处找到)。
就我所知,使用dot(x,x)+ dot(y,y)-2 都可能会遇到相同的问题,但是您最好问一些真正的数字专家为了这。 输入数据
我无法说出使用这种方法将float32转换为float64会带来多少开销。 至少对于float32,据我了解,进行所有计算并将点积存储为float64应该很好。
恕我直言,性能提升(不是指数级的,只是一个常数)并不值得(会意外地咬住您)精度损失,正确的方法是不要使用此有问题的技巧。 但是,很可能可以进一步优化执行“传统”计算的代码,例如使用AVX。 因为sum((xy) * 2)几乎很难在AVX中实现。至少,我建议将方法重命名为approximate_euclidean_distances ,因为有时精度较低(两个值越接近越差,*最初
@rth感谢您的插图。 但是,如果您尝试优化,例如将x朝向某个最佳值,该怎么办。 最优值很可能不会为零(如果它将始终是您的数据中心,那么生活将会很棒),并且最终您为梯度等计算的增量可能会有一些很小的差异。
类似地,在聚类中,聚类并非都将其中心都接近于零,但是特别是在许多聚类中,具有几位数的x≈中心是完全可能的。
总的来说,我同意这个问题需要解决。 无论如何,我们都需要尽快记录当前实现的精度问题。
总的来说,尽管我不认为这种讨论应该在scikit-learn中进行。 欧几里得距离用于科学计算和IMO scipy邮件列表的各个领域,是讨论它的更好场所:该社区在此类数值精度问题上也有更多经验。 实际上,我们这里是一个快速但有些近似的算法。 我们可能必须在短期内实施一些修复方法,但是从长远来看,最好能在此做出贡献。
对于32位, https://github.com/scikit-learn/scikit-learn/pull/11271可能确实是一个解决方案,我只是不太热衷于通过库进行多级分块,因为这会增加代码的复杂性,并希望确保没有更好的方法。
感谢您的回复@ kno10! (我的上述评论尚未考虑),稍后我会回复。
是的,收敛到原点之外的某个点可能是一个问题。
恕我直言,性能提升(不是指数级的,只是一个常数)并不值得(会意外地咬住您)精度损失,正确的方法是不要使用此有问题的技巧。
那么在64位计算中,> 10倍的速度降低将对用户产生非常实际的影响。
但是,很可能可以进一步优化执行“传统”计算的代码,例如使用AVX。 因为sum((xy)** 2)几乎很难在AVX中实现。
尝试使用numba(应使用SSE)进行快速幼稚的实现,
@numba.jit(nopython=True, fastmath=True)
def pairwise_distance_naive(A, B):
n_samples_a, n_features_a = A.shape
n_samples_b, n_features_b = B.shape
assert n_features_a == n_features_b
distance = np.empty((n_samples_a, n_samples_b), dtype=A.dtype)
for i in range(n_samples_a):
for j in range(n_samples_b):
psum = 0.0
for k in range(n_features_a):
psum += (A[i, k] - B[j, k])**2
distance[i, j] = math.sqrt(psum)
return distance
到目前为止,它的速度与scipy cdist相似(但我不是numba专家),并且不确定fastmath 。
使用dot(x,x)+ dot(y,y)-2 * dot(x,y)版本
仅供参考,我们目前正在大致执行以下操作(因为上面的符号中未显示尺寸),
def quadratic_pairwise_distance(A, B):
A2 = np.einsum('ij,ij->i', A, A)
B2 = np.einsum('ij,ij->i', B, B)
return np.sqrt(A2[:, None] + B2[None, :] - 2*np.dot(A, B.T))
现在, einsum和dot现在都使用BLAS。 我想知道,除了使用BLAS之外,这实际上是否还可以执行与上述第一个版本相同数量的数学运算。
我想知道,除了使用BLAS之外,这实际上是否还可以执行与上述第一个版本相同数量的数学运算。
否。[[(x-y) * 2.sum())执行* n_samples_x * n_samples_y * n_features *(1个减法+ 1个加法+ 1个乘法)
而xx + yy -2x.y执行
n_samples_x * n_samples_y * n_features *(1加+ 1乘) 。
两个版本之间的操作数之比为2/3。
经过以上讨论,
- 进行PR以选择性地允许精确计算欧式距离https://github.com/scikit-learn/scikit-learn/pull/12136
- 一些WIP以查看我们是否可以检测并缓解https://github.com/scikit-learn/scikit-learn/pull/12142中的问题点
对于32位,我们仍然需要通过IMO以某种形式合并https://github.com/scikit-learn/scikit-learn/pull/11271 ,上述PR在某种程度上与其正交。
仅供参考:在修正OPTICS中的某些问题并刷新测试以使用ELKI的参考结果时,这些操作会因metric="euclidean"而失败,但会导致metric="minkowski" 。 数值差异大到足以导致不同的处理顺序(仅降低阈值是不够的)。
我真的没有赶上这一步,但令我惊讶的是,目前没有解决方案。 这似乎是一个非常普通的计算,似乎我们正在重新发明轮子。 有没有人尝试过接触更广泛的科学计算社区?
尚未,但我同意我们应该的。 我在scipy中发现的唯一问题是https://github.com/scipy/scipy/pull/2815和链接的问题。
我觉得@jeremiedbb可能有个主意?
不幸的是,还不令人满意:(
我们希望依靠高度优化的库来进行此类计算,就像使用带有BLAS库(例如OpenBLAS或MKL)的线性代数一样。 但是欧几里德距离不是其中的一部分。 点技巧是一种依靠BLAS 3级矩阵-矩阵乘法子例程进行此操作的尝试。 但这并不精确,也无法使用相同的方法使其更加精确。 我们必须降低速度或精度方面的期望。
我认为在某些情况下,不是必须要求全精度,而保持快速方法是可以的。 这是将距离用于“查找最近”任务的时候。 当点之间的距离与标准距离相比较小时,快速方法中会出现精度问题(对于float 32,比率为〜<1e-4,对于float64为〜<1e-8)。 首先,要使这种情况发生,数据集必须非常密集。 然后要产生排序错误,您需要使两个最接近的点在几乎相同的距离内。 而且,在这种情况下,从机器学习的角度来看,两者都将导致几乎相同的良好拟合。
在上述情况下,我们可以采取一些措施来降低这些错误排序的频率(降低至0?)。 在成对距离argmin的情况下。 我们可以将失序移动到不是最接近的点。 本质上是使用不需要规范之一来找到argmin的事实,请参见comment 。 它有2个优点。 它更健壮(到目前为止,我还没有发现错误的顺序),并且甚至更快,因为它避免了一些计算。
一个缺点,仍然是在相同情况下,如果最后我们想要到最接近点的实际距离,则无法使用通过上述方法计算出的距离。 它们仅是部分计算的,而且还是不精确。 我们需要重新计算从每个点到最近点的距离。 但这很快,因为每个点只有一个距离可以计算。
我不知道我上面描述的内容涵盖了sklearn中的euclidean_distances的所有用例。 但是我建议在任何可以应用的地方都这样做。 为此,我们可以向euclidean_distances添加一个新参数,以仅计算必要的部分,以便与argmin链接。 然后在pairwise_distances_argmin和pairwise_distances_argmin_min中使用它(重新计算后者末尾的实际最小距离)。
当我们无法做到这一点时,请退回到缓慢而精确的位置,或者添加一个开关,如#12136中所示。
我们可以尝试对其进行一些优化,以降低性能下降的原因,因为我同意这似乎不是最佳选择。 我对此有一些想法。
继续使用BLAS的另一种可能性是将axpy与nrm2组合在一起,但这远非最优。 两者都是BLAS 1级功能,并且涉及一个副本。 仅在尺寸> 100时才会更快。
理想情况下,我们希望欧氏距离包含在BLAS中...
最后,还有另一种解决方案,包括上播。 这是在#11271中为float32完成的。 优点是速度仅为当前速度的一半,并且可以保持精度。 但是,它不能解决float64的问题。 也许我们可以找到一种在Cython中为float64做类似事情的方法。 我不知道确切如何,但是使用2个float64数字来模拟float128。 我可以尝试一下是否可行。
理想情况下,我们希望欧氏距离包含在BLAS中...
图书馆会考虑吗? 如果OpenBLAS做到这一点,我们将处在一个非常好的状况中……
另外,我们这样做与BLAS所做的确切区别是什么? 检测CPU功能并确定要使用哪种实现,或者类似的东西? 还是只是为更多样化的体系结构编写了版本?
还是只花更多时间/精力花在编写高效的实现上?
这很有趣:快速不稳定方法的另一种实现,但声称比sklearn快得多:
https://github.com/droyed/eucl_dist
(虽然大声笑根本无法解决这个问题)
这是茱莉亚(Julia)的工作: https :
它允许设置精度阈值以强制重新计算。
回答我自己的问题:OpenBLAS对于每个处理器(不是体系结构!)看起来像是手写的程序集,并具有启发式的特性,可以针对不同的问题大小选择内核。 因此,我认为将它放入openblas并没有找到要编写/优化所有这些内核的问题...
感谢您的额外想法!
在部分回应中,
我们希望依靠高度优化的库来进行此类计算,就像使用带有BLAS库(例如OpenBLAS或MKL)的线性代数一样。
是的,我也希望我们可以在BLAS中做更多的事情。 上一次我在标准BLAS API中看起来什么都没有,看上去很接近(但是那时我不是这些方面的专家)。 BLIS可能会提供更大的灵活性,但是由于默认情况下我们不使用它,因此它的用法有些局限(尽管numpy可能有朝一日https://github.com/numpy/numpy/issues/7372)
这是julia的工作:允许设置精度阈值以强制重新计算。
很高兴知道!
我们是否应该为上面链接的更快的近似计算打开一个单独的问题? 似乎很有趣
他们在x2-x4的CPU上的加速可能是由于https://github.com/scikit-learn/scikit-learn/pull/10212 。
一旦我们研究了足够多的问题以提出合理的解决方案(然后可能会将其移植),我宁愿在scipy上提出一个问题,因为我认为欧几里德距离已经足够基本,对于ML以外的许多人来说都应该感兴趣(同时,在那里有人们的意见,例如对准确性问题的看法将是自欺欺人的)。
高达60倍,对不对?
这很有趣:快速不稳定方法的另一种实现,但声称比sklearn快得多
哼哼对此不确定。 他们正在基准测试%timeit pairwise_distances(a,b, 'sqeuclidean') ,它使用scipy的那个。 他们应该做%timeit pairwise_distances(a,b, 'euclidean', metric_params={'squared': True})并且他们的提速效果不如:)
如讨论中前面所述,sklearn的速度可以比scipy快35倍
是的,使用metric="euclidean" (而不是squeclidean ),它们的基准只能提高约30%,
In [1]: from eucl_dist.cpu_dist import dist
... import numpy as np
In [4]: rng = np.random.RandomState(1)
... a = rng.rand(1000, 300)
...b = rng.rand(1000, 300)
In [7]: from sklearn.metrics.pairwise import pairwise_distances
In [8]: %timeit pairwise_distances(a, b, 'sqeuclidean')
214 ms ± 2.06 ms per loop (mean ± std. dev. of 7 runs, 1 loop each)
In [9]: %timeit pairwise_distances(a, b)
27.4 ms ± 2.48 ms per loop (mean ± std. dev. of 7 runs, 10 loops each)
In [10]: from eucl_dist.cpu_dist import dist
In [11]: %timeit dist(a, b, matmul='gemm', method='ext', precision='float32')
20.8 ms ± 330 µs per loop (mean ± std. dev. of 7 runs, 10 loops each)
In [12]: %timeit dist(a, b, matmul='gemm', method='ext', precision='float64')
20.4 ms ± 222 µs per loop (mean ± std. dev. of 7 runs, 10 loops each)
图书馆会考虑吗? 如果OpenBLAS做到这一点,我们将处在一个非常好的状况中……
听起来并不简单。 BLAS是线性代数例程的一组规范,并且有几种实现。 我不知道他们在增加原始规格中没有的新功能方面有多开放。 为此,也许blis会更开放,但是如前所述,它现在不是默认值。
开业https://github.com/scikit-learn/scikit-learn/issues/12600在sqeuclidean VS euclidean在处理pairwise_distances 。
我需要一些明确的信息。 我们是否要让pairwise_distances接近(在all_close的意义上)同时适用于'euclidean'和'sqeuclidean'?
这有点棘手。 因为x接近y并不意味着x²接近y²。 在平方时会丢失精度。
上面链接的julia解决方法非常有趣,并且易于实现。 但是我怀疑它对于“ sqeuclidean”不起作用。 我怀疑您必须在下面设置阈值方式才能获得所需的精度。
设置非常低的阈值的问题在于,它会引起大量重新计算和大量性能下降。 但是,这可以通过数据集的维度来缓解。 相同的阈值将触发高维中较少的重新计算(距离较大)。
也许我们可以有2种实现方式并根据数据集的维度进行切换。 慢速但安全的低维数(在那种情况下scipy和sklearn之间没有太大区别),而快速+门限的高维数。
这将需要一些基准来找到何时进行切换并设置阈值,但这可能是一线希望:)
这是scipy和sklearn之间速度比较的一些基准。 基准将sklearn.metrics.pairwise.euclidean_distances(X,X)与scipy.spatial.distance.cdist(X,X)的所有大小的X进行比较。 样本数从2(3)(16)增至2 13(8192),特征数从2(3)(1)增至2 13(8192)。
每个单元格中的值是sklearn与scipy的加速比,即低于1 sklearn较慢,高于1 sklearn较快。
第一个基准是使用BLAS的MKL实现和一个核心。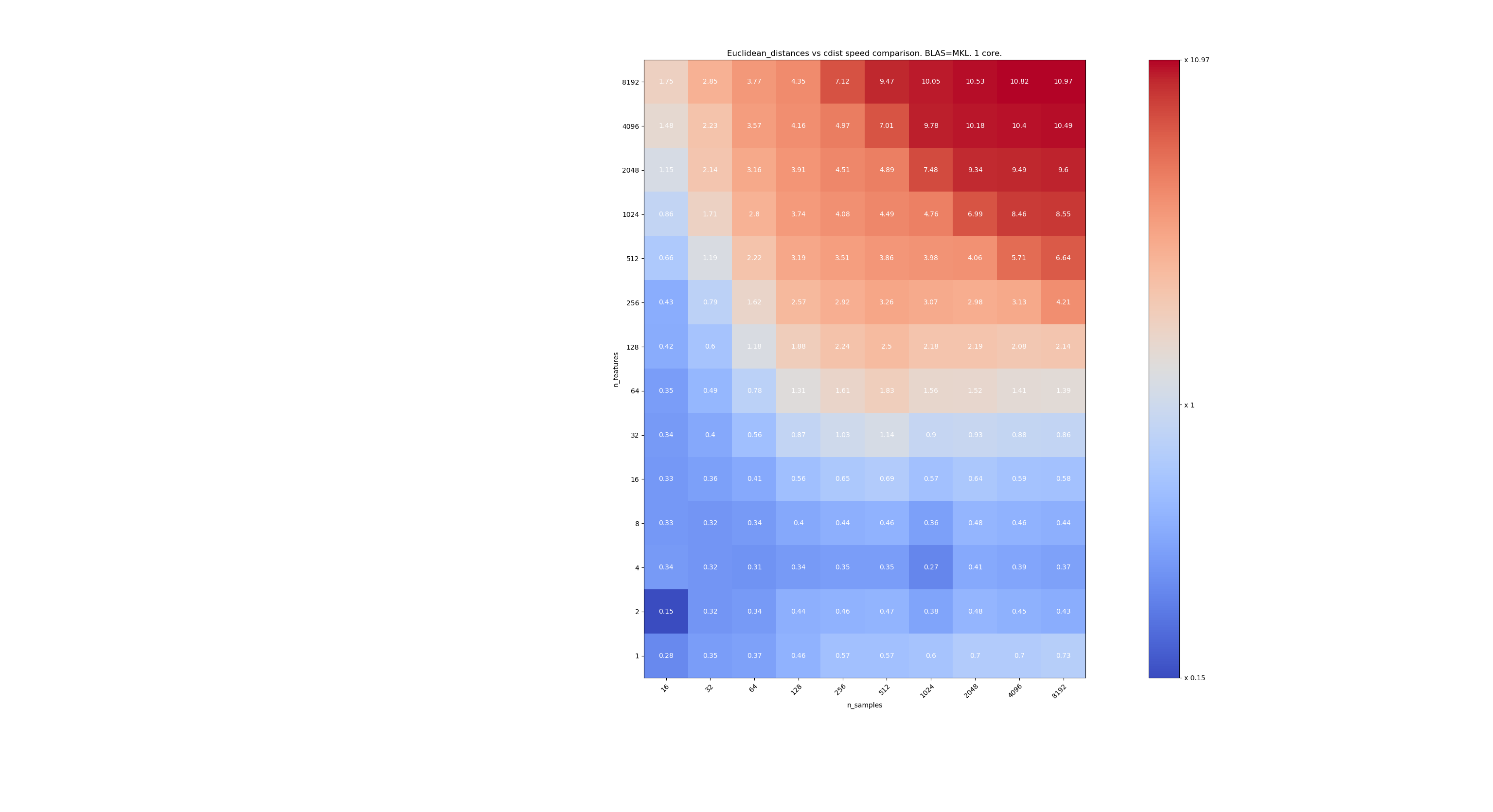
第二个是使用BLAS的OpenBLAS实现和一个核心。 只是要检查MKL和OpenBLAS是否具有相同的行为。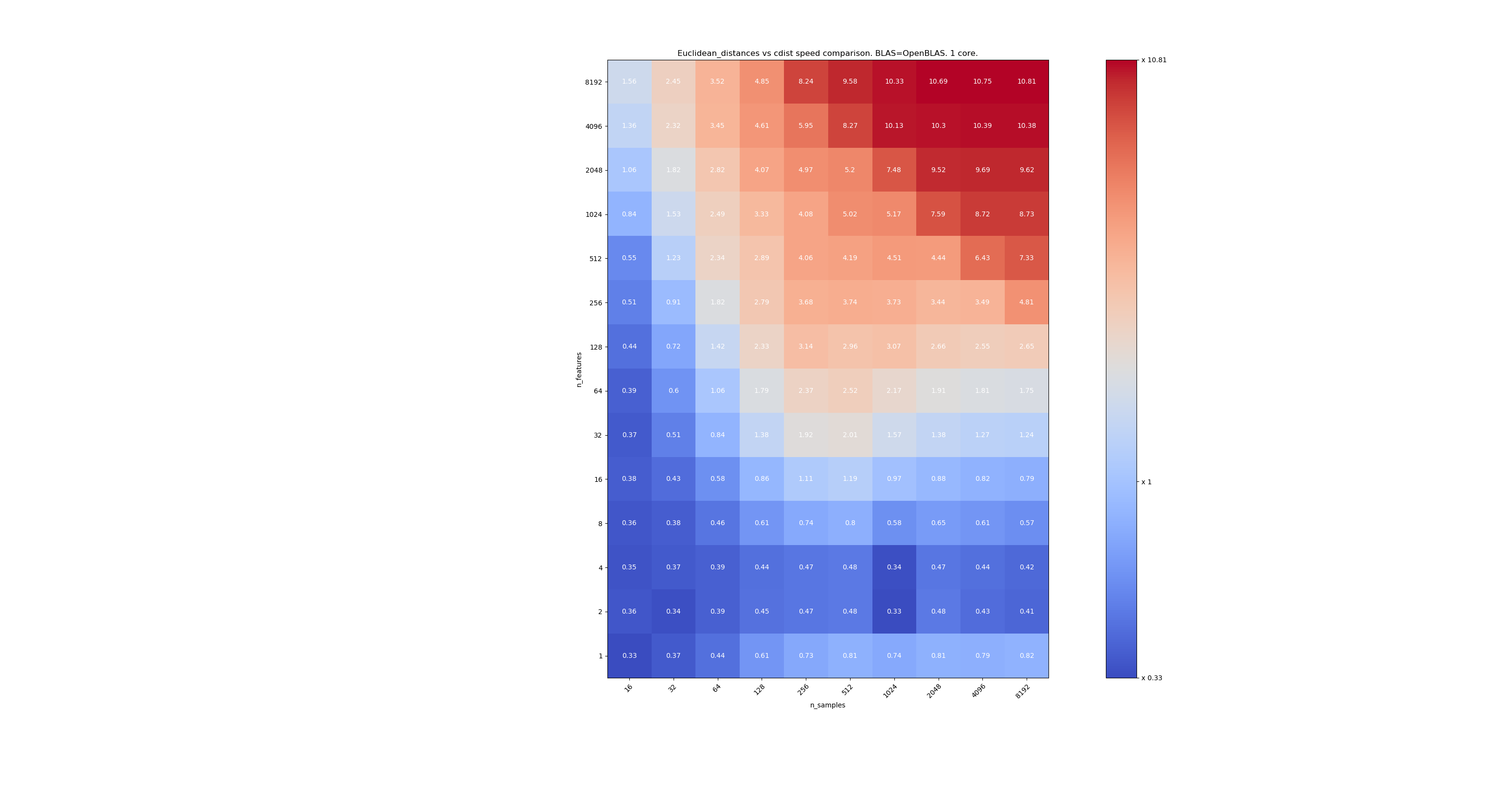
第三个是使用BLAS和4个核心的MKL实现。 问题是euclidean_distances是通过BLAS LEVEL 3函数并行化的,但是cdist只使用BLAS LEVEL 1函数。 有趣的是,它几乎不会改变边界。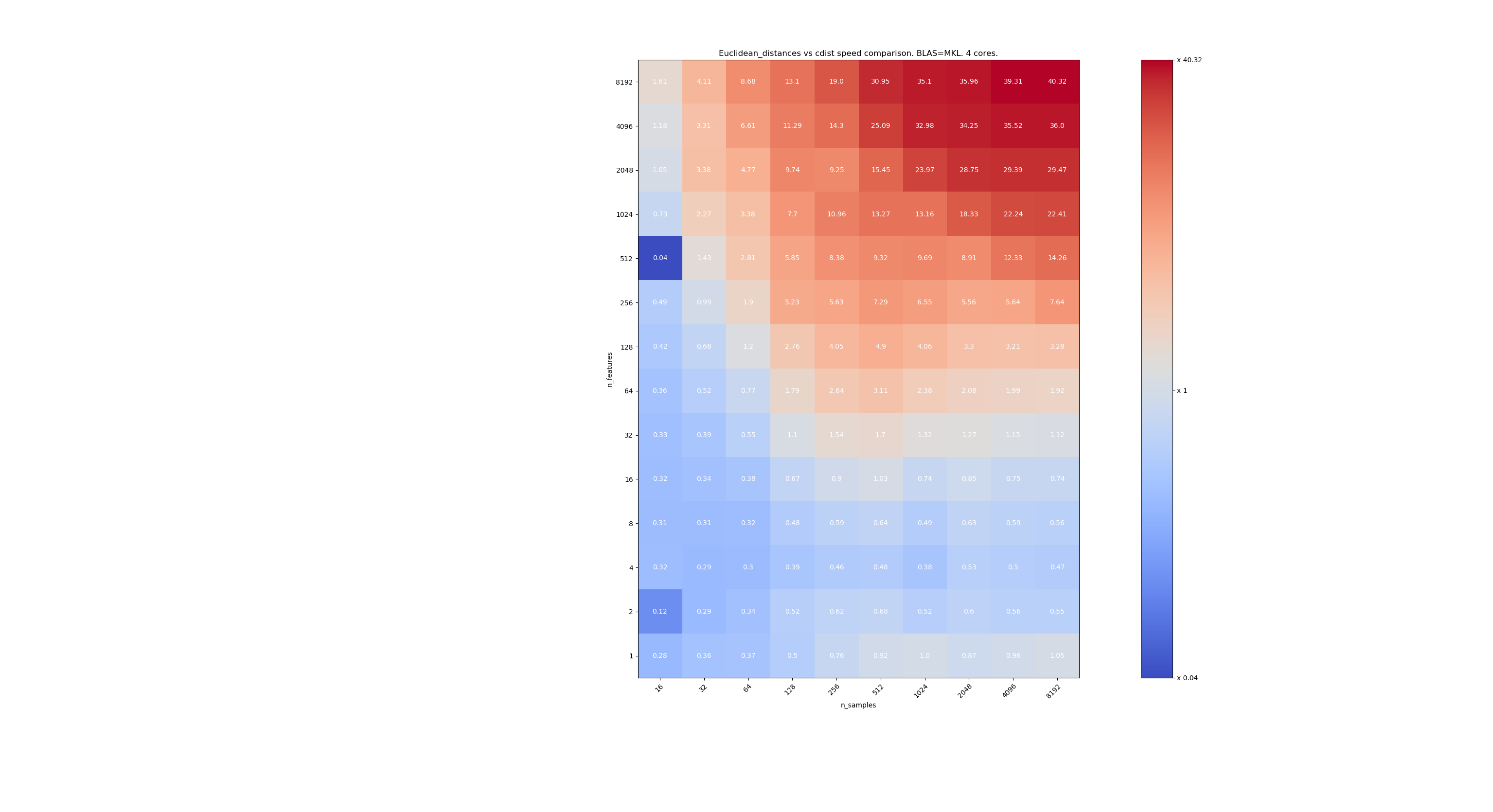
当n_samples不太低(> 100)时,似乎边界约为32个要素。 当n_features <32时,我们可以决定使用cdist;当n_features> 32时,我们可以决定使用euclidean_distances。这样做速度更快,并且没有精度问题。 这也具有以下优点:当n_features较小时,茱莉亚阈值会导致大量重新计算。 使用cdist可以避免这种情况。
当n_features> 32时,我们可以保留euclidean_distances实现,并用julia阈值进行更新。 添加阈值不应使euclidean_distances减慢太多,因为功能数量足够高,因此只需要进行几次重新计算即可。
@jeremiedbb太好了,谢谢您的分析。 结论听起来对我来说是一个很好的方法。
哦,我想这都是float64的全部,对吧? 我们如何处理float32? 总是不高兴吗? 是否对32个以上的功能感兴趣?
我没有仔细阅读评论(很快就会),只是供参考,float64有其局限性,请参阅#12128
@ qinhanmin2014是的,float64精度有局限性,但就我所知,它足以产生可靠的fp32结果。 问题是,向上传输到fp64的参数实际上比使用scipy的cdist便宜。
从上述基准中可以看出,即使是多核BLAS也通常不会更快。 对于高维数据(超过64维;在此之前,IMHO的价值通常不值得付出努力),这似乎大部分适用-并且由于欧氏距离在密集的高维数据中不是那么可靠,因此用例IMHO并不是最重要的。 许多用户的尺寸小于10。 在这些情况下,cdist通常看起来更快?
哦,我想这都是float64的全部,对吧?
实际上,它同时适用于float32和float64(我的意思是非常相似)。 我建议在n_features <32时始终使用cdist。
问题是,向上传输到fp64的参数实际上比使用scipy的cdist便宜。
上传速度将减慢〜2倍,因此我猜n_features = 64左右。
许多用户的尺寸小于10。
但是并不是所有人,因此我们仍然需要找到高维数据的解决方案。
@jeremiedbb的分析非常好!
对于低维数据,那么一定要使用cdist。
此外,FYI scipy的cdist将float32转换为float64 https://github.com/scipy/scipy/issues/8771#issuecomment -384015674,我不确定这是由于准确性问题还是其他原因。
总的来说,我认为将https://github.com/scikit-learn/scikit-learn/pull/12601#pullrequestreview -176076355中建议的“ algorithm”参数添加到euclidean_distance默认设置为“无”,因此也可以通过全局选项进行设置,例如https://github.com/scikit-learn/scikit-learn/pull/12136。
Eigen3中还有一种有趣的方法可以计算稳定的规范: https ://eigen.tuxfamily.org/dox/StableNorm_8h_source.html(我还没有真正地抱怨过)
很好的解释,增进了我的理解
我们在sprint上尚未取得任何进展,我们可能应该...而且@rth不在今天。
如果您设定时间,我可以远程加入。 也许在下午开始?
总结一下情况,
对于欧几里得距离计算中的精度问题,
- 在低维情况下,如上面的@jeremiedbb所示,我们可能应该使用cdist
- 在高维情况和float32中,我们可以选择
- 分块,以64位计算距离并进行级联
- 在精度成为问题的情况下退回到cdist(如何解决一个开放的问题-接触scipy可能是有用的https://github.com/scikit-learn/scikit-learn/issues/9354#issuecomment-438522881 )
然后,存在欧几里得,斯夸里丹,敏可夫斯基等之间不一致的所有问题。
关于精度,我和@jeremiedbb , @amueller进行了快速聊天,主要是因为他的专业知识而挤牛奶Jeremie。 他认为,我们不必担心在float64高维中ML上下文中的不稳定性问题。 杰里米(Jeremie)还暗示,很难找到一种有效的测试来确定是否已经返回了良好的结果(参见#12142)。
因此,我认为我们对@rth的先前评论(对于float32的向上转换)感到满意。 由于cdist也向上转换为float64,因此,如果我们希望在低调float32中进行较少的复制,则可以重新实现cdist以使用float32(但使用float64累加器?),或者可以使用分块。
@Celelibi是否要更改#
并确定此问题后,我认为我们应该使sqeuclidean和minkowski({0,1}中的p)使用我们的实现。 我们尚未与NearestNeighbors讨论差异。 另一个冲刺:)
在sprint上进行了快速讨论之后,我们最终采用以下方式:
在高维情况下(> 32或> 64选择最佳):当块为float32时,按块向上转换为float64,并保持“快速”方法。 对于此类数据,float64上的数值问题几乎可以忽略不计(我将为此提供基准)
在低维情况下:在sklearn中实现安全的计算(而不是因为上流而使用scipy cdist)。
(很容易将float32转换为0.20.3也很诱人)
这是scipy和sklearn之间速度比较的一些基准。
[...片段...]
这很有趣。 我实际上并没有期待这个结果。 我重新做了基准测试,发现了非常相似的结果。 除了我主张降低决策界限。 我的基准测试将建议8个功能。

错误的代价是不对称的。 cdist仅对持续时间少于几秒钟的计算更好,并且当功能数量增加时,它会变慢。 因此,如有疑问,最好使用BLAS实现。
编辑:该基准测试是针对float64的,但我还发现将float32矩阵转换为float64几乎只占总时间的百分之几,并且不会改变结论。
我注意到阈值取决于运行基准测试的计算机。 我怀疑这可能与AVX指令有关。 我意识到我发布的基准测试是在没有AVX2指令的机器上运行的,只有AVX。 在装有AVX2的计算机上,我得到了与您相似的结果。
但是,问题不仅仅关乎性能,还关乎精度,而且在尺寸较小的情况下更可能出现精度问题。 也许16是一个很好的折衷。 你怎么看 ?
关于此讨论,我想说我们需要对准确性进行基准测试,以便做出明智的决定。
但是,就您的PR而言,准确性不再是问题。 但是以稍微昂贵一些的计算为代价。 因此,该阈值可能应该通过基准PR来确定。
标杆精度不是那么容易。 因为困难的情况不会得到统一分配。
如果在极端情况下未检测到它,则可能会出现问题。 通常,您将希望在可承受的CPU限制内保证数值精度。
但是,就像在其他地方提到的那样,当使用已知问题方程式10000和10001与fp32时,具有10000000.01和10000000.00的单个特征应该足以触发fp64的数值不稳定性。 具有1024个功能,请尝试
>>> import sklearn.metrics.pairwise as sk, scipy.spatial.distance as sp
>>> X = [[10000.01] * 1024, [10000.00] * 1024]
>>> print(sk.euclidean_distances(X,X), "\n", sp.cdist(X,X))
[[ 0. 0.31895195]
[ 0.31895195 0. ]]
[[ 0. 0.32]
[ 0.32 0. ]]
(使用0.19.1)。正确的距离是0.32。
如您所见,数值不稳定性会随着要素数量的增加而恶化(除非您的数据稀疏)。 此处,FP64的结果精度不到两位数。
13410不能解决此特定情况。 即float64 +高尺寸。
它修复了float32。
但是我们决定对于float64 +高亮度,我们将其保持不变,因为准确性问题极不可能发生,并且并不真正适用于机器学习用例。
在您的示例中,X [0]和X [1]的范数等于320000.32和320000,距离为0.32,即其范数的1e-6倍。 在机器学习中,16个有效数字(在float64中)并不都相关。
但是我们决定对于float64 +高亮度,我们将其保持不变,因为准确性问题极不可能发生,并且并不真正适用于机器学习用例。
我对此会比较温和。 降低尺寸是ML中通常的第一步。 MDS可以用于此目的,并且大量使用了欧氏距离矩阵。
如果有人想提高float64大小写的准确性,可以使用两种float来表示中间结果。 尽管我认为它已经超出了scikit-learn的范围。
ftp://ftp.math.ethz.ch/users/wpp/CELL/qd.pdf
我不清楚。 我并不是说高维数据不适用于机器学习。 我是说float64中发生的那种精度问题涉及距离比其标准小6个数量级的点。 如此精确的精度对现实的机器学习模型毫无意义
在机器学习中,16个有效数字(在float64中)并不都相关。
我一点也不相信这通常是正确的。
在这个例子中,我们损失了16位数字中的15位。 我同意是否要使用一半的精度,但是我们没有这种关系。 由于测量精度的原因,从FP64向下投射到FP32造成的损失通常是可以容忍的。 例如,使用FP32的消费级GPU的速度要比使用FP64的速度快得多(尽管在某些情况下,它们现在允许FP32数据和FP64累加器),并且对于神经网络推断,您现在甚至可以看到int8。 但这并不适用于所有地方。
例如,在k均值聚类,存在群集在其装置显着不同的假设(和我们不知道事先的装置),因此我们在精度损失这里。 如果您有许多集群,则与分离相比,它们的某些规范可能会很大。
此外,在第一次初始迭代之后,它的距离通常很小,导致一个点切换到另一个群集。 此处精度的损失会影响结果,并可能导致不稳定。
现在考虑具有多个变量的时间序列片段的k均值。
随着数据大小的增加,我们必须假设到最近邻居的距离变小,并且除非您的范数为0,否则它们最终将小于矢量范数并引起问题。 因此,随着数据集大小的增加,这种情况可能会变得更加严重。 维度的诅咒说,最大和最小距离越来越相似。 因此,为了计算正确的最近邻排名,我们可能需要在高维数据中具有较高的精度。 在20news数据集上,最小的非零距离约为0.02(规范均为1)。 但这仅仅是1万个实例,而且内容相当多样化。 现在假设数据集是关于近重复检测...
我不确定这种“不太可能”发生在ML中……当然,它不会影响到所有人。
当我说“在机器学习中,16个有效数字(在float64中)并不都相关。”时,我不是在说计算的距离,而是在说数据X。
在机器学习中,您的数据来自一个度量,并且没有精确到第9位的度量(在粒子物理学中很少)。
因此,在您的10000000.01和10000000.00的示例中,当您对X值的不确定性更大时,您将如何重视0.01的距离?
对于KMeans,首先有一种方法可以克服很大一部分精度损失。 当您寻找观测x的最近中心时,您无需在距离计算中添加x的范数,从而避免了在大多数情况下的灾难性抵消。
然后,kmeans基于欧几里得距离进行聚类。 但是您不知道这是否是收集数据的准确方法。 实际上,您的数据以这种方式群集的可能性为0。 Kmeans估计如何对数据进行聚类,并且绝对不能认为位于两个聚类前沿的点肯定是属于另一个。 您对2个簇的相同距离处的点有何解释? 我的观点是两个集群应该只是一个集群,或者KMeans并不是对我的数据进行集群的最佳算法(或者甚至kmeans也使我对如何对数据进行集群有了一个很好的主意,但我知道集群的边界并不相关)。
仅使用“ | b | ^ 2-2ab”不会带来灾难性的取消-但是造成差异的数字精度也会降低。 结果与之后将a的范数添加到每个距离中的结果相同; 如果距离远小于a的范数,那么您将损失精度,这是可以避免的,而无需使用BLAS hacks的传统方法进行计算即可避免。
因此,您实际上无法以这种方式克服数值问题!
K均值是一个优化问题。 因此,此类黑客行为可能意味着sklearn只能找到比其他工具更糟糕的解决方案。 并且如前所述,这也可能导致不稳定。 在最坏的情况下,这可能会导致sklearn kmeans遍历相同的状态,直到max_iter没有任何改善(假设tol = 0,如果您想找到局部最优值),这在理论上是不可能的。
在k均值收敛之前,您不能对两个聚类具有“相同”距离的点说太多。 在下一次迭代中,方法可能已经移动,并且差异可能变得更大而且很重要!
我不是k均值的忠实拥护者,因为它在嘈杂的数据上效果不佳。 但是有一些变种可以更好地处理这种情况。 但是,尽管如此,如果您使用它,则可能应该尝试获得完整的质量(这就是为什么我也总是使用tol=0 )并且不要使其变得比必要的情况更糟。 进行适当的计算足够便宜(而且如上所述,问题随着数据大小而变得更加严重-因此对于小数据,运行时间通常并不重要,对于大数据集,精度变得更重要)。
取决于应用程序, 10000000.01和10000000.00之间的差异可能很重要。 正如我之前所展示的,如果您使用多个功能,则问题会更早出现。 对于fp32,只有一个功能只有10000和10001,而有100个功能的只有101对100,我想:
如前所述,均值可能具有您不希望失去的物理意义。 如果您有开氏温度数据,则不需要0:1缩放比例或将其居中; 那会破坏你的比例规模。 现在,例如,您要比较某些钢材冷却时温度的时间序列,并确定冷却过程是否会影响钢材的可靠性。 您的温度可能超过700 K,并且如果要分析冷却过程,则时间序列可能具有数百个数据点。 即使时间序列的长度只有5位输入精度(0.01K),也会出现数字问题。 您可能会再次只得到1-2位数字的结果。 我认为,如果您具有这种灾难性的影响,您不能仅仅排除精度对ML的重要性。 如果您可以保证始终获得精度,例如16位数中的10位数,则情况有所不同。 在这里您无法做到这一点,在最坏的情况下,您可能拥有0位数(这就是灾难性的原因)。
在机器学习中,您的数据来自一个度量,并且没有精确到第9位的度量(在粒子物理学中很少)。
真实世界中的原始值很少具有这种准确性,是的。 但是ML不仅限于这种输入。 可能需要将ML应用到数学问题上,例如将MDS应用到魔方状难题的图形上,或者将您的大量RL特工玩吃豆子找到成功的策略进行聚类。
即使信息的最初来源是真实世界,也可能会进行一些中途处理,使大多数数字与聚类算法相关。 就像某个函数的梯度下降的结果一样,该函数的参数在现实世界中被统计采样。
我实际上想知道为什么我们仍在讨论这个问题。 我猜我们都同意scikit-learn应该在权衡精度与计算时间之间尽力而为。 对当前状态不满意的人应该提交拉取请求。
仅使用“ | b | ^ 2-2ab”不会带来灾难性的取消-但是造成差异的数字精度也会降低。 结果与之后将a的范数添加到每个距离中的结果相同; 如果距离远小于a的范数,那么您将损失精度,这是可以避免的,而无需使用BLAS hacks的传统方法进行计算即可避免。
因此,您实际上无法以这种方式克服数值问题!
精度有所损失,但是它不会造成灾难性的取消(至少在a和b接近时),并且可以证明距离(不是距离)的相对误差很小。
对于KMeans,您只想查找最接近的中心,则您有足够的精度来保持正确的排序。 如果最后需要惯性,则可以使用精确的公式来计算每个点到其聚类中心的距离。
此外,KMeans并不是凸优化问题,因此,即使让它以tol = 0进行运行直到收敛,您仍然会遇到局部最小值,该最小值可能与全局最小值相差甚远(即使使用kmeans ++初始化)。 因此,我宁愿使用不同的init和相当少量的迭代多次运行kmeans。 您将有更好的机会达到更好的局部最小值。 然后,您可以重新运行最佳的,直到收敛。
与实际距离相比,相对误差可以任意大,从而导致错误的最近邻居。 考虑| a |²= | b |²= 1的情况,例如在tf-idf上。 假设向量非常接近。 那么ab也接近1,这时您已经失去了很多精度。
正如我上面所写,即使您没有灾难性的取消,也仍然存在错误。 考虑8位数的精度。 实际距离可以是0.000012345678,并且可以使用FP32和常规的欧几里得距离以八位数字进行计算。 但是,使用此等式,您改为计算值ab = 0.99998765432,使用FP32最多将其截断为大约0.99998765,因此在此示例中不必要地损失了三位数的精度。 损失与灾难性事件一样大。 如果距离远小于标准距离,则使用此方法可能会导致精度下降。
是的,kmeans不是凸的。 但是随后,您将希望至少找到一个局部最优值,并且不要因为精度太低而陷入困境(甚至因为产生的误差表现不规律而振荡)。 因此,您至少有机会在行为良好的情况下并进行多次尝试来找到全局变量。
我对这次讨论表示赞赏,但我们真正需要的是一个不比停止将事物上浮到float64之前所做的解决方案还要糟糕的解决方案。 从这个意义上讲, @ Celelibi的转换解决方案就足够了。 在较小的尺寸上使用精确的解决方案是对我们以前所做工作的进一步改进。
对于将来的版本,您是否更有信心有效地检测何时可以考虑在高维中进行精确计算?
我已经运行了一个基准来评估带有随机数的float64案例的平均准确性。 我比较了3种算法: neumaier_sum((x-y)**2) , numpy.sum((x-y)**2)和X2 - 2*X.dot(Y.T) + Y2.T 。 使用mpmath以256位的精度获得了要比较的确切结果。
X和Y具有100个样本和可变数量的要素,并以-2到2之间的随机数填充。
在以下gif上,每个特征数量(在1到200之间)有一张图像。 在每个图像上,每个点表示10000对矢量X和Y之间的平方欧几里德距离的相对误差。 为了提高可读性,相对误差乘以2 ^ 53,大致对应于ULP单位。
上面的曲线是近似分布(使用内核密度估计)。

请注意,为便于阅读,这些图是在6 ULP处剪切的。 它显示的是平均情况,而不是最坏的情况。 扩展公式的误差会变得非常大。
我对此结果的分析是,平均而言,扩展公式的相对误差可能非常大,几乎没有特征,但很快变得类似于差值和numpy和。 阈值介于5到10个特征之间。
我目前也在尝试寻找扩展公式以及病理示例错误的上限。
我认为@ kno10担心的是,我们经常对以下情况感兴趣
点不是随机分布的,而是彼此靠近或具有单位
规范。
确实,但是我需要确信,在实践中,它并不是完整的BS。 ^^
为了完成以上注释:公式x²+y²-2ab的相对误差似乎没有限制。 除非我的分析错误,否则当x和y彼此接近时,相对误差可能高达2^(52*2) 。 至少在理论上。 实际上,我发现的最坏情况是相对误差2^52+1 。
>>> a, b = (0xfffffec4d6282+1) * 2.0**(511-52), 0xfffffec4d6282 * 2.0**(511-52)
>>> a, b
(6.703903473040778e+153, 6.7039034730407766e+153)
>>> exactdiff = (a-b)**2
>>> exactdiff
2.2158278651204453e+276
>>> computeddiff = a**2 + b**2 - 2*a*b
>>> computeddiff
-9.9792015476736e+291
>>> abs((computeddiff - exactdiff) / exactdiff)
4503599627370497.0
>>> bin(int(abs((computeddiff - exactdiff) / exactdiff)))
'0b10000000000000000000000000000000000000000000000000001'
翻转结果的符号实际上将使其更接近事实。 这是我能找到的最生动的示例,但是实际上更改a和b的值的指数并不会改变相对误差。
>>> a, b = (0xfffffec4d6282+1) * 2.0**(-52), 0xfffffec4d6282 * 2.0**(-52)
>>> a, b
(0.9999999266202912, 0.999999926620291)
>>> exactdiff = (a-b)**2
>>> computeddiff = a**2 + b**2 - 2*a*b
>>> abs((computeddiff - exactdiff) / exactdiff)
4503599627370497.0
我认为ULP中的直方图比具有ULP内部误差分布的上述动画更有意义。 因此0个ULP错误和1个ULP错误“尽其所能”。 2由于sqrt,ULP不可避免。 我认为任何较大的错误都值得调查。
只要精确大,使用(computed - exact) / exact是合理的。 但是,一旦我们要获得确切值的数值挑战,就变得非常不稳定。 在这种情况下,可能值得使用(computed-exact)/norm来代替,即,将距离计算的精度与输入数据进行比较,而不是与导出的距离进行比较。
如果我们有两个仅相差1个ULP的一维值,则2个ULP的误差似乎很大; 但是我们已经达到输入数据分辨率,因此结果非常不稳定。
请注意,对于多维尺寸,我们可能会在输入数据中获得更高的分辨率。
考虑类型(1, 1e-16)与(1, 2e-16)输入数据。 例如,如果我们在输入数据中具有常量属性,例如MNIST中的白色像素。
使用基于差异的方程式会很好,但是点版本会遇到麻烦,不是吗? 这就是为什么一维实验可能不足以对此进行研究的原因。
我认为ULP中的直方图比具有ULP内部误差分布的上述动画更有意义。
我不确定我是否会看到您的表述。 每个特征数量和每个算法只有一个直方图。 除了3D图或动画,我无能为力。
只要精确大,使用
(computed - exact) / exact是合理的。 但是,一旦我们要获得确切值的数值挑战,就变得非常不稳定。
在这种情况下,我不确定您所说的不稳定是什么意思。 精确值应使用使其精确的所有方法来计算。
(说到这,我也应该在绘图中以任意精度计算相对误差,而不是与精确的舍入结果进行比较。我更新绘图后,奇怪的波消失了。)
在这种情况下,可能值得使用
(computed-exact)/norm来代替,即,将距离计算的精度与输入数据进行比较,而不是与导出的距离进行比较。
如果我正确理解您的想法,您宁愿将绝对误差与输入数据的大小进行比较。 使用向量范数作为输入幅度的合计度量。 而标准相对误差将其与精确结果的大小进行比较。
我认为使用该指标可以尝试捕获算法有多少错误。 但实际上由于某些原因,它似乎并不是特别有用。
- 它并没有真正说出结果中有多少个数字不准确。
- 实际上,大多数算法的得分都小于1e-15。 即使是扩展的公式(基于点的算法)也将具有由5 ULP(输入)限制的分数(粗略估算,我没有写出完整的证明)。
- 并且由于这两个指标只是绝对误差
computed - exact的重新缩放版本,因此在对相同输入进行评估时,它们将以相同顺序对算法进行排名。
因此,它与通常的相对误差相同,只是值解释不太有用(IMO)。
考虑类型
(1, 1e-16)与(1, 2e-16)输入数据。 例如,如果我们在输入数据中具有常量属性,例如MNIST中的白色像素。
使用基于差异的方程式会很好,但是点版本会遇到麻烦,不是吗? 这就是为什么一维实验可能不足以对此进行研究的原因。
基于点的算法将具有1的相对误差,这意味着该误差与精确结果一样大,因此结果的任何数字都不正确。 并且您的指标的值将为1e-16这意味着相对于矢量范数的标度,只有第16个数字处于关闭状态。
我不确定您要在此示例中显示什么。
如果我们仍然关注float64的euclidean_distances的精度,最好在新问题中总结此讨论,因为这里有100条评论。
最有用的评论
真实世界中的原始值很少具有这种准确性,是的。 但是ML不仅限于这种输入。 可能需要将ML应用到数学问题上,例如将MDS应用到魔方状难题的图形上,或者将您的大量RL特工玩吃豆子找到成功的策略进行聚类。
即使信息的最初来源是真实世界,也可能会进行一些中途处理,使大多数数字与聚类算法相关。 就像某个函数的梯度下降的结果一样,该函数的参数在现实世界中被统计采样。
我实际上想知道为什么我们仍在讨论这个问题。 我猜我们都同意scikit-learn应该在权衡精度与计算时间之间尽力而为。 对当前状态不满意的人应该提交拉取请求。