Scikit-learn: Числовая точность euclidean_distances с float32
Описание
Я заметил, что функция sklearn.metrics.pairwise.pairwise_distances согласуется с np.linalg.norm при использовании массивов np.float64, но не соглашается при использовании массивов np.float32. См. Фрагмент кода ниже.
Шаги / код для воспроизведения
import numpy as np
import scipy
import sklearn.metrics.pairwise
# create 64-bit vectors a and b that are very similar to each other
a_64 = np.array([61.221637725830078125, 71.60662841796875, -65.7512664794921875], dtype=np.float64)
b_64 = np.array([61.221637725830078125, 71.60894012451171875, -65.72847747802734375], dtype=np.float64)
# create 32-bit versions of a and b
a_32 = a_64.astype(np.float32)
b_32 = b_64.astype(np.float32)
# compute the distance from a to b using numpy, for both 64-bit and 32-bit
dist_64_np = np.array([np.linalg.norm(a_64 - b_64)], dtype=np.float64)
dist_32_np = np.array([np.linalg.norm(a_32 - b_32)], dtype=np.float32)
# compute the distance from a to b using sklearn, for both 64-bit and 32-bit
dist_64_sklearn = sklearn.metrics.pairwise.pairwise_distances([a_64], [b_64])
dist_32_sklearn = sklearn.metrics.pairwise.pairwise_distances([a_32], [b_32])
# note that the 64-bit sklearn results agree exactly with numpy, but the 32-bit results disagree
np.set_printoptions(precision=200)
print(dist_64_np)
print(dist_32_np)
print(dist_64_sklearn)
print(dist_32_sklearn)
Ожидаемые результаты
Я ожидаю, что результаты sklearn.metrics.pairwise.pairwise_distances будут соответствовать np.linalg.norm как для 64-битных, так и для 32-битных версий. Другими словами, я ожидаю следующего результата:
[ 0.0229059506440019884643266578905240749008953571319580078125]
[ 0.02290595136582851409912109375]
[[ 0.0229059506440019884643266578905240749008953571319580078125]]
[[ 0.02290595136582851409912109375]]
Фактические результаты
Приведенный выше фрагмент кода дает мне следующий результат:
[ 0.0229059506440019884643266578905240749008953571319580078125]
[ 0.02290595136582851409912109375]
[[ 0.0229059506440019884643266578905240749008953571319580078125]]
[[ 0.03125]]
Версии
Darwin-16.6.0-x86_64-i386-64bit
('Python', '2.7.11 | 64-bit | (default, Jun 11 2016, 03:41:56) \n[GCC 4.2.1 Compatible Apple LLVM 6.0 (clang-600.0.57)]')
('NumPy', '1.11.3')
('SciPy', '0.19.0')
('Scikit-Learn', '0.18.1')
Все 102 Комментарий
Те же результаты с python 3.5:
Darwin-15.6.0-x86_64-i386-64bit
Python 3.5.1 (v3.5.1:37a07cee5969, Dec 5 2015, 21:12:44)
[GCC 4.2.1 (Apple Inc. build 5666) (dot 3)]
NumPy 1.11.0
SciPy 0.18.1
Scikit-Learn 0.17.1
Это происходит только с евклидовым расстоянием и может быть воспроизведено напрямую с помощью sklearn.metrics.pairwise.euclidean_distances :
import scipy
import sklearn.metrics.pairwise
# create 64-bit vectors a and b that are very similar to each other
a_64 = np.array([61.221637725830078125, 71.60662841796875, -65.7512664794921875], dtype=np.float64)
b_64 = np.array([61.221637725830078125, 71.60894012451171875, -65.72847747802734375], dtype=np.float64)
# create 32-bit versions of a and b
a_32 = a_64.astype(np.float32)
b_32 = b_64.astype(np.float32)
# compute the distance from a to b using sklearn, for both 64-bit and 32-bit
dist_64_sklearn = sklearn.metrics.pairwise.euclidean_distances([a_64], [b_64])
dist_32_sklearn = sklearn.metrics.pairwise.euclidean_distances([a_32], [b_32])
np.set_printoptions(precision=200)
print(dist_64_sklearn)
print(dist_32_sklearn)
Я не смог отследить ошибку дальше.
Надеюсь, это поможет.
numpy может использовать аккумулятор более высокой точности. да это похоже на это
заслуживает исправления.
19 июля 2017 г. в 12:05 "nvauquie" [email protected] написал:
Те же результаты с python 3.5:
Дарвин-15.6.0-x86_64-i386-64bit
Python 3.5.1 (v3.5.1: 37a07cee5969, 5 декабря 2015 г., 21:12:44)
[GCC 4.2.1 (Apple Inc., сборка 5666) (точка 3)]
NumPy 1.11.0
SciPy 0.18.1
Scikit-Learn 0.17.1Это происходит только с евклидовым расстоянием и может быть воспроизведено с помощью
непосредственно sklearn.metrics.pairwise.euclidean_distances:импортный scipy
импортировать sklearn.metrics.pairwiseсоздать 64-битные векторы a и b, очень похожие друг на друга
a_64 = np.array ([61.221637725830078125, 71.60662841796875, -65.7512664794921875], dtype = np.float64)
b_64 = np.array ([61.221637725830078125, 71.60894012451171875, -65.72847747802734375], dtype = np.float64)создать 32-битные версии a и b
a_32 = a_64.astype (np.float32)
b_32 = b_64.astype (np.float32)вычислить расстояние от a до b с помощью sklearn, как для 64-битных, так и для 32-битных
dist_64_sklearn = sklearn.metrics.pairwise.euclidean_distances ([a_64], [b_64])
dist_32_sklearn = sklearn.metrics.pairwise.euclidean_distances ([a_32], [b_32])np.set_printoptions (точность = 200)
печать (dist_64_sklearn)
печать (dist_32_sklearn)Я не смог отследить ошибку дальше.
Надеюсь, это поможет.-
Вы получаете это, потому что подписаны на эту ветку.
Ответьте на это письмо напрямую, просмотрите его на GitHub
https://github.com/scikit-learn/scikit-learn/issues/9354#issuecomment-316074315 ,
или отключить поток
https://github.com/notifications/unsubscribe-auth/AAEz65yy8Aq2FcsDAcWHT8qkkdXF_MfPks5sPLu_gaJpZM4OXbpZ
.
Я бы хотел поработать над этим, если возможно
Действуй!
Поэтому я думаю, что проблема заключается в том, что мы используем sqrt(dot(x, x) - 2 * dot(x, y) + dot(y, y)) для вычисления евклидова расстояния.
Потому что, если я попробую - (-2 * np.dot(X, Y.T) + (X * X).sum(axis=1) + (Y * Y).sum(axis=1) я получу 0 для np.float32, а для np.float 64 - правильный ответ.
@jnothman Что мне тогда делать? Как упоминалось в моем комментарии выше, проблема, вероятно, заключается в вычислении евклидова расстояния с использованием sqrt(dot(x, x) - 2 * dot(x, y) + dot(y, y))
Значит, вы говорите, что точка возвращает менее точный результат, чем произведение затем сумма?
Нет, я пытаюсь сказать, что точка возвращает более точный результат, чем произведение затем сумма.
-2 * np.dot(X, Y.T) + (X * X).sum(axis=1) + (Y * Y).sum(axis=1) дает результат [[0.]]
а np.sqrt(((X-Y) * (X-Y)).sum(axis=1)) дает результат [ 0.02290595]
Непонятно, что вы делаете, отчасти потому, что вы не публикуете полностью автономный сниппет.
Бегло просматривая ваш последний пост, две вещи, которые вы пытаетесь сравнить, [[0.]] и [0.022...] не имеют одинаковых размеров (возможно, проблема копирования и вставки, но опять же трудно понять, потому что мы не есть полный фрагмент).
Хорошо, извини, я плохо
import numpy as np
import scipy
from sklearn.metrics.pairwise import check_pairwise_arrays, row_norms
from sklearn.utils.extmath import safe_sparse_dot
# create 64-bit vectors a and b that are very similar to each other
a_64 = np.array([61.221637725830078125, 71.60662841796875, -65.7512664794921875], dtype=np.float64)
b_64 = np.array([61.221637725830078125, 71.60894012451171875, -65.72847747802734375], dtype=np.float64)
# create 32-bit versions of a and b
X = a_64.astype(np.float32)
Y = b_64.astype(np.float32)
X, Y = check_pairwise_arrays(X, Y)
XX = row_norms(X, squared=True)[:, np.newaxis]
YY = row_norms(Y, squared=True)[np.newaxis, :]
#Euclidean distance computed using product-then-sum
distances = safe_sparse_dot(X, Y.T, dense_output=True)
distances *= -2
distances += XX
distances += YY
print(np.sqrt(distances))
#Euclidean distance computed using (X-Y)^2
print(np.sqrt(row_norms(X-Y, squared=True)[:, np.newaxis]))
ВЫВОД
[[ 0.03125]]
[[ 0.02290595136582851409912109375]]
Первый метод - это то, как это вычисляется функцией евклидова расстояния.
Также, чтобы прояснить, что я имел в виду выше, был тот факт, что сумма-затем-произведение имеет более низкую точность, даже когда мы используем для этого функции numpy.
Да, я могу повторить это. Я вижу, что сначала вычитание
позволяет сохранить точность разницы. Делаем точку
продукт, а затем вычитание (или отрицание и добавление), как мы сейчас делаем,
теряет эту точность, поскольку наиболее значимые цифры намного больше, чем
различия.
Текущая реализация более эффективна с точки зрения памяти для большого количества
функции. Но я полагаю, что евклидова дистанция становится все более неуместной.
в больших размерах, поэтому в памяти преобладает количество выходных
значения.
Поэтому я голосую за принятие более стабильной в численном отношении реализации по сравнению с
d-асимптотически эффективная реализация, которая у нас есть. Мнение,
@ogrisel? @agramfort?
И это, конечно, вызывает большую озабоченность, поскольку недавно мы разрешили float32s
быть более распространенным среди оценщиков.
Таким образом, для этого примера продукт-затем-сумма отлично работает для np.float64, поэтому возможным решением может быть преобразование ввода в float64, затем вычисление результата и возврат результата, преобразованного обратно в float32. Я думаю, это было бы более эффективно, но не уверен, что это сработает для другого примера.
преобразование в float64 не будет более эффективным в использовании памяти, чем
вычитание.
О да, вы правы, извините за это, но я думаю, что использование float64 и последующее выполнение «произведение затем сумма» было бы более эффективным с точки зрения вычислений, если не с точки зрения памяти.
И причина использования продукта затем суммы заключалась в большей вычислительной эффективности, а не в эффективности памяти.
конечно, но я не думаю, что есть основания предполагать, что это на самом деле
более эффективен с точки зрения вычислений, за исключением того, что не требуется реализовывать
промежуточный массив. Предполагая, что мы ограничиваем абсолютную рабочую память (например,
разбиение на части), зачем брать скалярное произведение, удваивать и вычитать нормы
быть намного эффективнее, чем вычитание и возведение в квадрат?
Приведи тесты?
Хорошо, поэтому я создал скрипт python для сравнения времени, затраченного на вычитание, затем возведение в квадрат и преобразование в float64, затем произведение затем сумма, и оказывается, что если мы выберем X и Y как очень большие векторы, тогда 2 результата будут очень разными. . Также @jnothman, вы были правы, вычитание, а затем возведение в квадрат быстрее.
Вот сценарий, который я написал, если возникнут проблемы, дайте мне знать
import numpy as np
import scipy
from sklearn.metrics.pairwise import check_pairwise_arrays, row_norms
from sklearn.utils.extmath import safe_sparse_dot
from timeit import default_timer as timer
for i in range(9):
X = np.random.rand(1,3 * (10**i)).astype(np.float32)
Y = np.random.rand(1,3 * (10**i)).astype(np.float32)
X, Y = check_pairwise_arrays(X, Y)
XX = row_norms(X, squared=True)[:, np.newaxis]
YY = row_norms(Y, squared=True)[np.newaxis, :]
#Euclidean distance computed using product-then-sum
distances = safe_sparse_dot(X, Y.T, dense_output=True)
distances *= -2
distances += XX
distances += YY
ans1 = np.sqrt(distances)
start = timer()
ans2 = np.sqrt(row_norms(X-Y, squared=True)[:, np.newaxis])
end = timer()
if ans1 != ans2:
print(end-start)
start = timer()
X = X.astype(np.float64)
Y = Y.astype(np.float64)
X, Y = check_pairwise_arrays(X, Y)
XX = row_norms(X, squared=True)[:, np.newaxis]
YY = row_norms(Y, squared=True)[np.newaxis, :]
distances = safe_sparse_dot(X, Y.T, dense_output=True)
distances *= -2
distances += XX
distances += YY
distances = np.sqrt(distances)
end = timer()
print(end-start)
print('')
if abs(ans2 - distances) > 1e-3:
# np.set_printoptions(precision=200)
print(ans2)
print(np.sqrt(distances))
print(X, Y)
break
стоит проверить, как он масштабируется с количеством образцов, а не только с
количество функций ... принятие норм может иметь преимущество в вычислении некоторых
вещи один раз на образец, а не один раз на пару образцов
20 октября 2017 г., 2:39, "Осаид Рехман Насир" [email protected]
написал:
Хорошо, поэтому я создал скрипт Python, чтобы сравнить время, затраченное на
вычитание, возведение в квадрат и преобразование в float64, затем произведение затем сумма
и оказывается, что если мы выберем X и Y как очень большие векторы, то 2
результаты очень разные. Также @jnothman https://github.com/jnothman
Вы были правы, вычитание - возведение в квадрат быстрее.
Вот сценарий, который я написал, если возникнут проблемы, дайте мне знатьимпортировать numpy как np
импортный scipy
из sklearn.metrics.pairwise импорт check_pairwise_arrays, row_norms
из sklearn.utils.extmath import safe_sparse_dot
from timeit импортировать default_timer как таймердля i в диапазоне (9):
X = np.random.rand (1,3 * (10 i)). Astype (np.float32)Y = np.random.rand (1,3 * (10 i)). Astype (np.float32)X, Y = массивы_контрольных пар (X, Y)
XX = row_norms (X, в квадрате = True) [:, np.newaxis]
YY = row_norms (Y, в квадрате = True) [np.newaxis,:]# Евклидово расстояние, вычисляемое с использованием метода произведение затем суммы
расстояния = safe_sparse_dot (X, YT, density_output = True)
расстояния * = -2
расстояния + = XX
расстояния + = YYans1 = np.sqrt (расстояния)
start = timer ()
ans2 = np.sqrt (row_norms (XY, в квадрате = True) [:, np.newaxis])
конец = таймер ()
если ans1! = ans2:
печать (конец-начало)start = timer() X = X.astype(np.float64) Y = Y.astype(np.float64) X, Y = check_pairwise_arrays(X, Y) XX = row_norms(X, squared=True)[:, np.newaxis] YY = row_norms(Y, squared=True)[np.newaxis, :] distances = safe_sparse_dot(X, Y.T, dense_output=True) distances *= -2 distances += XX distances += YY distances = np.sqrt(distances) end = timer() print(end-start) print('') if abs(ans2 - distances) > 1e-3: # np.set_printoptions(precision=200) print(ans2) print(np.sqrt(distances)) print(X, Y) break-
Вы получаете это, потому что вас упомянули.
Ответьте на это письмо напрямую, просмотрите его на GitHub
https://github.com/scikit-learn/scikit-learn/issues/9354#issuecomment-337948154 ,
или отключить поток
https://github.com/notifications/unsubscribe-auth/AAEz6z5o2Ao_7V5-Lflb4HosMrHCeOrVks5st209gaJpZM4OXbpZ
.
в любом случае, не могли бы вы отправить PR, @ragnerok?
да, конечно, что ты хочешь, чтобы я сделал?
обеспечить более стабильную реализацию, а также тест, который не прошел бы
текущая реализация, и в идеале тест, показывающий, что мы не теряем
многое от сдачи, в разумных случаях.
Я хотел спросить, можно ли найти расстояние между каждой парой строк с помощью векторизации. Не могу думать, как это сделать векторизованным.
Вы имеете в виду разницу (а не расстояние) между парами строк? Конечно, вы можете это сделать, если работаете с массивами numpy. Если у вас есть массивы с фигурами (n_samples1, n_features) и (n_samples2, n_features), вам просто нужно изменить его форму на (n_samples1, 1, n_features) и (1, n_samples2, n_features) и выполнить вычитание:
>>> X = np.random.randint(10, size=(10, 5))
>>> Y = np.random.randint(10, size=(11, 5))
X.reshape(-1, 1, X.shape[1]) - Y.reshape(1, -1, X.shape[1])
Да, спасибо, что действительно помогло 😄
Я также хотел спросить, предлагаю ли я более стабильную реализацию, я не буду использовать X_norm_squared и Y_norm_squared. Так что, убрать их тоже из аргументов или предупредить, что это бесполезно?
Я думаю, что они больше не рекомендуются, но, возможно, нам нужно сначала убедиться, что
нет случая, когда мы должны хранить эту версию.
мы будем очень осторожны при изменении этого. это широко используется и
многолетняя реализация. мы должны быть уверены, что не замедлили важные
случаи. нам может потребоваться выполнить операцию по частям, чтобы избежать высокой памяти
использование (что, возможно, усложняется тем, что это называется
внутри функций, которые блокируются, чтобы минимизировать изъятие выходной памяти из
попарные расстояния).
Я бы очень хотел услышать от других разработчиков ядра, которые знают о вычислительных
затраты и числовая точность ... @ogrisel , @lesteve , @rth ...
5 ноября 2017 г., 5:27, "Осаид Рехман Насир" [email protected]
написал:
Я также хотел спросить, предоставлю ли я более стабильную реализацию, я не буду
используя X_norm_squared и Y_norm_squared. Так что я убираю их из
аргументы тоже или я должен предупредить, что это бесполезно?-
Вы получаете это, потому что вас упомянули.
Ответьте на это письмо напрямую, просмотрите его на GitHub
https://github.com/scikit-learn/scikit-learn/issues/9354#issuecomment-341919282 ,
или отключить поток
https://github.com/notifications/unsubscribe-auth/AAEz63izdpQGDEuW32m8Aob6rrsvV6q-ks5szKyHgaJpZM4OXbpZ
.
но обсуждать будет проще, если открыть PR
Хорошо, тогда я открою PR с очень простой реализацией этой функции.
Вопрос в том, что с этим делать для версии 0.20. Могут ли быть рассмотрены какие-то простые / временные улучшения (например, за счет использования памяти)?
Решение и анализ, предложенные в # 11271, безусловно, очень ценны, но может потребоваться дополнительное обсуждение, чтобы убедиться, что это оптимальное решение. В частности, меня беспокоит тот факт, что сейчас у нас есть незавершенное обсуждение оптимальной глобальной рабочей памяти в https://github.com/scikit-learn/scikit-learn/issues/11506 в зависимости от типа процессора и т. Д., В то время как это добавит еще один уровень разбиения на части, и сложность всего будет немного контролировать ИМО. Но, может быть, это просто я ищу второе мнение.
Как вы думаете, что нужно сделать с этой проблемой для выпуска @jnothman @amueller @ogrisel ?
Стабильность важнее эффективности. Проблемы со стабильностью следует устранять, даже если
эффективность все еще требует доработки.
Working_memory фокусировался на создании таких вещей, как силуэт с большим образцом
размеры работают. Это также повысило эффективность, но это можно уменьшить
линия.
Я твердо уверен, что мы должны попытаться исправить euclidean_distances с помощью
float32 в. Мы разобрали его в 0.19, предполагая, что можем сделать
euclidean_distances наивно работает на 32-битной версии.
Я согласен, что нам нужно исправить. Меня беспокоит не эффективность, а дополнительная сложность базы кода.
Сделав шаг назад, евклидова реализация scipy, похоже, представляет собой 10 строк кода C, а для 32-битной версии просто приведите их к 64-битной. Я понимаю, что он не самый быстрый, но концептуально его легко понять и понять. В scikit-learn мы используем трюк для ускорения вычислений в BLAS, затем возможны улучшения в https://github.com/scikit-learn/scikit-learn/pull/10212, а теперь возможное разбитое решение для евклидова расстояние в 32 бита.
Я просто ищу информацию о том, каким должно быть общее направление по этой теме (например, попробуйте передать некоторые из них в scipy и т. Д.).
scipy, похоже, не заботится о копировании данных ...
Перейдите к 0,21 после PR.
Убрать блокиратор?
sqrt(dot(x, x) - 2 * dot(x, y) + dot(y, y))
численно нестабильно, если точка (x, x) и точка (y, y) имеют такую же величину, что и точка (x, y), из-за того, что известно как катастрофическая отмена .
Это не только влияет на точность FP32, но, конечно, более заметно и выйдет из строя намного раньше.
Вот простой тестовый пример, который показывает, насколько это плохо даже с двойной точностью:
import numpy
from sklearn.metrics.pairwise import euclidean_distances
a = numpy.array([[100000001, 100000000]])
b = numpy.array([[100000000, 100000001]])
print "skelarn:", euclidean_distances(a, b)[0,0]
print "correct:", numpy.sqrt(numpy.sum((a-b)**2))
a = numpy.array([[10001, 10000]], numpy.float32)
b = numpy.array([[10000, 10001]], numpy.float32)
print "skelarn:", euclidean_distances(a, b)[0,0]
print "correct:", numpy.sqrt(numpy.sum((a-b)**2))
sklearn вычисляет здесь оба раза расстояние, равное 0, а не sqrt (2).
Обсуждение численных вопросов для дисперсии и ковариации - и это тривиально переносится на этот подход ускорения евклидова расстояния - можно найти здесь:
Эрих Шуберт и Майкл Герц.
Численно устойчивое параллельное вычисление (ко) дисперсии.
В: Материалы 30-й Международной конференции по управлению научными и статистическими базами данных (SSDBM), Больцано-Бозен, Италия. 2018, 10: 1–10: 12
На самом деле координата y может быть удалена из этого тестового примера, тогда правильное расстояние тривиально становится 1. Я сделал запрос на перенос, который запускает эту числовую проблему:
XA = np.array([[10000]], np.float32)
XB = np.array([[10001]], np.float32)
assert_equal(euclidean_distances(XA, XB)[0,0], 1)
Я не думаю, что моя статья, упомянутая выше, дает решение этой проблемы - просто вычислите евклидово расстояние как sqrt (sum (power ())), и оно будет однопроходным и имеет разумную точность. Потеря заключается в том, что уже используются квадраты, т.е. сама точка (x, x) уже теряет точность.
@amueller, поскольку проблема может быть более серьезной, чем ожидалось, я предлагаю повторно добавить ярлык блокировщика ...
Спасибо за этот очень простой пример.
Причина, по которой он реализован таким образом, в том, что он намного быстрее. Смотри ниже:
x = np.random.random_sample((1000, 1000))
%timeit euclidean_distances(x,x)
20.6 ms ± 452 µs per loop (mean ± std. dev. of 7 runs, 10 loops each)
%timeit cdist(x,x)
695 ms ± 4.06 ms per loop (mean ± std. dev. of 7 runs, 1 loop each)
Хотя количество операций одинакового порядка в обоих методах (в 1,5 раза больше во втором), ускорение происходит за счет возможности использовать хорошо оптимизированные библиотеки BLAS для умножения матриц.
Это было бы огромным замедлением для нескольких оценщиков в scikit-learn.
Да, но только 3-4 цифры точности с FP32 и 7-8 цифр с FP64 делают причину существенных неточностей, не так ли? В частности, поскольку такие ошибки имеют тенденцию усиливаться ...
Я не говорю, что сейчас все в порядке. :)
Я говорю, что нам нужно найти промежуточное решение.
Существует PR (# 11271), в котором предлагается использовать float64 для выполнения вычислений. In не решает проблему для float64, но дает лучшую точность для float32.
У вас есть пример, когда использование оценщика, использующего euclidean_distances, дает неверные результаты из-за потери точности?
Я, конечно, все еще думаю, что это большое дело и должно быть блокировщиком для 0.21. Это была проблема, появившаяся для 32-битной версии в версии 0.19, и оставлять ее - не лучший вариант. Мне жаль, что мы не решили это раньше в версии 0.20, и я был бы в порядке, или даже хотел бы, чтобы на промежуточном этапе # 11271 был объединен. Единственная проблема в этом PR, которую я знаю об оптимизации эффективности памяти, - это глубокая кроличья нора.
У нас уже давно есть эта «быстрая» версия, но всегда в формате float64. Я знаю, @ kno10 , что у него проблемы с точностью. Есть ли у вас хорошая и быстрая эвристика, чтобы мы могли ее решить, когда это может быть проблемой, и использовать более медленное, но более надежное решение?
Да, но только 3-4 цифры точности с FP32 и 7-8 цифр с FP64 действительно вызывают существенную неточность, не так ли?
Спасибо, что проиллюстрировали эту проблему очень простым примером!
Однако я не думаю, что эта проблема настолько распространена, как вы предполагаете - она в основном затрагивает образцы, взаимное расстояние которых мало по сравнению с их нормами.
На рисунке ниже это показано для 2e6 пар случайных выборок, где каждая 1D выборка находится в интервале [-100, 100]. Относительная ошибка между реализациями scikit-learn и scipy отображается как функция расстояния между выборками, нормализованная их нормами L2, т. Е.
d_norm(A, B) = d(A, B) / sqrt(‖A‖₂*‖B‖₂)
(не уверен, что это правильная параметризация, но просто для получения результатов, несколько инвариантных к масштабу данных),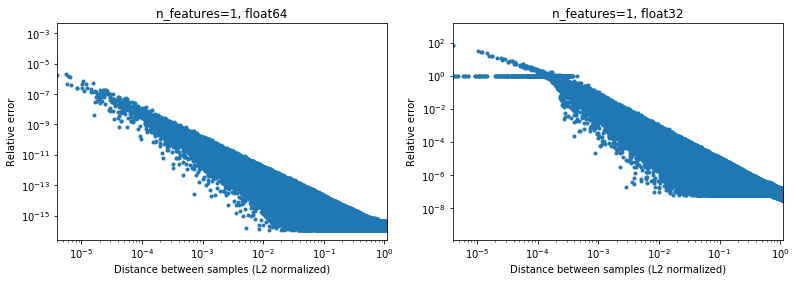
Например,
- если взять
[10000]и[10001]нормализованное расстояние L2 будет 1e-4, а относительная ошибка при вычислении расстояния будет 1e-8 в 64-битном режиме и> 1 в 32-битном (или 1e -8 и> 1 по модулю соответственно). В 32-битном случае это действительно ужасно. - с другой стороны, для
[1]и[10001]относительная ошибка будет ~ 1e-7 в 32-битном формате, или максимально возможной точности.
Вопрос в том, как часто случай 1. будет происходить на практике в приложениях ML.
Интересно, что если мы перейдем к 2D, опять же с равномерным случайным распределением, будет трудно найти точки, которые очень близки,
Конечно, в действительности наши данные не будут выбираться равномерно, но для любого распределения из-за проклятия размерности расстояние между любыми двумя точками будет медленно сходиться к очень похожим значениям (отличным от 0) по мере увеличения размерности. Хотя это общая проблема машинного обучения, здесь она может несколько смягчить проблему точности даже для относительно низкой размерности. Ниже результатов для n_features=5 ,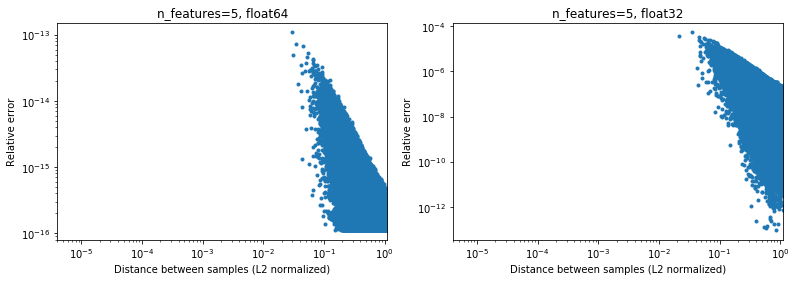 .
.
Таким образом, для центрированных данных, по крайней мере, в 64-битной версии, это может быть не такой уж большой проблемой на практике (при условии, что имеется более двух функций). 50-кратное ускорение вычислений (как показано выше) может того стоить (в 64-битной версии). Конечно, всегда можно добавить 1e6 к некоторым данным, нормализованным в [-1, 1], и сказать, что результаты неточны, но я бы сказал, что то же самое относится к ряду числовых алгоритмов и работе с данными, выраженными в 6-м значащая цифра просто ищет неприятностей.
(Код для приведенных выше цифр можно найти здесь ).
Любой быстрый подход с использованием версии dot (x, x) + dot (y, y) -2 dot (x, y), вероятно, будет иметь ту же проблему для всего, что я могу сказать, но вам лучше спросить какого-нибудь настоящего эксперта по числам за это. точность * входных данных (и я бы предположил, что если пользователь предоставляет данные float32, тогда им потребуется точность float32, а с float64 им потребуется точность float64). Возможно, вы сможете сделать это с помощью некоторых уловок (подумайте о суммировании Кахана), но, скорее всего, это будет стоить вам гораздо больше, чем вы изначально получили.
Я не могу сказать, сколько накладных расходов вы получите от преобразования float32 в float64 на лету при использовании этого подхода. По крайней мере, для float32, насколько я понимаю, выполнение всех вычислений и сохранение скалярных произведений как float64 должно быть нормальным.
ИМХО, прирост производительности (который не является экспоненциальным, а является постоянным коэффициентом) не стоит потери точности (которая может вас неожиданно укусить), и правильный способ - не использовать этот проблемный трюк. Однако вполне возможно дальнейшая оптимизация кода, выполняющего «традиционные» вычисления, например, для использования AVX. Потому что sum ((xy) * 2) практически сложно реализовать в AVX.Как минимум, я бы предложил переименовать метод в approximate_euclidean_distances из-за иногда низкой точности (которая становится хуже, чем ближе два значения, что * может быть хорошо изначально, а затем начинает иметь значение при приближении к некоторому оптимуму) , чтобы пользователи знали об этой проблеме.
@rth спасибо за иллюстрации. Но что, если вы пытаетесь оптимизировать, например, x до некоторого оптимума. Скорее всего, оптимум не будет равен нулю (если бы это всегда был ваш центр обработки данных, жизнь была бы прекрасна), и в конечном итоге дельты, которые вы вычисляете для градиентов и т. Д., Могут иметь очень небольшие различия.
Точно так же при кластеризации не все кластеры будут иметь свои центры, близкие к нулю, но, в частности, для многих кластеров вполне возможен x ≈ center с несколькими цифрами.
Однако в целом я согласен, что эту проблему нужно исправить. В любом случае нам нужно как можно скорее задокументировать проблемы точности текущей реализации.
В целом, хотя я не думаю, что это обсуждение должно происходить в scikit-learn. Евклидово расстояние используется в различных областях научных вычислений, и scipy список рассылки IMO или вопросы - лучшее место для его обсуждения: это сообщество также имеет больше опыта в таких вопросах числовой точности. Фактически, у нас есть быстрый, но несколько приблизительный алгоритм. Возможно, нам придется внедрить некоторые обходные пути исправлений в краткосрочной перспективе, но в долгосрочной перспективе было бы хорошо знать, что это будет сделано там.
Для 32-разрядной версии https://github.com/scikit-learn/scikit-learn/pull/11271 действительно может быть решением, я просто не очень заинтересован в нескольких уровнях разбиения по всей библиотеке, поскольку это увеличивает сложность кода , и хотите убедиться, что нет лучшего способа обойти это.
Спасибо за ответ @ kno10! (Мои вышеупомянутые комментарии пока не учитывают это) Я отвечу чуть позже.
Да, сближение с какой-то точкой за пределами источника может быть проблемой.
ИМХО, прирост производительности (который не является экспоненциальным, а является постоянным коэффициентом) не стоит потери точности (которая может вас неожиданно укусить), и правильный способ - не использовать этот проблемный трюк.
Что ж, более чем 10-кратное замедление их вычислений в 64-битной версии будет иметь очень реальный эффект на пользователей.
Однако вполне возможно дальнейшая оптимизация кода, выполняющего «традиционные» вычисления, например, для использования AVX. Потому что sum ((xy) ** 2) практически сложно реализовать в AVX.
Пробовал быструю наивную реализацию с numba (которая должна использовать SSE),
@numba.jit(nopython=True, fastmath=True)
def pairwise_distance_naive(A, B):
n_samples_a, n_features_a = A.shape
n_samples_b, n_features_b = B.shape
assert n_features_a == n_features_b
distance = np.empty((n_samples_a, n_samples_b), dtype=A.dtype)
for i in range(n_samples_a):
for j in range(n_samples_b):
psum = 0.0
for k in range(n_features_a):
psum += (A[i, k] - B[j, k])**2
distance[i, j] = math.sqrt(psum)
return distance
получая скорость, аналогичную scipy cdist (но я не эксперт по numba), а также не уверен в эффекте fastmath .
используя версию точка (x, x) + точка (y, y) -2 * точка (x, y)
Просто для справки в будущем, то, что мы сейчас делаем, примерно следующее (потому что есть измерение, которое не отображается в приведенных выше обозначениях),
def quadratic_pairwise_distance(A, B):
A2 = np.einsum('ij,ij->i', A, A)
B2 = np.einsum('ij,ij->i', B, B)
return np.sqrt(A2[:, None] + B2[None, :] - 2*np.dot(A, B.T))
где и einsum и dot теперь используют BLAS. Интересно, если помимо использования BLAS, он также выполняет то же количество математических операций, что и первая версия выше.
Интересно, если помимо использования BLAS, он также выполняет то же количество математических операций, что и первая версия выше.
Нет. ((X - y) * 2.sum ()) выполняет* n_samples_x * n_samples_y * n_features * (1 вычитание + 1 сложение + 1 умножение)
тогда как xx + yy -2x.y выполняет
n_samples_x * n_samples_y * n_features * (1 сложение + 1 умножение) .
Количество операций между двумя версиями составляет 2/3.
Следуя приведенному выше обсуждению,
- Сделал PR, чтобы можно было точно вычислить евклидовы расстояния https://github.com/scikit-learn/scikit-learn/pull/12136
- Некоторый WIP, чтобы увидеть, можем ли мы обнаружить и смягчить проблемные точки в https://github.com/scikit-learn/scikit-learn/pull/12142
Для 32-битной версии нам все еще нужно объединить https://github.com/scikit-learn/scikit-learn/pull/11271 в той или иной форме, хотя ИМО, вышеупомянутые PR несколько ортогональны ему.
К вашему сведению: при исправлении некоторых проблем в OPTICS и обновлении теста для использования эталонных результатов из ELKI они терпят неудачу с metric="euclidean" но успешно с metric="minkowski" . Численные различия достаточно велики, чтобы вызвать другой порядок обработки (простого уменьшения порога недостаточно).
Я действительно не догнал это, но я удивлен, что решения нет. Это кажется очень распространенным вычислением, и похоже, что мы изобретаем колесо. Кто-нибудь пробовал обратиться к более широкому научному компьютерному сообществу?
Пока нет, но я согласен, что мы должны. Единственное, что я нашел об этом в scipy, это https://github.com/scipy/scipy/pull/2815 и связанные проблемы.
Мне кажется, у @jeremiedbb есть идея?
К сожалению, пока не удовлетворительный :(
Мы хотели бы полагаться на высокооптимизированную библиотеку для такого рода вычислений, как мы делаем для линейной алгебры с библиотеками BLAS, такими как OpenBLAS или MKL. Но евклидово расстояние не является его частью. Трюк с точками - это попытка сделать это, опираясь на подпрограмму умножения матрицы на матрицу уровня 3 BLAS. Но это неточно, и нет возможности сделать это более точным с помощью того же метода. Мы должны снизить наши ожидания либо с точки зрения скорости, либо с точки зрения точности.
Я думаю, что в некоторых ситуациях полная точность не является обязательной, и можно использовать быстрый метод. Это когда расстояния используются для «поиска ближайших» задач. Проблемы точности в быстром методе возникают, когда расстояние между точками мало по сравнению с их нормой (в соотношении ~ <1e-4 для float 32 и ~ <1e-8 для float64). Во-первых, чтобы эта ситуация произошла, набор данных должен быть достаточно плотным. Тогда, чтобы иметь место ошибка упорядочивания, вам нужно, чтобы две ближайшие точки находились примерно на одинаковом расстоянии. Более того, в этом случае, с точки зрения машинного обучения, оба варианта приведут к почти одинаково хорошим совпадениям.
В приведенной выше ситуации мы можем кое-что сделать, чтобы снизить частоту неправильного упорядочивания (до 0?). В ситуации попарного расстояния argmin. Мы можем переместить перестановку в точки, которые не являются ближайшими. По сути, используя тот факт, что одна из норм не является обязательной для поиска argmin, см. Комментарий . У него есть 2 преимущества. Это более надежно (до сих пор я еще не нашел неправильного порядка) и еще быстрее, потому что позволяет избежать некоторых вычислений.
Один недостаток, все еще в той же ситуации, если в конце мы хотим фактические расстояния до ближайших точек, расстояния, вычисленные с помощью вышеуказанного метода, не могут быть использованы. Они вычислены лишь частично и в любом случае неточны. Нам нужно заново вычислить расстояния от каждой точки до ближайшей точки. Но это быстро, потому что для каждой точки нужно вычислить только одно расстояние.
Интересно, что описанное выше охватывает все варианты использования euclidean_distances в sklearn. Но я предлагаю делать это везде, где это возможно. Для этого мы можем добавить новый параметр в euclidean_distances, чтобы вычислить только необходимую часть, чтобы связать ее с argmin. Затем используйте его в pairwise_distances_argmin и в pairwise_distances_argmin_min (повторно вычисляя фактические минимальные расстояния в конце в последнем).
Когда мы не можем этого сделать, вернитесь к медленному, но точному, или добавьте переключатель, как в # 12136.
Мы можем попытаться немного оптимизировать его, чтобы снизить падение производительности, потому что я согласен, что это не кажется оптимальным. У меня есть несколько идей на этот счет.
Еще одна возможность продолжить использование BLAS - это сочетание axpy с nrm2 но это далеко не оптимально. Обе функции являются функциями BLAS уровня 1 и включают в себя копию. Это будет быстрее только в размерности> 100.
В идеале мы хотели бы, чтобы евклидово расстояние было включено в BLAS ...
Наконец, есть еще одно решение - апкастинг. Это сделано в # 11271 для float32. Преимущество состоит в том, что скорость вдвое меньше текущей и сохраняется точность. Однако это не решает проблему для float64. Возможно, мы найдем способ сделать то же самое в cython для float64. Я точно не знаю, как, но используя 2 числа float64 для имитации float128. Я могу попробовать, чтобы увидеть, возможно ли это.
В идеале мы хотели бы, чтобы евклидово расстояние было включено в BLAS ...
Это то, что библиотеки рассмотрят? Если OpenBLAS сделает это, мы уже были бы в довольно хорошей ситуации ...
Кроме того, каковы точные различия между тем, что мы делаем, и BLAS? Определение возможностей процессора и решение, какую реализацию использовать, или что-то в этом роде? Или просто скомпилировали версии для более разнообразных архитектур?
Или просто потратить больше времени / энергии на написание эффективных реализаций?
Это интересно: альтернативная реализация метода fast unstable, но утверждающая, что он намного быстрее, чем sklearn:
https://github.com/droyed/eucl_dist
(вообще не решает эту проблему, лол)
Это обсуждение кажется связанным https://github.com/scipy/scipy/issues/5657
Вот что делает Юлия: https://github.com/JuliaStats/Distances.jl/blob/master/README.md#precision -for-euclidean-and-sqeuclidean
Это позволяет установить порог точности для принудительного пересчета.
Отвечая на свой вопрос: OpenBLAS имеет то, что выглядит как рукописная сборка для каждого процессора (не архитектура!) И эвтистика для выбора ядер для разных размеров проблем. Так что я не думаю, что проблема заключается в том, чтобы ввести его в openblas, а в том, чтобы найти кого-то, кто написал / оптимизировал все эти ядра ...
Спасибо за дополнительные мысли!
В частичном ответе
Мы хотели бы полагаться на высокооптимизированную библиотеку для такого рода вычислений, как мы делаем для линейной алгебры с библиотеками BLAS, такими как OpenBLAS или MKL.
Да, я также надеялся, что мы сможем сделать больше в BLAS. В прошлый раз, когда я ничего не искал в стандартном BLAS API, он выглядит достаточно близко (но тогда я не эксперт в этом). BLIS может предложить большую гибкость, но поскольку мы не используем его по умолчанию, он имеет несколько ограниченное использование (хотя numpy когда-нибудь может https://github.com/numpy/numpy/issues/7372)
Вот что делает julia: позволяет установить порог точности для принудительного пересчета.
Приятно знать!
Следует ли нам открыть отдельный вопрос для более быстрого приблизительного вычисления, указанного выше? Кажется интересным
Их ускорение на процессоре x2-x4 может быть связано с https://github.com/scikit-learn/scikit-learn/pull/10212 .
Я бы предпочел открыть вопрос о scipy после того, как мы изучим этот вопрос достаточно, чтобы найти там разумное решение (а затем, возможно, выполнить его бэкпорт), поскольку я чувствую, что евклидова дистанция - это что-то достаточно базовое, что должно быть интересно многим людям за пределами ML. (и в то же время наличие мнения людей, например, по вопросам точности, было бы полезным).
Это до 60x, правда?
Это интересно: альтернативная реализация метода fast unstable, но утверждающая, что он намного быстрее, чем sklearn
гул, не уверен в этом. Они тестируют %timeit pairwise_distances(a,b, 'sqeuclidean') , в котором используется scipy. Они должны сделать %timeit pairwise_distances(a,b, 'euclidean', metric_params={'squared': True}) и их ускорение будет не таким хорошим :)
Как было показано ранее в обсуждении, sklearn может быть в 35 раз быстрее, чем scipy.
Да, их тесты только на ~ 30% лучше с metric="euclidean" (вместо squeclidean ),
In [1]: from eucl_dist.cpu_dist import dist
... import numpy as np
In [4]: rng = np.random.RandomState(1)
... a = rng.rand(1000, 300)
...b = rng.rand(1000, 300)
In [7]: from sklearn.metrics.pairwise import pairwise_distances
In [8]: %timeit pairwise_distances(a, b, 'sqeuclidean')
214 ms ± 2.06 ms per loop (mean ± std. dev. of 7 runs, 1 loop each)
In [9]: %timeit pairwise_distances(a, b)
27.4 ms ± 2.48 ms per loop (mean ± std. dev. of 7 runs, 10 loops each)
In [10]: from eucl_dist.cpu_dist import dist
In [11]: %timeit dist(a, b, matmul='gemm', method='ext', precision='float32')
20.8 ms ± 330 µs per loop (mean ± std. dev. of 7 runs, 10 loops each)
In [12]: %timeit dist(a, b, matmul='gemm', method='ext', precision='float64')
20.4 ms ± 222 µs per loop (mean ± std. dev. of 7 runs, 10 loops each)
Это то, что библиотеки рассмотрят? Если OpenBLAS сделает это, мы уже были бы в довольно хорошей ситуации ...
Звучит непросто. BLAS - это набор спецификаций для процедур линейной алгебры, и есть несколько его реализаций. Я не знаю, насколько они открыты для добавления новых функций, которых нет в исходных спецификациях. Для этого, возможно, блис будет более открытым, но, как было сказано ранее, на данный момент это не по умолчанию.
Открыт https://github.com/scikit-learn/scikit-learn/issues/12600 при обработке sqeuclidean vs euclidean в pairwise_distances .
Мне нужна некоторая ясность в том, что мы хотим для этого. Хотим ли мы, чтобы pairwise_distances был близок - в смысле all_close - как для "евклидова", так и для "квевклидова"?
Это немного сложно. Поскольку x близок к y, это не означает, что x² близко к y². При возведении в квадрат теряется точность.
Обходной путь julia, указанный выше, очень интересен и довольно прост в реализации. Однако я подозреваю, что он работает не так, как ожидалось для sqeuclidean. Я подозреваю, что вам нужно установить порог ниже, чтобы получить желаемую точность.
Проблема с установкой очень низкого порога заключается в том, что это вызывает множество повторных вычислений и огромное падение производительности. Однако это смягчается размером набора данных. Тот же порог вызовет намного меньше повторных вычислений в высоком измерении (расстояния больше).
Возможно, мы сможем иметь 2 реализации и переключаться в зависимости от размера набора данных. Медленный, но безопасный для низкоразмерных (в любом случае большой разницы между scipy и sklearn) и быстрый + пороговый для высокомерных.
Для этого потребуются некоторые тесты, чтобы определить, когда переключаться и установить порог, но это может быть проблеском надежды :)
Вот несколько тестов для сравнения скорости между scipy и sklearn. Тесты сравнивают sklearn.metrics.pairwise.euclidean_distances(X,X) с scipy.spatial.distance.cdist(X,X) для X всех размеров. Количество образцов изменяется от 2⁴ (16) до 2¹³ (8192), а количество элементов изменяется от 2⁰ (1) до 2¹³ (8192).
Значение в каждой ячейке - это ускорение sklearn по сравнению с scipy, т.е. менее 1 sklearn медленнее, а более 1 sklearn - быстрее.
Первый тест использует реализацию BLAS в MKL и одно ядро.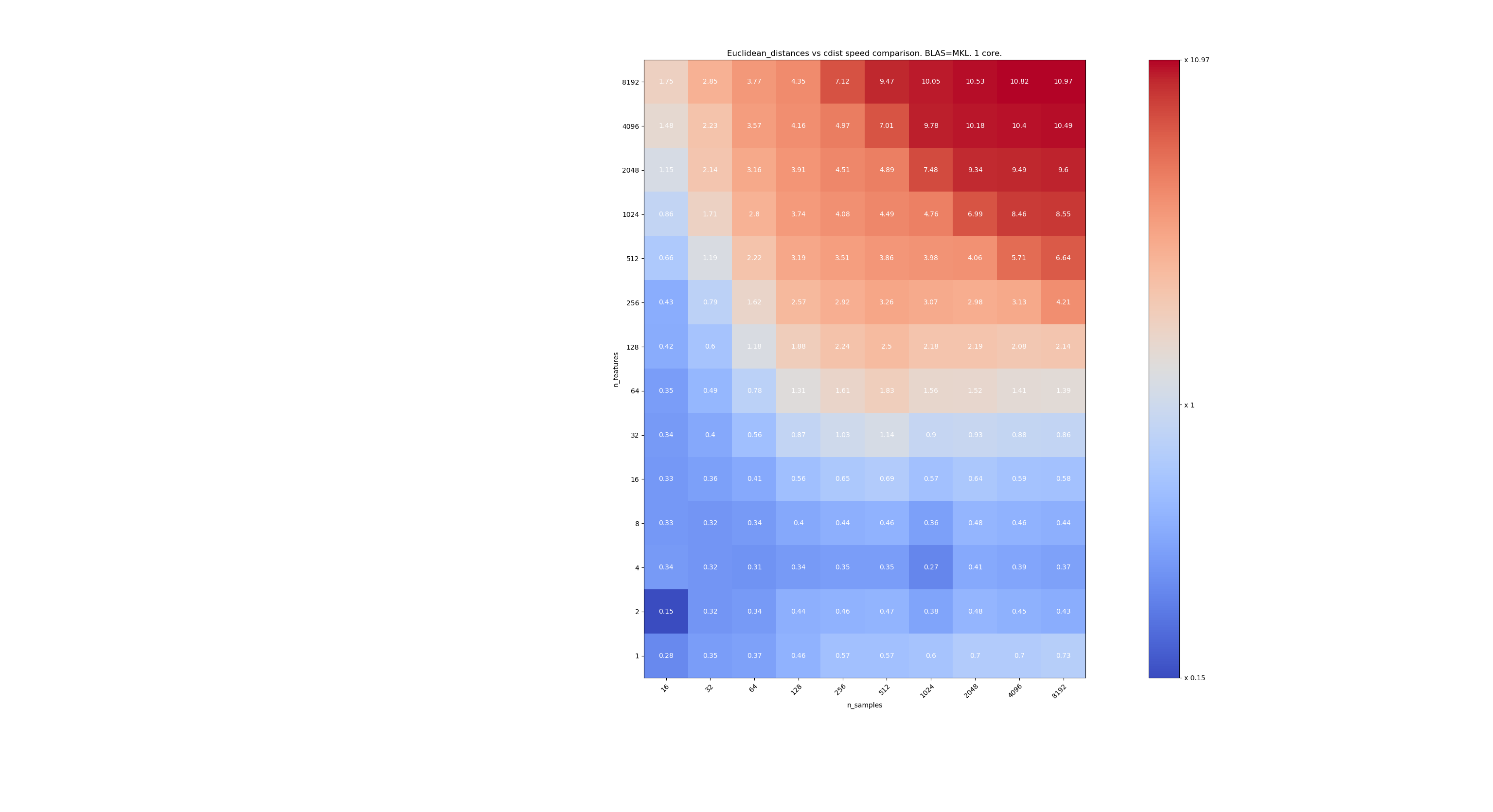
Второй - использует реализацию BLAS OpenBLAS и одно ядро. Просто чтобы убедиться, что MKL и OpenBLAS ведут себя одинаково.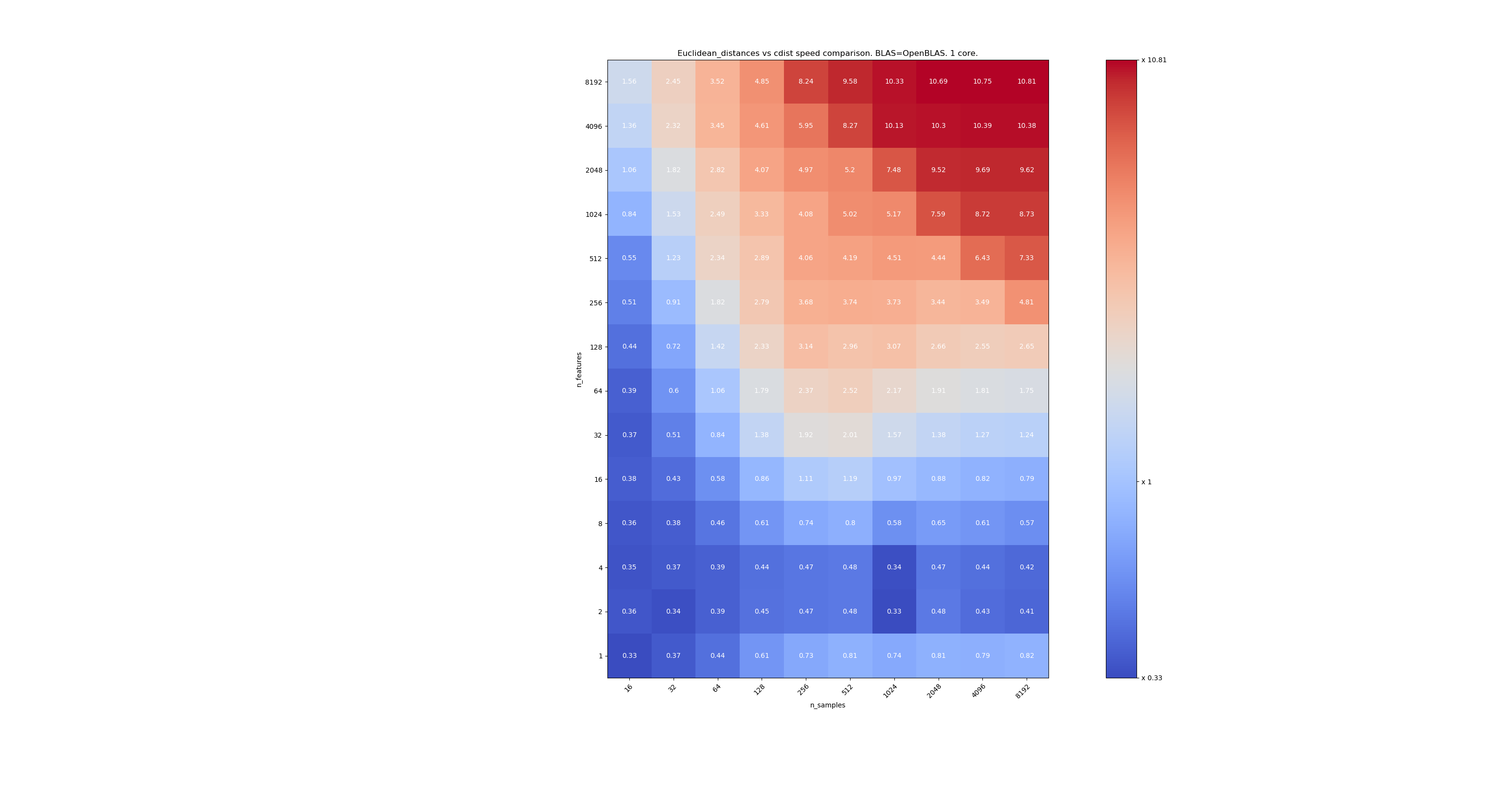
Третий использует реализацию BLAS и 4 ядра MKL. Дело в том, что euclidean_distances распараллеливается с помощью функции BLAS LEVEL 3, но cdist использует только функцию BLAS LEVEL 1. Что интересно, границы это почти не меняет.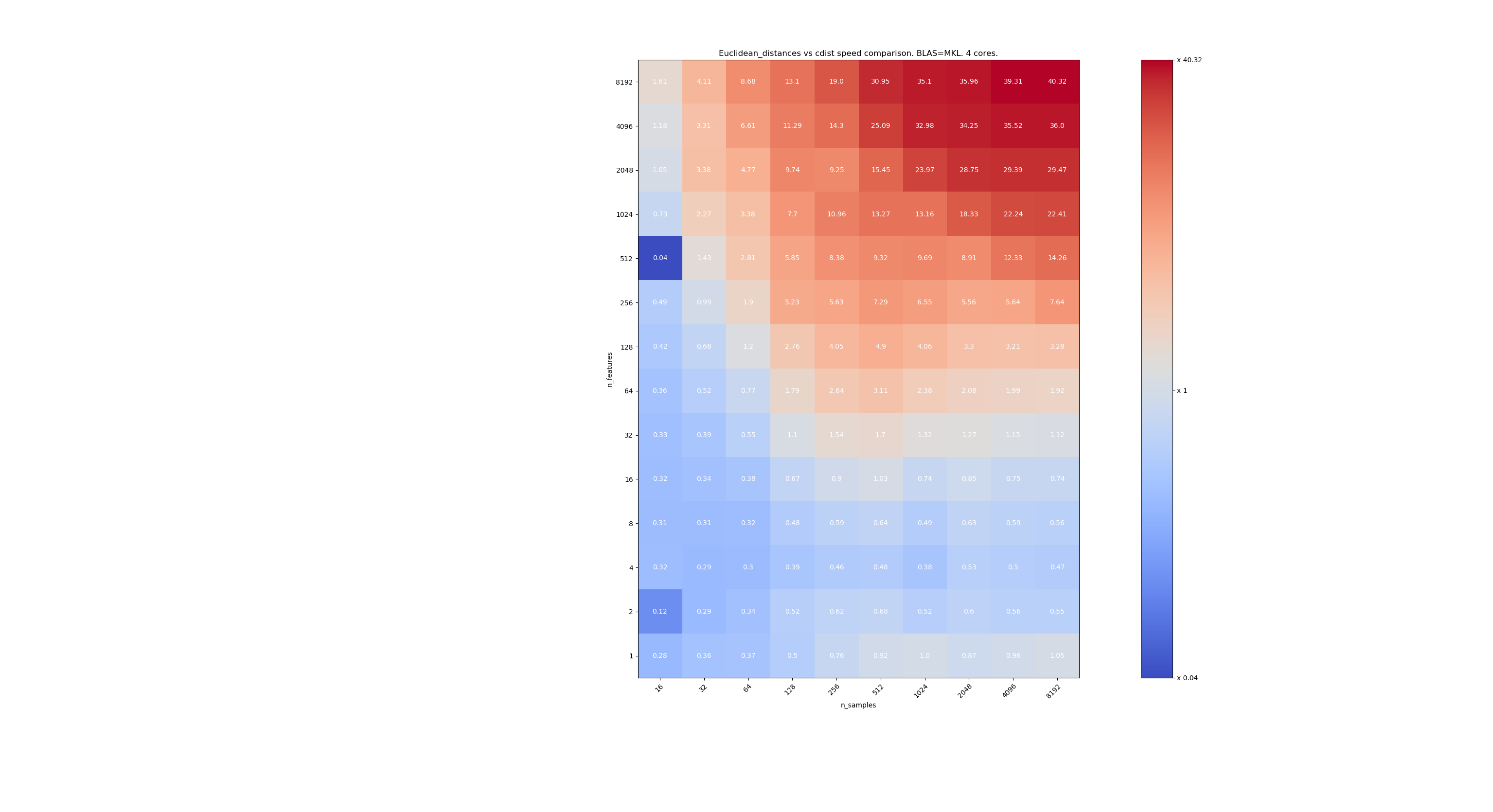
Когда n_samples не слишком низкое (> 100), кажется, что граница составляет около 32 функций. Мы могли бы решить использовать cdist, когда n_features <32, и euclidean_distances, когда n_features> 32. Это быстрее и нет проблем с точностью. Это также имеет то преимущество, что, когда n_features мало, порог julia приводит к большому количеству повторных вычислений. Использование cdist позволяет избежать этого.
Когда n_features> 32, мы можем сохранить реализацию euclidean_distances , обновленную с помощью порога julia. Добавление порога не должно слишком сильно замедлять euclidean_distances потому что количество функций достаточно велико, поэтому требуется лишь несколько повторных вычислений.
@jeremiedbb отлично, спасибо за анализ. Вывод звучит для меня как отличный путь вперед.
О, я полагаю, это все для float64, верно? Что нам делать с float32? всегда приподнятый? upcast для> 32 функций?
Я не читал внимательно комментарии (скоро), просто к сведению, что у float64 есть ограничения, см. # 12128
@ qinhanmin2014 да, точность float64 имеет ограничения, но она достаточно точна для получения надежных результатов fp32, насколько я могу судить. Вопрос в том, при каких параметрах upcast до fp64 на самом деле дешевле, чем использование cdist от scipy.
Как видно из приведенных выше тестов, даже многоядерный BLAS обычно не быстрее. Это, по-видимому, в основном справедливо для многомерных данных (более 64 измерений; до этого выгода обычно не стоит усилий ИМХО) - и поскольку евклидовы расстояния не так надежны в плотных многомерных данных, этот вариант использования ИМХО не имеет наивысшего значения . У многих пользователей будет меньше 10 измерений. В этих случаях cdist обычно работает быстрее?
О, я полагаю, это все для float64, верно?
На самом деле это как для float32, так и для float64 (то есть очень похоже). Я предлагаю всегда использовать cdist, когда n_features <32.
Вопрос в том, при каких параметрах upcast до fp64 на самом деле дешевле, чем использование cdist от scipy.
Апкастинг замедлится примерно в 2 раза, поэтому я предполагаю, что n_features = 64.
У многих пользователей будет меньше 10 измерений.
Но не всем, поэтому нам все еще нужно найти решение для данных большой размерности.
Очень хороший анализ @jeremiedbb !
Для низкоразмерных данных определенно имеет смысл использовать cdist.
Кроме того, cdist FYI scipy обновляет float32 до float64 https://github.com/scipy/scipy/issues/8771#issuecomment -384015674, я не уверен, связано ли это с проблемами точности или чем-то еще.
В целом, я думаю, что имеет смысл добавить параметр «алгоритм» к euclidean_distance как предлагается в https://github.com/scikit-learn/scikit-learn/pull/12601#pullrequestreview -176076355, возможно со значением по умолчанию «Нет», чтобы его также можно было установить с помощью глобального параметра, как в https://github.com/scikit-learn/scikit-learn/pull/12136.
В Eigen3 также есть интересный подход для вычисления стабильных норм: https://eigen.tuxfamily.org/dox/StableNorm_8h_source.html (который я еще не изучил)
Хорошее объяснение, улучшилось мое понимание
Мы не добились каких-либо успехов в этом спринте, и, вероятно, нам стоит ... а @rth сегодня нет.
Я могу присоединиться удаленно, если вы установите время. Может, в начале дня?
Подводя итог ситуации,
По вопросам точности вычислений евклидова расстояния
- в случае низкой размерности, как @jeremiedbb показал выше, мы, вероятно, должны использовать cdist
- в случае большой размерности и float32 мы могли выбирать между,
- разбиение на части, вычисление расстояния в 64 битах и конкатенация
- возврат к cdist в случаях, когда точность является проблемой (как открытый вопрос - обращение, например, к scipy, может быть полезно https://github.com/scikit-learn/scikit-learn/issues/9354#issuecomment-438522881 )
Затем есть все вопросы несоответствия между евклидовым, скевклидовым, минковским и т. Д.
Что касается точности, мы с @jeremiedbb , @amueller быстро поболтали , в основном просто доили Джереми за его опыт. Он считает, что нам не нужно так сильно беспокоиться о проблемах нестабильности в контексте машинного обучения в больших измерениях в float64. Джереми также намекал, что трудно найти эффективный тест на получение хороших результатов (см. # 12142).
Так что я думаю , что мы довольны @rth «ы предшествующий комментарий с float32 для приведения к базовому типу. Поскольку cdist также обновляется до float64, мы могли бы переопределить cdist, чтобы он принимал float32 (но с аккумуляторами float64?), Или можем использовать разбиение на части, если мы хотим меньше копировать в float32 с низким разрешением.
@Celelibi хочет изменить PR в # 11271, или кто-то другой (один из нас?) Должен сделать полный запрос на вытягивание?
И как только это будет исправлено, я думаю, мы должны заставить sqeuclidean и minkowski (p in {0,1}) использовать наши реализации. Мы не обсуждали расхождения с NearestNeighbours. Еще один спринт :)
После быстрого обсуждения на спринте мы пришли к следующему:
в высокомерном случае (> 32 или> 64 выберите лучший): преобразование по фрагментам в float64, когда это float32, и сохранение «быстрого» метода. Для такого рода данных числовые проблемы с float64 почти несущественны (для этого я дам тесты)
в низкоразмерном случае: реализовать безопасное вычисление (вместо использования scipy cdist из-за восходящего преобразования) в sklearn.
(Заманчиво также добавить апкастинг float32 в 0.20.3)
Вот несколько тестов для сравнения скорости между scipy и sklearn.
[... вырезать ...]
Это очень интересно. На самом деле я не ожидал такого результата. Я перепроверил ваш тест и нашел очень похожий результат. За исключением того, что я бы выступал за более низкую границу принятия решения. Мой тест предлагал 8 функций.

Цена ошибки несимметрична. cdist лучше только для вычислений продолжительностью менее нескольких секунд, и он становится очень быстро медленным, когда количество функций увеличивается. Так что, если сомневаетесь, лучше используйте реализацию BLAS.
Изменить: этот тест был для float64, но я также обнаружил, что преобразование матриц float32 в float64 едва ли добавляет несколько процентов от общего времени и не меняет вывод.
Я заметил, что порог зависит от машины, на которой вы запускаете тесты. Я подозреваю, что это может быть связано с инструкциями AVX. Я понял, что опубликованные мной тесты были запущены на машине, на которой не было инструкций AVX2, только AVX. И на машине с AVX2 я получил результаты, аналогичные вашим.
Но вопрос не только в производительности, но и в точности, и более вероятно возникновение проблем с точностью, когда размер небольшой. Может быть, 16 - хороший компромисс. Что вы думаете ?
Что касается этого обсуждения, я бы сказал, что нам нужно оценить точность, чтобы принять обоснованное решение.
Однако, что касается вашего PR, точность больше не должна быть проблемой. Но ценой более дорогих вычислений. Поэтому порог, вероятно, следует определять путем сравнения вашего PR.
Точность сравнительного анализа не так проста. Потому что сложные случаи не будут распределены равномерно.
И это может быть проблематично, если это произойдет незамеченным в угловом случае. Обычно вам нужна гарантированная числовая точность в пределах доступного ЦП.
Но, как упоминалось в другом месте, одной функции с 10000000.01 и 10000000.00 должно быть достаточно, чтобы вызвать числовую нестабильность с fp64 при использовании известного проблемного уравнения 10000 и 10001 с fp32. С 1024 функциями попробуйте
>>> import sklearn.metrics.pairwise as sk, scipy.spatial.distance as sp
>>> X = [[10000.01] * 1024, [10000.00] * 1024]
>>> print(sk.euclidean_distances(X,X), "\n", sp.cdist(X,X))
[[ 0. 0.31895195]
[ 0.31895195 0. ]]
[[ 0. 0.32]
[ 0.32 0. ]]
(здесь использовалось 0,19.1) Правильное расстояние - 0,32.
Как видите, числовые нестабильности имеют тенденцию ухудшаться с увеличением количества функций (если только ваши данные не скудны). Здесь результат имеет менее двух знаков точности с FP64.
13410 не исправляет этот конкретный случай. т.е. float64 + высокая размерность.
Однако он исправляет это для float32.
Но мы решили, что для float64 + high dim мы оставим его как есть, потому что проблемы с точностью очень маловероятны и не относятся к случаям использования машинного обучения.
В вашем примере X [0] и X [1] имеют нормы, равные 320000,32 и 320000, а их расстояние составляет 0,32, то есть в 1e-6 раз больше их нормы. В машинном обучении не все 16 значащих цифр (в float64) имеют значение.
Но мы решили, что для float64 + high dim мы оставим его как есть, потому что проблемы с точностью очень маловероятны и не относятся к случаям использования машинного обучения.
Я был бы более умеренным в этом вопросе. Уменьшение размерности - обычный первый шаг в ML. Для этого можно использовать MDS, и в нем активно используется матрица евклидовых расстояний.
Если кто-то хочет взглянуть на повышение точности случая float64, есть способ использовать два числа с плавающей запятой для представления промежуточных результатов. Хотя я думаю, что это начинает выходить за рамки scikit-learn.
ftp://ftp.math.ethz.ch/users/wpp/CELL/qd.pdf
Мне было непонятно. Я не говорю, что многомерные данные неприменимы к машинному обучению. Я говорю, что проблемы с точностью, которые возникают в float64, связаны с точками, расстояние которых на 6 порядков меньше их нормы. Такая точность не имеет значения в реалистичной модели машинного обучения.
В машинном обучении не все 16 значащих цифр (в float64) имеют значение.
Я вовсе не уверен, что это в целом так.
В этом примере мы потеряли 15 из 16 цифр в точности. Я бы согласился, если бы мы использовали половину точности, но у нас нет таких отношений. Потери из-за понижающего преобразования FP64 в FP32 часто могут быть допустимыми из-за точности измерения. И графические процессоры потребительского уровня намного быстрее с FP32, чем, например, с FP64 (хотя в некоторых случаях они теперь позволяют использовать данные FP32 и аккумуляторы FP64), а для вывода нейронных сетей вы даже можете увидеть int8 сейчас. Но это не везде.
Например , в к-средства кластеризации, есть предположение , что кластеры существенно различаются по средствам (и что мы не знаем , средства заранее), и , следовательно , мы имеем потерю точности здесь. Если у вас много кластеров, некоторые из их норм могут быть большими по сравнению с их разделением.
Кроме того, после первых начальных итераций часто небольшие различия в расстоянии заставляют одну точку переключаться на другой кластер. Потеря точности здесь может повлиять на результаты и вызвать нестабильность.
Теперь рассмотрим k-средние на фрагментах временных рядов со многими переменными.
При увеличении размеров данных мы должны предполагать, что расстояния до ближайшего соседа становятся меньше, и, если ваши нормы не равны 0, они в конечном итоге будут меньше, чем векторные нормы, и вызовут проблемы. Таким образом, это, вероятно, станет более серьезным с увеличением размеров набора данных. Проклятие размерности говорит, что самые большие и самые маленькие расстояния становятся все более и более похожими; поэтому для вычисления правильного ранжирования ближайших соседей нам может потребоваться хорошая точность в данных большой размерности. В наборе данных 20news наименьшее ненулевое расстояние составляет около 0,02 (все нормы равны 1). Но это всего 10к экземпляров и довольно разнообразное содержание. Теперь предположим, что вместо этого набор данных был посвящен обнаружению почти дубликатов ...
Я бы не был уверен, что это «маловероятно» произойдет в ML ... конечно, это не коснется всех.
Когда я говорю: «В машинном обучении 16 значащих цифр (в float64) не имеют значения», я не говорю о вычисленном расстоянии, я говорю о данных X.
В машинном обучении ваши данные берутся из меры, и нет меры с точностью до 9-й цифры (кроме очень немногих в физике элементарных частиц).
Итак, в вашем примере 10000000.01 и 10000000.00 , как бы вы придавали какое-то значение расстоянию 0,01, когда ваша неопределенность в значениях X намного больше?
Для KMeans, во-первых, есть способ преодолеть значительную часть потерь точности. Когда вы ищете ближайший центр наблюдения x, вам не нужно добавлять норму x к вычислению расстояния, что в большинстве случаев позволяет избежать катастрофической отмены.
Затем k означает кластеры на основе евклидовых расстояний. Но вы не знаете, именно так ли собираются ваши данные. На самом деле вероятность того, что ваши данные сгруппированы таким образом, равна нулю. Kmeans дает оценку того, как ваши данные могут быть сгруппированы, и точки, которые находятся на границе двух кластеров, определенно не могут считаться принадлежащими с уверенностью одному или другому. Как вы интерпретируете точку на одинаковом расстоянии в 2 кластера? У меня либо 2 кластера должны быть только одним кластером, либо KMeans не лучший алгоритм для кластеризации моих данных (или даже kmeans дает мне некоторое представление о том, как мои данные кластеризованы, но я знаю, что границы кластеров не имеют отношения).
Использование только «| b | ^ 2-2ab» не приводит к катастрофической отмене - но такая же потеря точности в цифрах, которые имеют значение. Результаты такие же, как если бы вы впоследствии добавили норму a к каждому расстоянию; если расстояния намного меньше нормы a, тогда вы получите потерю точности, которой можно избежать, выполняя вычисления традиционным способом без взлома BLAS.
Таким образом, вы НЕ МОЖЕТЕ решить числовую проблему!
K-means - это проблема оптимизации. Таким образом, такие хаки могут означать, что sklearn находит только худшие решения, чем другие инструменты. И, как указывалось ранее, это также может вызвать нестабильность. В худшем случае это может привести к тому, что sklearn kmeans будет перебирать одни и те же состояния до тех пор, пока не будет max_iter без каких-либо улучшений (при условии, что tol = 0, если вы хотите найти локальный оптимум), что согласно теории невозможно.
Пока k-means не сойдется, вы не сможете много сказать о точках с «одинаковым» расстоянием до двух кластеров. В следующей итерации средства могут измениться, и разница может стать намного больше и иметь значение!
Я не большой поклонник k-средних, потому что он не очень хорошо работает с зашумленными данными. Но есть варианты, которые лучше справляются с такими случаями. Но тем не менее, если вы используете его, вам, вероятно, следует попытаться получить полное качество (поэтому я также всегда использую tol=0 ) и не ухудшать его, чем необходимо. Это достаточно дешево, чтобы выполнять правильные вычисления (и, как уже упоминалось, проблемы усугубляются с размером данных - поэтому для небольших данных более медленное время выполнения обычно не имеет значения, для больших наборов данных точность становится более важной).
В зависимости от приложения разница между 10000000.01 и 10000000.00 может иметь значение. И, как я показал ранее, если вы используете несколько функций, проблемы возникают раньше. С fp32 всего 10000 и 10001 с одной функцией и 100 против 101 со 100 функциями, я думаю:
Как уже упоминалось, среднее значение может иметь физическое значение, которое вы не хотите терять. Если у вас есть данные с температурами в Кельвинах, вы не хотите масштабировать их 0: 1 или центрировать их; это испортит вашу шкалу соотношений . Теперь, если вы хотите сравнить, например, временной ряд температуры некоторого стального изделия при его остывании, и выяснить, влияет ли процесс охлаждения на надежность вашего стального изделия. У вас могут быть температуры выше 700 К, а временные ряды могут иметь сотни точек данных, если вы хотите проанализировать процесс охлаждения. Даже при точности ввода всего 5 цифр (0,01 КБ) с длиной временного ряда может возникнуть числовая проблема. Вы можете снова получить только 1-2 цифры в результате. Я не думаю, что вы можете просто исключить, что точность когда-либо имеет значение в машинном обучении, если у вас есть такой катастрофический эффект. Другое дело, если вы всегда можете гарантировать точность, скажем, 10 из 16 цифр. Здесь этого сделать нельзя, в худшем случае у вас может быть 0 цифр (поэтому катастрофически).
В машинном обучении ваши данные берутся из меры, и нет меры с точностью до 9-й цифры (кроме очень немногих в физике элементарных частиц).
Необработанные значения из реального мира редко бывают такими точными, верно. Но ML не ограничивается таким вводом. Кто-то может захотеть применить ML к математическим задачам, например, применить MDS к графику головоломки, похожей на кубик Рубика, или кластеризовать успешные стратегии, найденные вашим роем агентов RL, играющих в pacman.
Даже если исходным источником информации является реальный мир, может быть некоторая промежуточная обработка, которая делает большинство цифр релевантными для алгоритма кластеризации. Как результат градиентного спуска функции, параметры которой статистически выбираются в реальном мире.
Мне вообще интересно, почему мы все еще обсуждаем это. Я думаю, мы все согласны с тем, что scikit-learn должен постараться найти компромисс между точностью и временем вычислений. И тот, кого не устраивает текущее состояние, должен отправить запрос на перенос.
Использование только «| b | ^ 2-2ab» не приводит к катастрофической отмене - но такая же потеря точности в цифрах, которые имеют значение. Результаты такие же, как если бы вы впоследствии добавили норму a к каждому расстоянию; если расстояния намного меньше нормы a, тогда вы получите потерю точности, которой можно избежать, выполняя вычисления традиционным способом без взлома BLAS.
Таким образом, вы НЕ МОЖЕТЕ решить числовую проблему!
Имеется потеря точности, но это не может вызвать катастрофическую отмену (по крайней мере, когда a и b близки), и вы можете показать, что относительная ошибка на расстоянии (которое не является расстоянием) остается небольшой.
В случае KMeans, где вас интересует только нахождение ближайшего центра, у вас достаточно точности, чтобы сохранить правильный порядок. Если в конце вам нужна инерция, вы можете просто вычислить расстояния от каждой точки до центра ее кластера с помощью точной формулы.
Кроме того, KMeans не является проблемой выпуклой оптимизации, поэтому даже если вы позволите ему работать с tol = 0 до сходимости, вы окажетесь в локальных минимумах, которые могут быть далеко от глобальных минимумов (даже с инициализацией kmeans ++). Так что я бы предпочел запускать kmeans много раз с разными init и достаточно небольшим количеством итераций. У вас будет больше шансов попасть в лучшие локальные минимумы. Затем вы можете повторить лучший результат до схождения.
Относительная ошибка по сравнению с реальным расстоянием может быть произвольно большой и, следовательно, вызывать неправильные ближайшие соседи. Рассмотрим случай, когда | a | ² = | b | ² = 1, например, на tf-idf. Предположим, что векторы очень близки. Тогда ab также близко к 1, и к этому моменту вы уже потеряли большую часть своей точности.
Как я уже писал выше, ошибка есть, даже если у вас нет катастрофической отмены. Учтите, что точность составляет 8 знаков. Реальное расстояние может быть 0,000012345678 и может быть вычислено с помощью восьми цифр с использованием FP32 и обычного евклидова расстояния. Но с помощью этого уравнения вместо этого вы вычисляете значение ab = 0,99998765432, которое с FP32 будет в лучшем случае усечено примерно до 0,99998765, поэтому в этом примере вы без необходимости потеряли три цифры точности. Убыток такой же большой, как и в случае катастрофы. Если расстояния намного меньше нормы, при таком подходе ваша точность может стать сколь угодно плохой.
Да, ксредн не выпуклый. Но тогда вы захотите хотя бы найти локальный оптимум, а не застревать (или даже колебаться, потому что результирующие ошибки ведут себя хаотично) из-за слишком низкой точности. Таким образом, у вас, по крайней мере, есть шанс найти глобальный вариант в хорошо выполненных случаях и с несколькими попытками.
Я ценю это обсуждение, но что нам действительно нужно, так это решение, которое не хуже того, что мы делали до того, как перестали повышать качество до float64. В этом смысле решения @Celelibi для апкастинга было достаточно. Использование точного решения в малых размерах - это дополнительное улучшение того, что мы делали раньше.
Что касается будущей версии, чувствуете ли вы больше уверенности в том, что сможете эффективно определять, когда мы можем рассмотреть возможность точных вычислений в больших измерениях?
Я провел тест, чтобы оценить среднюю точность случая float64 со случайными числами. Я сравниваю 3 алгоритма: neumaier_sum((x-y)**2) , numpy.sum((x-y)**2) и X2 - 2*X.dot(Y.T) + Y2.T . Точный результат для сравнения был получен с помощью mpmath с точностью 256 бит.
X и Y имеют 100 образцов и переменное количество функций и заполнены случайными числами от -2 до 2.
На следующем гифке есть одно изображение на количество функций (от 1 до 200). На каждом изображении каждая точка представляет относительную ошибку квадрата евклидова расстояния между одним из 10000 пар векторов X и Y . Относительная ошибка умножается на 2 ^ 53 для удобочитаемости, что примерно соответствует единице ULP.
Кривые выше являются приблизительным распределением (с использованием оценки плотности ядра).

Обратите внимание, что графики были вырезаны на 6 ULP для удобства чтения. Это средний случай, а не худший. Погрешность расширенной формулы может быть довольно большой.
Мой анализ этого результата показывает, что в среднем относительная ошибка расширенной формулы может быть очень большой при небольшом количестве функций, но быстро стать похожей на ошибку разницы и числовой суммы. Порог составляет от 5 до 10 характеристик.
Я также в настоящее время пытаюсь найти верхнюю границу ошибки расширенной формулы, а также патологические примеры.
Я думаю, что @ kno10 беспокоит то, что нас часто интересуют случаи, когда
точки не распределяются случайным образом, но находятся рядом друг с другом или имеют единицу
норма.
Действительно, но мне нужно было убедиться, что на практике это не полная чушь. ^^
В завершение комментария выше: относительная погрешность формулы x²+y²-2ab кажется неограниченной. Если мой анализ не ошибочен, когда x и y находятся близко друг к другу, относительная ошибка может достигать 2^(52*2) . По крайней мере теоретически. На практике, худший случай, который я обнаружил, - это относительная ошибка 2^52+1 .
>>> a, b = (0xfffffec4d6282+1) * 2.0**(511-52), 0xfffffec4d6282 * 2.0**(511-52)
>>> a, b
(6.703903473040778e+153, 6.7039034730407766e+153)
>>> exactdiff = (a-b)**2
>>> exactdiff
2.2158278651204453e+276
>>> computeddiff = a**2 + b**2 - 2*a*b
>>> computeddiff
-9.9792015476736e+291
>>> abs((computeddiff - exactdiff) / exactdiff)
4503599627370497.0
>>> bin(int(abs((computeddiff - exactdiff) / exactdiff)))
'0b10000000000000000000000000000000000000000000000000001'
Изменение знака результата на самом деле приблизит его к истине. Это наиболее драматичный пример, который я смог найти, но фактическое изменение показателя степени в значениях a и b не меняет относительной ошибки.
>>> a, b = (0xfffffec4d6282+1) * 2.0**(-52), 0xfffffec4d6282 * 2.0**(-52)
>>> a, b
(0.9999999266202912, 0.999999926620291)
>>> exactdiff = (a-b)**2
>>> computeddiff = a**2 + b**2 - 2*a*b
>>> abs((computeddiff - exactdiff) / exactdiff)
4503599627370497.0
Я думаю, что график гистограммы в ULP будет иметь больше смысла, чем приведенная выше анимация с распределением ошибок внутри ULP. Таким образом, 0 ошибок ULP и 1 ошибка ULP "настолько хороши, насколько это возможно". 2 ULP, вероятно, неизбежен из-за sqrt. Полагаю, стоит изучить любые более крупные ошибки.
Использование (computed - exact) / exact разумно, если точное значение велико. Но как только мы получаем численные проблемы для точного значения, это становится очень нестабильным. В таких случаях, возможно, стоит использовать (computed-exact)/norm , т. Е. Посмотреть на точность наших вычислений расстояния по сравнению с входными данными, а не с полученными расстояниями.
Если у нас есть два одномерных значения, которые отличаются только на 1 ULP, и ошибка 2 ULP может показаться огромной; но мы уже находимся на разрешении входных данных, поэтому результаты весьма нестабильны.
Обратите внимание, что с несколькими измерениями мы можем получить более высокое разрешение входных данных.
Рассмотрим входные данные типа (1, 1e-16) vs. (1, 2e-16) . Например, если у нас есть постоянный атрибут во входных данных, скажем, белый пиксель в MNIST.
С уравнением, основанным на разностях, все будет хорошо, но с точечной версией будут проблемы, не так ли? Поэтому одномерных экспериментов может быть недостаточно для изучения этого.
Я думаю, что график гистограммы в ULP будет иметь больше смысла, чем приведенная выше анимация с распределением ошибок внутри ULP.
Не уверен, что понимаю, как вы это изобразили. Будет одна гистограмма на количество функций и на алгоритм. Я мало что могу сделать, кроме трехмерного сюжета или анимации.
Использование
(computed - exact) / exactразумно, если точное значение велико. Но как только мы получаем численные проблемы для точного значения, это становится очень нестабильным.
Я не уверен, что вы подразумеваете под нестабильным в этом контексте. Точное значение должно быть вычислено с учетом всех необходимых факторов.
(Кстати, я должен был вычислить относительную ошибку с произвольной точностью на моем графике, вместо того, чтобы сравнивать с точно округленным результатом. Я обновил свой график, странные волны исчезли.)
В таких случаях, возможно, стоит использовать
(computed-exact)/norm, т. Е. Посмотреть на точность наших вычислений расстояния по сравнению с входными данными, а не с полученными расстояниями.
Если я правильно понимаю вашу идею, вы бы предпочли сравнить абсолютную ошибку с величиной входных данных. Использование векторных норм в качестве агрегированной меры величины входов. В то время как стандартная относительная ошибка сравнивает ее с величиной точного результата.
Я думаю, что с помощью этой метрики вы пытаетесь определить, насколько ошибочен алгоритм. Но на самом деле это не кажется особенно полезным по нескольким причинам.
- На самом деле это не говорит о том, сколько цифр в результате неточно.
- На самом деле, большинство алгоритмов имеют оценку менее 1e-15. Даже расширенная формула (алгоритм на основе точек) будет иметь оценку, ограниченную чем-то вроде 5 ULP (входные данные) (приблизительная оценка, я не писал полное доказательство).
- И поскольку обе метрики - это просто измененная версия абсолютной ошибки
computed - exact, они будут ранжировать алгоритмы в том же порядке при оценке на одних и тех же входных данных.
Таким образом, это то же самое, что и обычная относительная ошибка, только с менее полезной интерпретацией значения (IMO).
Рассмотрим входные данные типа
(1, 1e-16)vs.(1, 2e-16). Например, если у нас есть постоянный атрибут во входных данных, скажем, белый пиксель в MNIST.
С уравнением, основанным на разностях, все будет хорошо, но с точечной версией будут проблемы, не так ли? Поэтому одномерных экспериментов может быть недостаточно для изучения этого.
Алгоритм, основанный на точках, будет иметь относительную ошибку 1 , что означает, что ошибка равна точному результату, и, следовательно, ни одна цифра результата не будет правильной. И ваша метрика будет иметь значение 1e-16 что означает, что относительно масштаба векторной нормы отключена только 16-я цифра.
Я не уверен, что вы пытаетесь показать этим примером.
Если нас по-прежнему беспокоит точность euclidean_distances с float64, возможно, лучше подвести итог этому обсуждению в новом выпуске, так как здесь 100 комментариев.
Самый полезный комментарий
Необработанные значения из реального мира редко бывают такими точными, верно. Но ML не ограничивается таким вводом. Кто-то может захотеть применить ML к математическим задачам, например, применить MDS к графику головоломки, похожей на кубик Рубика, или кластеризовать успешные стратегии, найденные вашим роем агентов RL, играющих в pacman.
Даже если исходным источником информации является реальный мир, может быть некоторая промежуточная обработка, которая делает большинство цифр релевантными для алгоритма кластеризации. Как результат градиентного спуска функции, параметры которой статистически выбираются в реальном мире.
Мне вообще интересно, почему мы все еще обсуждаем это. Я думаю, мы все согласны с тем, что scikit-learn должен постараться найти компромисс между точностью и временем вычислений. И тот, кого не устраивает текущее состояние, должен отправить запрос на перенос.