Scikit-learn: float32を使用したeuclidean_distancesの数値精度
説明
sklearn.metrics.pairwise.pairwise_distances関数は、np.float64配列を使用する場合はnp.linalg.normと一致しますが、np.float32配列を使用する場合は一致しないことに気付きました。 以下のコードスニペットを参照してください。
再現する手順/コード
import numpy as np
import scipy
import sklearn.metrics.pairwise
# create 64-bit vectors a and b that are very similar to each other
a_64 = np.array([61.221637725830078125, 71.60662841796875, -65.7512664794921875], dtype=np.float64)
b_64 = np.array([61.221637725830078125, 71.60894012451171875, -65.72847747802734375], dtype=np.float64)
# create 32-bit versions of a and b
a_32 = a_64.astype(np.float32)
b_32 = b_64.astype(np.float32)
# compute the distance from a to b using numpy, for both 64-bit and 32-bit
dist_64_np = np.array([np.linalg.norm(a_64 - b_64)], dtype=np.float64)
dist_32_np = np.array([np.linalg.norm(a_32 - b_32)], dtype=np.float32)
# compute the distance from a to b using sklearn, for both 64-bit and 32-bit
dist_64_sklearn = sklearn.metrics.pairwise.pairwise_distances([a_64], [b_64])
dist_32_sklearn = sklearn.metrics.pairwise.pairwise_distances([a_32], [b_32])
# note that the 64-bit sklearn results agree exactly with numpy, but the 32-bit results disagree
np.set_printoptions(precision=200)
print(dist_64_np)
print(dist_32_np)
print(dist_64_sklearn)
print(dist_32_sklearn)
期待される結果
sklearn.metrics.pairwise.pairwise_distancesの結果は、64ビットと32ビットの両方でnp.linalg.normと一致すると思います。 言い換えれば、私は次の出力を期待しています:
[ 0.0229059506440019884643266578905240749008953571319580078125]
[ 0.02290595136582851409912109375]
[[ 0.0229059506440019884643266578905240749008953571319580078125]]
[[ 0.02290595136582851409912109375]]
実績
上記のコードスニペットは、次の出力を生成します。
[ 0.0229059506440019884643266578905240749008953571319580078125]
[ 0.02290595136582851409912109375]
[[ 0.0229059506440019884643266578905240749008953571319580078125]]
[[ 0.03125]]
バージョン
Darwin-16.6.0-x86_64-i386-64bit
('Python', '2.7.11 | 64-bit | (default, Jun 11 2016, 03:41:56) \n[GCC 4.2.1 Compatible Apple LLVM 6.0 (clang-600.0.57)]')
('NumPy', '1.11.3')
('SciPy', '0.19.0')
('Scikit-Learn', '0.18.1')
全てのコメント102件
Python 3.5でも同じ結果:
Darwin-15.6.0-x86_64-i386-64bit
Python 3.5.1 (v3.5.1:37a07cee5969, Dec 5 2015, 21:12:44)
[GCC 4.2.1 (Apple Inc. build 5666) (dot 3)]
NumPy 1.11.0
SciPy 0.18.1
Scikit-Learn 0.17.1
これはユークリッド距離でのみ発生し、直接sklearn.metrics.pairwise.euclidean_distancesを使用して再現できます。
import scipy
import sklearn.metrics.pairwise
# create 64-bit vectors a and b that are very similar to each other
a_64 = np.array([61.221637725830078125, 71.60662841796875, -65.7512664794921875], dtype=np.float64)
b_64 = np.array([61.221637725830078125, 71.60894012451171875, -65.72847747802734375], dtype=np.float64)
# create 32-bit versions of a and b
a_32 = a_64.astype(np.float32)
b_32 = b_64.astype(np.float32)
# compute the distance from a to b using sklearn, for both 64-bit and 32-bit
dist_64_sklearn = sklearn.metrics.pairwise.euclidean_distances([a_64], [b_64])
dist_32_sklearn = sklearn.metrics.pairwise.euclidean_distances([a_32], [b_32])
np.set_printoptions(precision=200)
print(dist_64_sklearn)
print(dist_32_sklearn)
エラーをさらに追跡することはできませんでした。
これがお役に立てば幸いです。
numpyはより高精度のアキュムレータを使用する場合があります。 はい、こんな感じです
修正に値する。
2017年7月19日午前0時5分、「nvauquie」 [email protected]は次のように書いています。
Python 3.5でも同じ結果:
ダーウィン-15.6.0-x86_64-i386-64ビット
Python 3.5.1(v3.5.1:37a07cee5969、2015年12月5日、21:12:44)
[GCC 4.2.1(Apple Inc.ビルド5666)(ドット3)]
NumPy 1.11.0
SciPy 0.18.1
Scikit-Learn 0.17.1これはユークリッド距離でのみ発生し、を使用して再現できます。
直接sklearn.metrics.pairwise.euclidean_distances:scipyをインポートする
sklearn.metrics.pairwiseをインポートします互いに非常に類似した64ビットベクトルaとbを作成します
a_64 = np.array([61.221637725830078125、71.60662841796875、-65.7512664794921875]、dtype = np.float64)
b_64 = np.array([61.221637725830078125、71.60894012451171875、-65.72847747802734375]、dtype = np.float64)aとbの32ビットバージョンを作成する
a_32 = a_64.astype(np.float32)
b_32 = b_64.astype(np.float32)64ビットと32ビットの両方について、sklearnを使用してaからbまでの距離を計算します
dist_64_sklearn = sklearn.metrics.pairwise.euclidean_distances([a_64]、[b_64])
dist_32_sklearn = sklearn.metrics.pairwise.euclidean_distances([a_32]、[b_32])np.set_printoptions(precision = 200)
print(dist_64_sklearn)
print(dist_32_sklearn)エラーをさらに追跡することはできませんでした。
これがお役に立てば幸いです。—
このスレッドにサブスクライブしているため、これを受け取っています。
このメールに直接返信し、GitHubで表示してください
https://github.com/scikit-learn/scikit-learn/issues/9354#issuecomment-316074315 、
またはスレッドをミュートします
https://github.com/notifications/unsubscribe-auth/AAEz65yy8Aq2FcsDAcWHT8qkkdXF_MfPks5sPLu_gaJpZM4OXbpZ
。
できればこれに取り組みたいです
頑張れ!
したがって、問題は、ユークリッド距離の計算にsqrt(dot(x, x) - 2 * dot(x, y) + dot(y, y))を使用しているという事実にあると思います。
- (-2 * np.dot(X, Y.T) + (X * X).sum(axis=1) + (Y * Y).sum(axis=1)を試してみると、np.float32の答えは0になりますが、np.float64の正しい答えは得られます。
@jnothmanでは、私は何をすべきだと思いますか? 上記の私のコメントで述べたように、問題はおそらくsqrt(dot(x, x) - 2 * dot(x, y) + dot(y, y))を使用してユークリッド距離を計算することです
つまり、dotはproduct-then-sumよりも精度の低い結果を返しているということですか?
いいえ、私が言おうとしているのは、dotはproduct-then-sumよりも正確な結果を返しているということです
-2 * np.dot(X, Y.T) + (X * X).sum(axis=1) + (Y * Y).sum(axis=1)は出力[[0.]]与えます
np.sqrt(((X-Y) * (X-Y)).sum(axis=1))は出力[ 0.02290595]
完全にスタンドアロンのスニペットを投稿していないこともあり、何をしているのかが明確ではありません。
前回の投稿をすばやく見ると、 [[0.]]と[0.022...]を比較しようとしている2つのもののサイズが同じではありません(コピーアンドペーストの問題かもしれませんが、わからないためわかりにくいです)。完全なスニペットがあります)。
申し訳ありませんが私の悪い
import numpy as np
import scipy
from sklearn.metrics.pairwise import check_pairwise_arrays, row_norms
from sklearn.utils.extmath import safe_sparse_dot
# create 64-bit vectors a and b that are very similar to each other
a_64 = np.array([61.221637725830078125, 71.60662841796875, -65.7512664794921875], dtype=np.float64)
b_64 = np.array([61.221637725830078125, 71.60894012451171875, -65.72847747802734375], dtype=np.float64)
# create 32-bit versions of a and b
X = a_64.astype(np.float32)
Y = b_64.astype(np.float32)
X, Y = check_pairwise_arrays(X, Y)
XX = row_norms(X, squared=True)[:, np.newaxis]
YY = row_norms(Y, squared=True)[np.newaxis, :]
#Euclidean distance computed using product-then-sum
distances = safe_sparse_dot(X, Y.T, dense_output=True)
distances *= -2
distances += XX
distances += YY
print(np.sqrt(distances))
#Euclidean distance computed using (X-Y)^2
print(np.sqrt(row_norms(X-Y, squared=True)[:, np.newaxis]))
出力
[[ 0.03125]]
[[ 0.02290595136582851409912109375]]
最初の方法は、ユークリッド距離関数によって計算される方法です。
また、上記の意味を明確にするために、numpy関数を使用してもsum-then-productの精度が低くなるという事実がありました。
はい、これを複製できます。 最初に減算を行うことがわかります
差の精度を維持することができます。 ドットをする
現在行っているように、積を引いてから減算(または否定と加算)します。
最も有効数字がよりもはるかに大きいため、この精度が失われます
違い。
現在の実装は、多数の
特徴。 しかし、ユークリッド距離はますます無関係になると思います
高次元では、メモリは出力の数によって支配されます
値。
だから私はより数値的に安定した実装を採用することに投票します
d-現在の漸近的に効率的な実装。 意見、
@ogrisel? @agramfort?
最近float32を許可したので、これはもちろんもっと心配です
推定量全体でより一般的になります。
したがって、この例のproduct-then-sumはnp.float64に対して完全に正常に機能するため、考えられる解決策は、入力をfloat64に変換してから結果を計算し、変換された結果をfloat32に戻すことです。 これはより効率的だと思いますが、他の例でこれがうまくいくかどうかはわかりません。
float64への変換は、メモリ使用量においてより効率的ではありません。
減算。
そうそう、それについては申し訳ありませんが、float64を使用してから、product-then-sumを実行すると、メモリの観点からではなくても、計算効率が高くなると思います。
そして、product-then-sumを使用する理由は、メモリ効率ではなく、計算効率を高めるためでした。
確かに、しかしそれが実際にあると仮定する理由はないと思います
を実現する必要がないことを除いて、より計算効率が高い
中間配列。 絶対作業メモリーを制限すると仮定します(例:
チャンク)、なぜドット積を取り、ノルムを2倍および減算するのでしょうか
減算して二乗するよりもはるかに効率的ですか?
ベンチマークを提供しますか?
さて、私はPythonスクリプトを作成して、減算、次に2乗、float64、次に積、次に合計に変換するのにかかる時間を比較しました。非常に大きなベクトルとしてXとYを選択すると、2つの結果は大きく異なります。 。 また、 @ jnothmanはあなたが正しい減算
これが私が書いたスクリプトです。何か問題があれば教えてください
import numpy as np
import scipy
from sklearn.metrics.pairwise import check_pairwise_arrays, row_norms
from sklearn.utils.extmath import safe_sparse_dot
from timeit import default_timer as timer
for i in range(9):
X = np.random.rand(1,3 * (10**i)).astype(np.float32)
Y = np.random.rand(1,3 * (10**i)).astype(np.float32)
X, Y = check_pairwise_arrays(X, Y)
XX = row_norms(X, squared=True)[:, np.newaxis]
YY = row_norms(Y, squared=True)[np.newaxis, :]
#Euclidean distance computed using product-then-sum
distances = safe_sparse_dot(X, Y.T, dense_output=True)
distances *= -2
distances += XX
distances += YY
ans1 = np.sqrt(distances)
start = timer()
ans2 = np.sqrt(row_norms(X-Y, squared=True)[:, np.newaxis])
end = timer()
if ans1 != ans2:
print(end-start)
start = timer()
X = X.astype(np.float64)
Y = Y.astype(np.float64)
X, Y = check_pairwise_arrays(X, Y)
XX = row_norms(X, squared=True)[:, np.newaxis]
YY = row_norms(Y, squared=True)[np.newaxis, :]
distances = safe_sparse_dot(X, Y.T, dense_output=True)
distances *= -2
distances += XX
distances += YY
distances = np.sqrt(distances)
end = timer()
print(end-start)
print('')
if abs(ans2 - distances) > 1e-3:
# np.set_printoptions(precision=200)
print(ans2)
print(np.sqrt(distances))
print(X, Y)
break
サンプル数だけでなく、サンプル数に応じてどのようにスケーリングするかをテストする価値があります。
機能の数...規範を取ることは、いくつかを計算する利点があるかもしれません
サンプルのペアごとに1回ではなく、サンプルごとに1回
2017年10月20日午前2時39分、「OsaidRehmanNasir」 [email protected]
書きました:
さて、Pythonスクリプトを作成して、
減算-次に二乗し、float64に変換してから積-次に-合計
XとYを非常に大きなベクトルとして選択すると、2
結果は大きく異なります。 また、 @ jnothman https://github.com/jnothman
あなたは正しい引き算でした-そして二乗はより速いです。
これが私が書いたスクリプトです。何か問題があれば教えてくださいnumpyをnpとしてインポート
scipyをインポートする
sklearn.metrics.pairwiseからimportcheck_pairwise_arrays、row_norms
sklearn.utils.extmathからimportsafe_sparse_dot
from timeit import default_timer as timerrange(9)のiの場合:
X = np.random.rand(1,3 *(10 i))。astype(np.float32)Y = np.random.rand(1,3 *(10 i))。astype(np.float32)X、Y = check_pairwise_arrays(X、Y)
XX = row_norms(X、squared = True)[:, np.newaxis]
YY = row_norms(Y、squared = True)[np.newaxis 、:]#product-then-sumを使用して計算されたユークリッド距離
距離= safe_sparse_dot(X、YT、dense_output = True)
距離* = -2
距離+ = XX
距離+ = YYans1 = np.sqrt(distances)
start = timer()
ans2 = np.sqrt(row_norms(XY、squared = True)[:, np.newaxis])
end = timer()
ans1!= ans2の場合:
print(end-start)start = timer() X = X.astype(np.float64) Y = Y.astype(np.float64) X, Y = check_pairwise_arrays(X, Y) XX = row_norms(X, squared=True)[:, np.newaxis] YY = row_norms(Y, squared=True)[np.newaxis, :] distances = safe_sparse_dot(X, Y.T, dense_output=True) distances *= -2 distances += XX distances += YY distances = np.sqrt(distances) end = timer() print(end-start) print('') if abs(ans2 - distances) > 1e-3: # np.set_printoptions(precision=200) print(ans2) print(np.sqrt(distances)) print(X, Y) break—
あなたが言及されたのであなたはこれを受け取っています。
このメールに直接返信し、GitHubで表示してください
https://github.com/scikit-learn/scikit-learn/issues/9354#issuecomment-337948154 、
またはスレッドをミュートします
https://github.com/notifications/unsubscribe-auth/AAEz6z5o2Ao_7V5-Lflb4HosMrHCeOrVks5st209gaJpZM4OXbpZ
。
とにかく、PRを提出しますか、@ ragnerok?
ええ、確かに、あなたは私に何をしてほしいですか?
より安定した実装を提供し、テストも失敗します
現在の実装、そして理想的には私たちが失わないことを示すベンチマーク
合理的な場合には、変更から多くのこと。
ベクトル化を使用して、行の各ペア間の距離を見つけることが可能かどうかを尋ねたいと思いました。 私はそれをベクトル化する方法を考えることができません。
行のペア間の違い(距離ではない)を意味しますか? numpy配列を使用している場合は、それを実行できます。 形状(n_samples1、n_features)および(n_samples2、n_features)の配列がある場合は、それを(n_samples1、1、n_features)および(1、n_samples2、n_features)に再形成し、減算を行う必要があります。
>>> X = np.random.randint(10, size=(10, 5))
>>> Y = np.random.randint(10, size=(11, 5))
X.reshape(-1, 1, X.shape[1]) - Y.reshape(1, -1, X.shape[1])
うん、本当に助けてくれてありがとう😄
また、X_norm_squaredとY_norm_squaredを使用しない、より安定した実装を提供するかどうかも尋ねたいと思いました。 それで、私もそれらを議論から削除しますか、それともそれが役に立たないことについて警告する必要がありますか?
それらは非推奨になると思いますが、最初にそれを保証する必要があるかもしれません
そのバージョンを維持する必要がある場合はありません。
これを変更する際には、細心の注意を払います。 それは広く使用されており、
長年の実装。 重要なものを遅くしないように注意する必要があります
ケース。 大量のメモリを回避するために、チャンクで操作を実行する必要がある場合があります
使用法(これはおそらくこれが呼ばれるという事実によってトリッキーになります
からの出力メモリのリタイアを最小限に抑えるためにチャンクする関数内
ペアワイズ距離)。
計算について知っている他のコア開発者から本当に聞きたいです
コストと数値精度... @ ogrisel 、 @ lesteve 、@ rth .. ..
2017年11月5日午前5時27分、「OsaidRehmanNasir」 [email protected]
書きました:
私はまた、私がそうしないより安定した実装を提供するかどうか尋ねたかった
X_norm_squaredとY_norm_squaredを使用します。 だから私はそれらをから削除しますか
議論も同様ですか、それとも役に立たないことについて警告する必要がありますか?—
あなたが言及されたのであなたはこれを受け取っています。
このメールに直接返信し、GitHubで表示してください
https://github.com/scikit-learn/scikit-learn/issues/9354#issuecomment-341919282 、
またはスレッドをミュートします
https://github.com/notifications/unsubscribe-auth/AAEz63izdpQGDEuW32m8Aob6rrsvV6q-ks5szKyHgaJpZM4OXbpZ
。
しかし、PRを開くと、正確に話し合うのが簡単になります
では、この関数の非常に基本的な実装でPRを開きます。
問題は、0.20リリースでこれについて何をすべきかということです。 考慮できるいくつかの単純/一時的な改善(メモリ使用量などを犠牲にしてイベント)がありますか?
#11271で提案されたソリューションと分析は間違いなく非常に価値がありますが、これが最適なソリューションであることを確認するには、さらに議論が必要になる場合があります。 特に、CPUの種類などに応じて、 https://github.com/scikit-learn/scikit-learn/issues/11506で最適なグローバルワーキングメモリについて保留中の議論が行われていることを懸念してい
リリース@ jnothman @ amueller @ogriselのこの問題について、何をすべきだと思いますか?
安定性は効率よりも優れています。 安定性の問題は、次の場合でも修正する必要があります
効率にはまだ微調整が必要です。
working_memoryの焦点は、大きなサンプルでシルエットのようなものを作ることでした
サイズは動作します。 また、効率も向上しましたが、それを微調整することができます
ライン。
euclidean_distancesの修正を次のように行う必要があると強く信じています
float32 in。作成できると仮定して、0.19で分割しました。
euclidean_distancesは、単純な方法で32ビットで機能します。
修正が必要であることに同意します。 ここでの私の懸念は、効率ではなく、コードベースの複雑さです。
一歩後退すると、scipyのユークリッド実装は10行のCコードのようで、32ビットの場合は64ビットにキャストするだけです。 最速ではないことは理解していますが、概念的には簡単に理解できます。 scikit-learnでは、このトリックを使用してBLASでの計算を高速化します。その後、 https://github.com/scikit-learn/scikit-learn/pull/10212で改善の可能性があり、ユークリッドのチャンクソリューションの可能性があり
このトピックの一般的な方向性についての入力を探しています(たとえば、その一部をscipyなどにアップストリームしてみてください)。
scipyはデータをコピーすることに関心がないようです...
PRに続いて0.21に移動します。
ブロッカーを削除しますか?
sqrt(dot(x, x) - 2 * dot(x, y) + dot(y, y))
dot(x、x)とdot(y、y)がdot(x、y)と同じ大きさである場合、壊滅的なキャンセルとして知られているため、数値的に不安定です。
これはFP32の精度に影響を与えるだけでなく、もちろんより顕著であり、はるかに早く失敗します。
これは、倍精度でもこれがどれほど悪いかを示す簡単なテストケースです。
import numpy
from sklearn.metrics.pairwise import euclidean_distances
a = numpy.array([[100000001, 100000000]])
b = numpy.array([[100000000, 100000001]])
print "skelarn:", euclidean_distances(a, b)[0,0]
print "correct:", numpy.sqrt(numpy.sum((a-b)**2))
a = numpy.array([[10001, 10000]], numpy.float32)
b = numpy.array([[10000, 10001]], numpy.float32)
print "skelarn:", euclidean_distances(a, b)[0,0]
print "correct:", numpy.sqrt(numpy.sum((a-b)**2))
sklearnは、sqrt(2)ではなく、両方の時間で0の距離を計算します。
分散と共分散の数値問題の説明-そしてこれはユークリッド距離を加速するこのアプローチに簡単に引き継がれます-ここで見つけることができます:
エーリヒシューベルト、マイケルガーツ。
(共)分散の数値的に安定した並列計算。
で:科学的および統計的データベース管理に関する第30回国際会議(SSDBM)の議事録、ボルツァーノ-ボーゼン、イタリア。 2018、10:1–10:12
実際には、y座標はそのテストケースから削除でき、正しい距離は簡単に1になります。この数値の問題をトリガーするプルリクエストを作成しました。
XA = np.array([[10000]], np.float32)
XB = np.array([[10001]], np.float32)
assert_equal(euclidean_distances(XA, XB)[0,0], 1)
上記の私の論文がこの問題の解決策を提供するとは思わない-ユークリッド距離をsqrt(sum(power()))として計算するだけで、シングルパスであり、妥当な精度を持っている。 損失は、すでに正方形を使用することです。つまり、dot(x、x)自体はすでに精度を失っています。
@amueller問題は予想よりも深刻である可能性があるため、ブロッカーラベルを再度追加することをお勧めします...
この非常に単純な例をありがとう。
このように実装されている理由は、はるかに高速だからです。 下記参照:
x = np.random.random_sample((1000, 1000))
%timeit euclidean_distances(x,x)
20.6 ms ± 452 µs per loop (mean ± std. dev. of 7 runs, 10 loops each)
%timeit cdist(x,x)
695 ms ± 4.06 ms per loop (mean ± std. dev. of 7 runs, 1 loop each)
操作の数は両方の方法で同じオーダーですが(2番目の方法では1.5倍)、行列行列の乗算に十分に最適化されたBLASライブラリを使用できるため、高速化が実現します。
これは、scikit-learnのいくつかの推定量にとって大きな減速になります。
はい。ただし、FP32では3〜4桁、 FP64では7〜8桁の精度で、かなりの精度が低下しますね。 特に、そのようなエラーは増幅する傾向があるので...
さて、今は大丈夫だと言っているのではありません。 :)
その間に解決策を見つける必要があると言っています。
計算を行うためにfloat64にキャストすることを提案するPR(#11271)があります。 Inは、float64の問題を修正しませんが、float32の精度を向上させます。
euclidean_distancesを使用する推定量を使用すると、精度が低下して間違った結果が得られる例はありますか?
私は確かにこれは大したことであり、0.21のブロッカーになるはずだと思います。 これは0.19で32ビットに導入された問題であり、離れるのは良い状況ではありません。 0.20の早い段階で解決できればよかったのですが、その間に#11271がマージされるのを見るのは大丈夫です。 私が知っているそのPRの唯一の問題は、メモリ効率のサラウンド最適化であり、それはうさぎの深い穴です。
この「高速」バージョンは長い間使用されてきましたが、常にfloat64で使用されています。 @ kno10は、精度に問題があることを知っています。 それが問題になる可能性があるときに私たちが解決し、より遅いがより確実なソリューションを使用するための優れた高速ヒューリスティックがありますか?
はい。ただし、FP32では3〜4桁、FP64では7〜8桁の精度で、かなりの精度が低下しますね。
非常に簡単な例でこの問題を説明していただきありがとうございます。
しかし、この問題はあなたが示唆するほど広範囲に及んでいるとは思いません。それは主に、基準に対して相互距離が小さいサンプルに影響を及ぼします。
次の図は、2e6のランダムサンプルペアの場合のこれを示しています。各1Dサンプルは[-100、100]の間隔にあります。 scikit-learnとscipyの実装間の相対誤差は、サンプル間の距離の関数としてプロットされ、L2ノルムで正規化されます。
d_norm(A, B) = d(A, B) / sqrt(‖A‖₂*‖B‖₂)
(それが正しいパラメーター化であるかどうかはわかりませんが、データスケールに対していくらか不変の結果を得るためだけです)、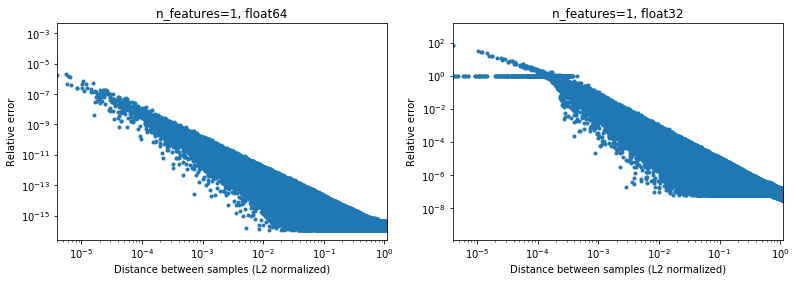
例えば、
[10000]と[10001]取る場合、L2正規化距離は1e-4であり、距離計算の相対誤差は64ビットで1e-8、32ビットで> 1(または1e)になります。絶対値でそれぞれ-8および> 1)。 32ビットでは、このケースは確かにかなりひどいです。- 一方、
[1]と[10001]場合、相対誤差は32ビットで〜1e-7、つまり可能な最大精度になります。
問題は、ケース1がMLアプリケーションで実際に発生する頻度です。
興味深いことに、2Dに移動すると、再び均一なランダム分布で、非常に近い点を見つけるのが困難になります。
もちろん、実際にはデータは均一にサンプリングされませんが、次元の呪いのためにどの分布でも、次元が増加するにつれて、任意の2点間の距離は非常に類似した値(0とは異なる)にゆっくりと収束します。 これは一般的なMLの問題ですが、ここでは、比較的低次元であっても、この精度の問題をいくらか軽減できる可能性があります。 n_features=5の結果の下に、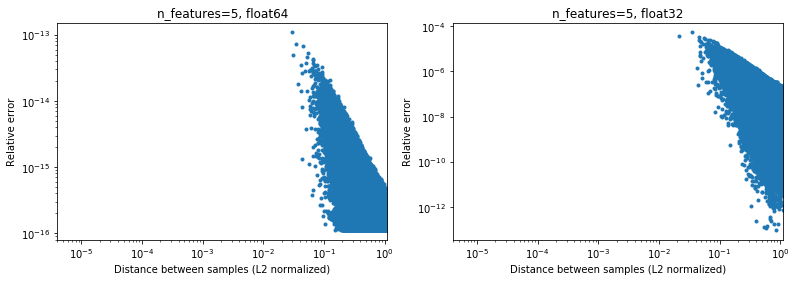 。
。
したがって、少なくとも64ビットの中央データの場合、実際にはそれほど問題にはならない可能性があります(2つ以上の機能があると仮定)。 (上に示したように)50倍の計算速度の向上はそれだけの価値があるかもしれません(64ビットで)。 もちろん、[-1、1]で正規化されたデータに常に1e6を追加して、結果が正確ではないと言うことはできますが、同じことが多くの数値アルゴリズムにも当てはまり、6番目に表現されたデータを処理することもできます。有効数字は問題を探しているだけです。
(上記の図のコードはここにあります)。
dot(x、x)+ dot(y、y)-2 dot(x、y)バージョンを使用する高速なアプローチで約2倍にするには、内積の精度を2倍にする必要があると思います。 の精度(ユーザーがfloat32データを提供する場合は、float32精度が必要であり、float64の場合は、float64精度が必要になると思います)。 いくつかのトリック(カハンの加算を考えてください)でこれを行うことができるかもしれませんが、それはあなたが最初に得たよりもはるかに多くの費用がかかる可能性が非常に高いです。
このアプローチを使用するためにfloat32をfloat64にオンザフライで変換することでどれだけのオーバーヘッドが得られるかはわかりません。 少なくともfloat32の場合、私の理解では、すべての計算を実行し、内積をfloat64として格納することは問題ないはずです。
私見では、パフォーマンスの向上(指数関数的ではなく、一定の係数)は、精度を低下させる価値がありません(予期せず噛み付く可能性があります)。適切な方法は、この問題のあるトリックを使用しないことです。 ただし、たとえばAVXを使用するなど、「従来の」計算を実行するコードをさらに最適化することは十分に可能です。 sum((xy) * 2)をAVXに実装するのはほとんど難しいためです。少なくとも、精度が低い場合があるため、メソッドの名前をapproximate_euclidean_distancesに変更することをお勧めします(2つの値が近いほど悪化し、*最初は問題なく、最適に収束するときに問題になります) 、ユーザーがこの問題を認識できるようにします。
@rthイラストありがとう
同様に、クラスタリングでは、クラスターの中心がすべてゼロに近いわけではありませんが、特にクラスターが多い場合は、数桁のx≈中心になる可能性があります。
ただし、全体として、この問題を修正する必要があることに同意します。 いずれにせよ、現在の実装の精度の問題をできるだけ早く文書化する必要があります。
一般的に、この議論はscikit-learnで行われるべきではないと思いますが。 ユークリッド距離は科学計算のさまざまな分野で使用されており、IMO scipyメーリングリストまたは問題はそれを議論するのに適した場所です。そのコミュニティは、そのような数値精度の問題についてもより多くの経験を持っています。 実際、ここにあるのは高速ですが、やや近似的なアルゴリズムです。 短期的にはいくつかの修正回避策を実装する必要があるかもしれませんが、長期的にはこれがそこで貢献されることを知っておくとよいでしょう。
32ビットの場合、 https://github.com/scikit-learn/scikit-learn/pull/11271は確かに解決策になる可能性があり
@ kno10のご回答ありがとうございます! (私の上記のコメントはまだそれを考慮していません)私は少し後で答えます。
はい、原点の外側のある点への収束が問題になる可能性があります。
私見では、パフォーマンスの向上(指数関数的ではなく、一定の係数)は、精度を低下させる価値がありません(予期せず噛み付く可能性があります)。適切な方法は、この問題のあるトリックを使用しないことです。
64ビットでの計算が10倍を超えると、ユーザーに非常に現実的な影響を及ぼします。
ただし、たとえばAVXを使用するなど、「従来の」計算を実行するコードをさらに最適化することは十分に可能です。 sum((xy)** 2)をAVXに実装するのはほとんど難しいためです。
numba(SSEを使用する必要があります)を使用して、簡単な単純な実装を試しました。
@numba.jit(nopython=True, fastmath=True)
def pairwise_distance_naive(A, B):
n_samples_a, n_features_a = A.shape
n_samples_b, n_features_b = B.shape
assert n_features_a == n_features_b
distance = np.empty((n_samples_a, n_samples_b), dtype=A.dtype)
for i in range(n_samples_a):
for j in range(n_samples_b):
psum = 0.0
for k in range(n_features_a):
psum += (A[i, k] - B[j, k])**2
distance[i, j] = math.sqrt(psum)
return distance
これまでのところ、scipy cdistと同様の速度が得られており(ただし、私はnumbaの専門家ではありません)、 fastmath効果についてもわかりません。
dot(x、x)+ dot(y、y)-2 * dot(x、y)バージョンを使用
将来の参考のために、現在行っていることは大まかに次のとおりです(上記の表記に示されていない次元があるため)、
def quadratic_pairwise_distance(A, B):
A2 = np.einsum('ij,ij->i', A, A)
B2 = np.einsum('ij,ij->i', B, B)
return np.sqrt(A2[:, None] + B2[None, :] - 2*np.dot(A, B.T))
ここで、 einsumとdot両方がBLASを使用するようになりました。 BLASを使用する以外に、これは実際には上記の最初のバージョンと同じ数の数学演算を実行するのではないかと思います。
BLASを使用する以外に、これは実際には上記の最初のバージョンと同じ数の数学演算を実行するのではないかと思います。
いいえ。((x--y) * 2.sum())は実行します* n_samples_x * n_samples_y * n_features *(1つの減算+1つの加算+1つの乗算)
一方、xx + yy-2x.yは
n_samples_x * n_samples_y * n_features *(1加算+ 1乗算) 。
2つのバージョン間の操作数には2/3の比率があります。
上記の議論に続いて、
- オプションでユークリッド距離を正確に計算できるようにするPRを作成しましたhttps://github.com/scikit-learn/scikit-learn/pull/12136
- https://github.com/scikit-learn/scikit-learn/pull/12142で問題のあるポイントを検出して軽減できるかどうかを確認するためのWIP
32ビットの場合でも、IMOを使用して、 https://github.com/scikit-learn/scikit-learn/pull/11271を何らかの形でマージする必要があり
FYI:光学でいくつかの問題を修正し、ELKIから使用の参照結果にテストをリフレッシュする場合、これらはで失敗metric="euclidean"が、成功してmetric="minkowski" 。 数値の違いは、処理順序が異なるほど大きくなります(しきい値を下げるだけでは不十分です)。
私は実際にはこれに追いついていないが、既存の解決策がないことに驚いている。 これは非常に一般的な計算のようで、車輪の再発明をしているようです。 誰かがより広い科学計算コミュニティに手を差し伸べようとしたことがありますか?
まだですが、そうすべきだと私は同意します。 scipyでこれについて私が見つけたのは、 https://github.com/scipy/scipy/pull/2815と関連する問題だけでした。
@jeremiedbbにアイデアがあるのではないかと思いますか?
残念ながらまだ満足のいくものではありません:(
OpenBLASやMKLなどのBLASライブラリを使用した線形代数の場合と同様に、この種の計算には高度に最適化されたライブラリを使用したいと考えています。 しかし、ユークリッド距離はその一部ではありません。 ドットトリックは、BLASレベル3の行列-行列乗算サブルーチンに依存してそれを行う試みです。 しかし、これは正確ではなく、同じ方法を使用してより正確にする方法はありません。 速度または精度のいずれかの観点から、期待値を下げる必要があります。
状況によっては、完全な精度が必須ではなく、高速な方法を維持することは問題ないと思います。 これは、距離が「最も近い」タスクを見つけるために使用される場合です。 高速メソッドの精度の問題は、ポイント間の距離がノルムと比較して小さい場合に発生します(float 32の場合は〜<1e-4、float64の場合は〜<1e-8の比率)。 まず、この状況が発生するためには、データセットが非常に密である必要があります。 次に、順序付けエラーが発生するには、ほぼ同じ距離内に最も近い2つのポイントが必要です。 さらに、その場合、MLの観点では、どちらもほぼ同等に良好な適合につながります。
上記の状況では、これらの間違った順序の頻度を下げるためにできることがあります(0?まで)。 ペアワイズ距離argminの状況で。 順序の誤りを、最も近くないポイントに移動できます。 基本的に、argminを見つけるために標準の1つが必要ではないという事実を使用して、コメントを参照してください。 2つの利点があります。 これはより堅牢で(今のところ間違った順序はまだ見つかりませんでした)、一部の計算を回避するため、さらに高速です。
1つの欠点は、同じ状況でも、最終的に最も近いポイントまでの実際の距離が必要な場合、上記の方法で計算された距離を使用できないことです。 それらは部分的にしか計算されておらず、とにかく正確ではありません。 各ポイントから最も近いポイントまでの距離を再計算する必要があります。 ただし、各ポイントに対して計算する距離は1つしかないため、これは高速です。
上で説明したことは、sklearnのeuclidean_distancesのすべてのユースケースをカバーしているのだろうか。 しかし、私はそれが適用できるところならどこでもそれをすることを提案します。 これを行うには、euclidean_distancesに新しいパラメーターを追加して、argminとチェーンするために必要な部分のみを計算します。 次に、pairwise_distances_argminとpairwise_distances_argmin_minで使用します(後者の最後で実際の最小距離を再計算します)。
それができない場合は、低速で正確なものにフォールバックするか、#12136のようにスイッチを追加します。
パフォーマンスの低下を抑えるために少し最適化を試みることができます。これは最適ではないように思われることに同意します。 そのためのアイデアがいくつかあります。
BLASを使い続けるもう1つの可能性は、 axpyとnrm2を組み合わせることですが、これは最適とは言えません。 どちらもBLASレベル1の機能であり、コピーが含まれます。 これは、100を超える次元でのみ高速になります。
理想的には、ユークリッド距離をBLASに含める必要があります...
最後に、アップキャストからなる別の解決策があります。 これは、float32の#11271で行われます。 利点は、速度が現在の半分であり、精度が維持されることです。 ただし、float64の問題は解決されません。 たぶん、float64のcythonで同様のことを行う方法を見つけることができます。 正確な方法はわかりませんが、2つのfloat64数値を使用してfloat128をシミュレートします。 ある程度実行可能かどうか試してみることができます。
理想的には、ユークリッド距離をBLASに含める必要があります...
それは図書館が検討することですか? OpenBLASがそれを行うなら、私たちはすでにかなり良い状況にあるでしょう...
また、私たちがそれを行うこととBLASがそれを行うことの正確な違いは何ですか? CPU機能を検出し、使用する実装を決定する、またはそのようなものですか? それとも、より多様なアーキテクチャのバージョンをコンパイルしただけですか?
それとも、効率的な実装を書くためにより多くの時間/エネルギーが費やされますか?
これは興味深いことです。高速で不安定なメソッドの代替実装ですが、sklearnよりもはるかに高速であると主張しています。
https://github.com/droyed/eucl_dist
(笑)この問題はまったく解決しません)
この議論は関連しているようですhttps://github.com/scipy/scipy/issues/5657
juliaの機能は次のとおりです: https :
これにより、再計算を強制するための精度のしきい値を設定できます。
私自身の質問に答える:OpenBLASには、各プロセッサ(アーキテクチャではありません!)の手書きのアセンブリのように見えるものと、さまざまな問題サイズのカーネルを選択するためのヒューリスティックがあります。 ですから、それらすべてのカーネルを作成/最適化する人を見つけることほど、openblasに組み込むことの問題ではないと思います...
追加の考えをありがとう!
部分的な応答では、
OpenBLASやMKLなどのBLASライブラリを使用した線形代数の場合と同様に、この種の計算には高度に最適化されたライブラリを使用したいと考えています。
ええ、私もBLASでこれをもっとできることを望んでいました。 前回、標準のBLAS APIで何も見えなかったので、十分に近いように見えます(ただし、私はそれらの専門家ではありません)。 BLISはより柔軟性を提供する可能性がありますが、デフォルトでは使用されていないため、使用が多少制限されています(ただし、numpyはいつかhttps://github.com/numpy/numpy/issues/7372になる可能性があります)
juliaの機能は次のとおりです。精度のしきい値を設定して再計算を強制できます。
知ってよかった!
上にリンクされたより高速な近似計算のために別の問題を開く必要がありますか? 面白そう
x2-x4のCPUでの高速化は、 https://github.com/scikit-learn/scikit-learn/pull/10212が原因である可能性があり
ユークリッド距離はML以外の多くの人々が興味を持つはずの基本的なものであると感じているので、この質問を十分に研究してそこで合理的な解決策を考え出す(そしておそらくそれをバックポートする)と、scipyで問題を開きたいと思います(同時に、精度の問題など、そこにいる人々の意見を聞くのは大変なことです)。
最大60倍ですよね?
これは興味深いです:高速で不安定なメソッドの代替実装ですが、sklearnよりもはるかに高速であると主張しています
それについてはよくわかりません。 彼らは、scipyのものを使用する%timeit pairwise_distances(a,b, 'sqeuclidean')ベンチマークを行っています。 彼らは%timeit pairwise_distances(a,b, 'euclidean', metric_params={'squared': True})をするべきであり、彼らのスピードアップはそれほど良くないでしょう:)
議論のずっと前に示したように、sklearnはscipyより35倍高速になる可能性があります
はい、彼らのベンチマークは、( squeclidean代わりに) metric="euclidean"使用すると、わずか30%向上します。
In [1]: from eucl_dist.cpu_dist import dist
... import numpy as np
In [4]: rng = np.random.RandomState(1)
... a = rng.rand(1000, 300)
...b = rng.rand(1000, 300)
In [7]: from sklearn.metrics.pairwise import pairwise_distances
In [8]: %timeit pairwise_distances(a, b, 'sqeuclidean')
214 ms ± 2.06 ms per loop (mean ± std. dev. of 7 runs, 1 loop each)
In [9]: %timeit pairwise_distances(a, b)
27.4 ms ± 2.48 ms per loop (mean ± std. dev. of 7 runs, 10 loops each)
In [10]: from eucl_dist.cpu_dist import dist
In [11]: %timeit dist(a, b, matmul='gemm', method='ext', precision='float32')
20.8 ms ± 330 µs per loop (mean ± std. dev. of 7 runs, 10 loops each)
In [12]: %timeit dist(a, b, matmul='gemm', method='ext', precision='float64')
20.4 ms ± 222 µs per loop (mean ± std. dev. of 7 runs, 10 loops each)
それは図書館が検討することですか? OpenBLASがそれを行うなら、私たちはすでにかなり良い状況にあるでしょう...
簡単に聞こえません。 BLASは線形代数ルーチンの一連の仕様であり、いくつかの実装があります。 元の仕様にない新機能を追加することに彼らがどれほどオープンであるかはわかりません。 そのため、blisはもっとオープンになるかもしれませんが、前に述べたように、今のところデフォルトではありません。
sqeuclideanとpairwise_distances euclidean処理について、 https://github.com/scikit-learn/scikit-learn/issues/12600を開きました。
これに何が欲しいのか明確にする必要があります。 'euclidean'と 'sqeuclidean'の両方でpairwise_distancesを( all_closeの意味で)近くに配置しますか?
少し注意が必要です。 xがyに近いからといって、x²がy²に近いという意味ではありません。 二乗中に精度が失われます。
上にリンクされているjuliaの回避策は非常に興味深いものであり、実装は簡単です。 ただし、「sqeuclidean」では期待どおりに機能しないと思われます。 希望の精度を得るには、以下のしきい値を設定する必要があると思います。
非常に低いしきい値を設定する場合の問題は、多くの再計算とパフォーマンスの大幅な低下を引き起こすことです。 ただし、これはデータセットのディメンションによって軽減されます。 同じしきい値を使用すると、高次元での再計算が大幅に少なくなります(距離が大きくなります)。
たぶん、データセットの次元に応じて2つの実装と切り替えが可能です。 低次元のものには低速ですが安全なもの(とにかくその場合はscipyとsklearnの間に大きな違いはありません)と高次元のものには高速+しきい値のものです。
これには、いつ切り替えるかを見つけてしきい値を設定するためのベンチマークが必要になりますが、これは希望のかすかな光かもしれません:)
scipyとsklearnの速度を比較するためのベンチマークをいくつか示します。 ベンチマークでは、すべてのサイズのXについて、 sklearn.metrics.pairwise.euclidean_distances(X,X)とscipy.spatial.distance.cdist(X,X)を比較しています。 サンプル数は2⁴(16)から2¹³(8192)になり、特徴の数は2⁰(1)から2¹³(8192)になります。
各セルの値は、sklearnとscipyの高速化です。つまり、1 sklearn未満は遅く、1sklearnを超えると速くなります。
最初のベンチマークは、BLASのMKL実装とシングルコアを使用することです。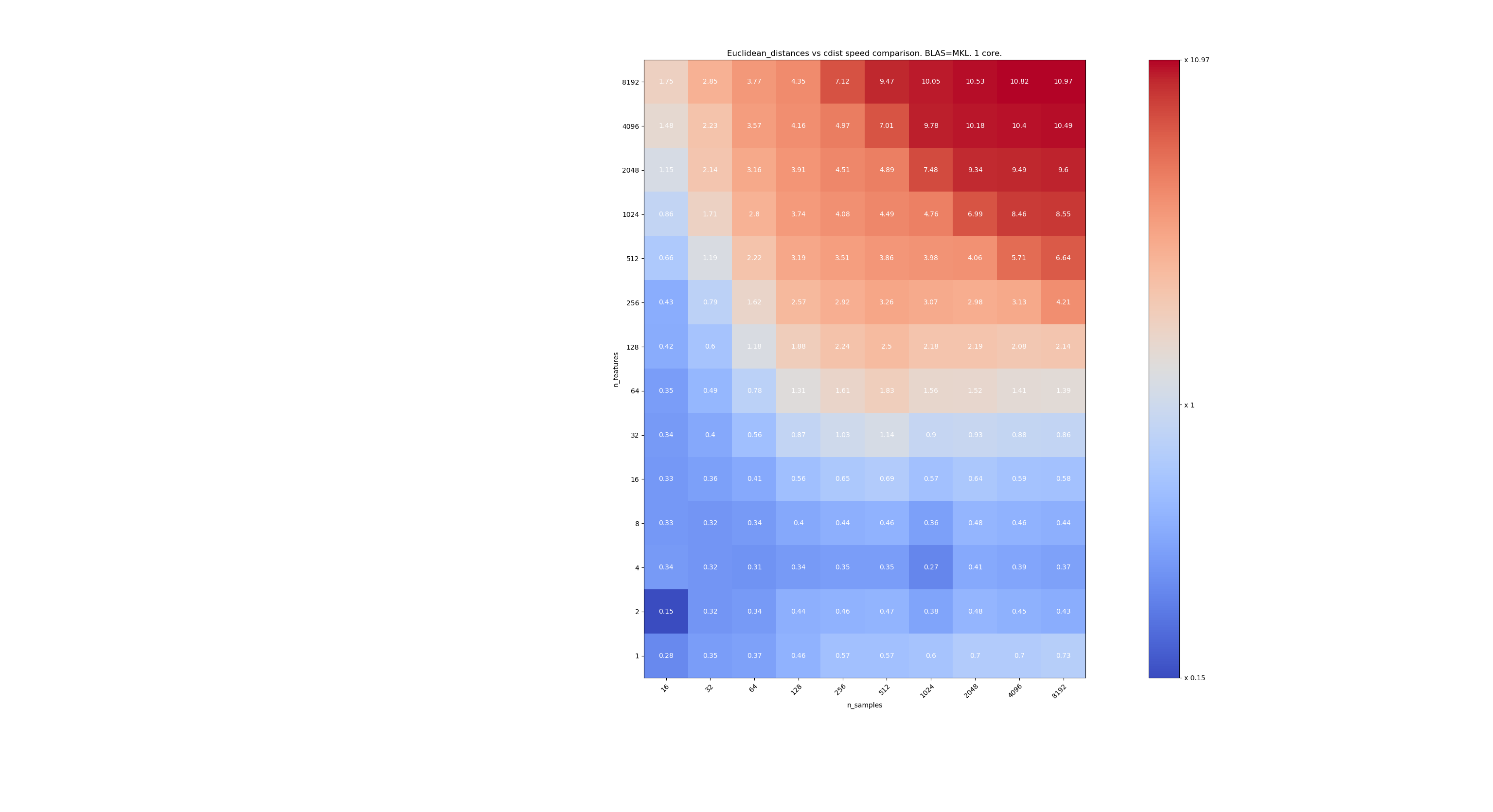
2つ目は、BLASのOpenBLAS実装とシングルコアを使用することです。 MKLとOpenBLASの両方が同じ動作をすることを確認するだけです。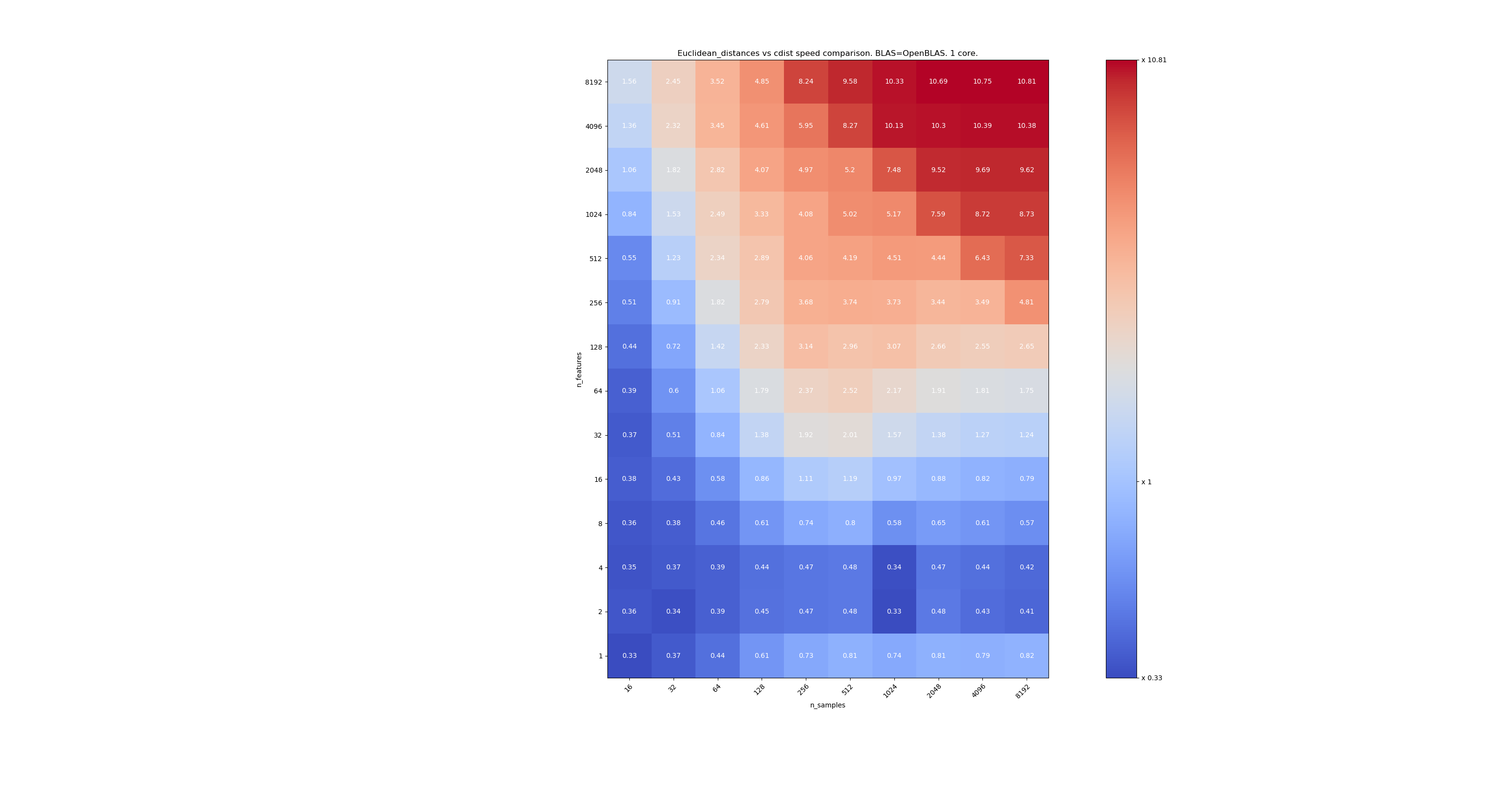
3つ目は、BLASと4コアのMKL実装を使用することです。 euclidean_distancesはBLAS LEVEL 3関数を介して並列化されますが、 cdistはBLAS LEVEL1関数のみを使用します。 興味深いことに、それはフロンティアをほとんど変えません。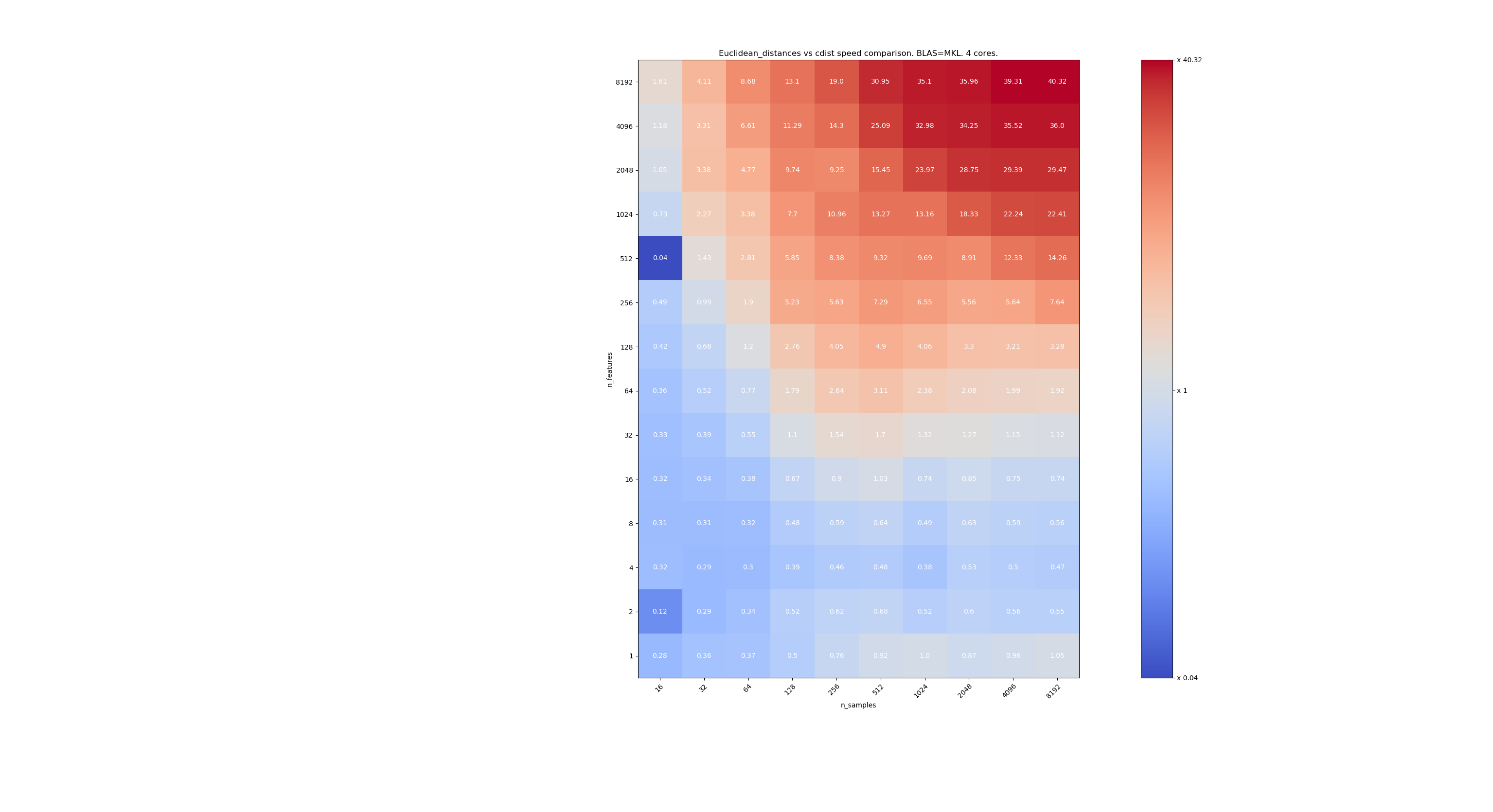
n_samplesが低すぎない場合(> 100)、フロンティアは約32のフィーチャであるように見えます。 n_features <32の場合はcdistを使用し、n_features> 32の場合はeucliden_distancesを使用することを決定できます。これはより高速で精度の問題はありません。 これには、n_featuresが小さい場合、juliaのしきい値が多くの再計算につながるという利点もあります。 cdistを使用すると、それを回避できます。
n_features> 32の場合、 euclidean_distances実装を維持し、juliaのしきい値で更新できます。 しきい値を追加しても、機能の数が十分に多く、再計算が数回しか必要ないため、 euclidean_distancesが遅くなりすぎないようにする必要があります。
@jeremiedbbすばらしい、分析していただきありがとうございます。 結論は私にとって素晴らしい方法のように思えます。
ああ、これはすべてfloat64の場合だったと思いますよね? float32で何をしますか? 常にアップキャスト? 32を超える機能のアップキャスト?
コメントを注意深く読んでいません(まもなく)、float64には制限があることを参考にしてください。#12128を参照してください。
@ qinhanmin2014はい、float64の精度には制限がありますが、私が知る限り、信頼できるfp32の結果を生成するのに十分な精度です。 問題は、fp64へのアップキャストがscipyのcdistを使用するよりも実際に安価なパラメーターです。
上記のベンチマークに見られるように、マルチコアBLASでさえ一般的に高速ではありません。 これは主に高次元データ(64次元以上、それ以前はIMHOの努力の価値がない)に当てはまるようです。ユークリッド距離は高密度の高次元データではそれほど信頼できないため、そのユースケースIMHOは最も重要ではありません。 。 多くのユーザーのディメンションは10未満です。 これらの場合、cdistは通常より速いようです?
ああ、これはすべてfloat64の場合だったと思いますよね?
実際には、float32とfloat64の両方に当てはまります(つまり、非常に似ています)。 n_features <32の場合は、常にcdistを使用することをお勧めします。
問題は、fp64へのアップキャストがscipyのcdistを使用するよりも実際に安価なパラメーターです。
アップキャストは約2倍遅くなるので、n_features = 64前後だと思います。
多くのユーザーのディメンションは10未満です。
しかし、全員ではないので、高次元データの解決策を見つける必要があります。
とても素敵な分析@jeremiedbb !
低次元のデータの場合、cdistを使用することは間違いなく理にかなっています。
また、FYI scipyのcdistはfloat32をfloat64にアップキャストしますhttps://github.com/scipy/scipy/issues/8771#issuecomment-384015674 、これが精度の問題によるものか、それとも他の原因によるものかはわかりません。
全体として、 https: //github.com/scikit-learn/scikit-learn/pull/12601#pullrequestreview -176076355で提案されているように、「algorithm」パラメーターをeuclidean_distanceに追加することは理にかなっていると思います。デフォルトは「なし」で、 https://github.com/scikit-learn/scikit-learn/pull/12136のようにグローバルオプションを介して設定することもでき
Eigen3には、安定した基準を計算するための興味深いアプローチもあります:
良い説明、私の理解を向上させた
スプリントではこれについて何の進展もありません。おそらくそうすべきです...そして@rthは今日は
時間を設定すればリモートで参加できます。 多分午後の初めに?
状況を要約すると、
ユークリッド距離計算の精度の問題については、
- 低次元の場合、 @ jeremiedbbが上に示したように、おそらくcdistを使用する必要があります
- 高次元の場合とfloat32では、次のいずれかを選択できます。
- チャンク化、64ビットでの距離の計算、および連結
- 精度が問題になる場合はcdistにフォールバックします(未解決の質問はどのようになりますか?たとえばscipyに連絡すると役立つ場合がありますhttps://github.com/scikit-learn/scikit-learn/issues/9354#issuecomment-438522881 )
次に、ユークリッド、スクイクリッド、ミンコフスキーなどの間の不整合のすべての問題があります。
精度に関しては、 @ jeremiedbb 、 @ amuellerと私は簡単なチャットをしましたが、ほとんどはジェレミーの専門知識を搾乳するだけでした。 彼は、float64の高次元でのMLコンテキストでの不安定性の問題についてそれほど心配する必要はないと考えています。 ジェレミーはまた、良い結果が返されたかどうかの効率的なテストを見つけるのは難しいことを示唆しました(#12142を参照)
したがって、 float32のアップキャストに関する@rthの前のコメントに満足していると思います。 cdistもfloat64にアップキャストするため、cdistを再実装してfloat32を取得するか(ただし、float64アキュムレータを使用しますか?)、低精度のfloat32でのコピーを減らしたい場合は、チャンクを使用できます。
@Celelibiは#11271のPRを変更したいですか、それとも他の誰か(私たちの1人?)が完全なプルリクエストを作成する必要がありますか?
そして、これが修正されたら、sqeuclideanとminkowski(p in {0,1})に実装を使用させる必要があると思います。 NearestNeighborsとの不一致については話し合いませんでした。 別のスプリント:)
スプリントで簡単に話し合った後、次のようになりました。
高次元の場合(> 32または> 64が最適を選択します):float32の場合はチャンクごとにfloat64にアップキャストし、「fast」メソッドを維持します。 この種のデータの場合、float64での数値の問題はほとんど無視できます(そのためのベンチマークを提供します)
低次元の場合:sklearnで安全な計算を実装します(アップキャストのためにscipy cdistを使用する代わりに)。
(upcasting float32を0.20.3にスローするのも魅力的です)
scipyとsklearnの速度を比較するためのベンチマークをいくつか示します。
[... をちょきちょきと切る ...]
これはとても興味深いです。 私は実際にこの結果を期待していませんでした。 ベンチマークをやり直したところ、非常によく似た結果が見つかりました。 私がより低い決定境界を主張することを除いて。 私のベンチマークは8つの機能を提案します。

間違っていることのコストは対称的ではありません。 cdistは、数秒未満の計算にのみ適しています。機能の数が増えると、非常に速く遅くなります。 したがって、疑わしい場合はBLAS実装を使用することをお勧めします。
編集:このベンチマークはfloat64用でしたが、float32行列をfloat64にアップキャストしても、合計時間にわずか数パーセントしか追加されず、結論は変わりません。
しきい値は、ベンチマークを実行しているマシンによって異なることに気付きました。 AVXの指示に関係しているのではないかと思います。 私が公開したベンチマークは、AVX2命令がなく、AVXのみのマシンで実行されていることに気付きました。 そして、AVX2を搭載したマシンで、私はあなたと同様の結果を得ました。
しかし、問題はパフォーマンスだけでなく精度にも関係しており、寸法が小さいと精度の問題が発生する可能性が高くなります。 たぶん16は良い妥協点です。 どう思いますか ?
この議論に関して、私たちは情報に基づいた決定を下すために正確さをベンチマークする必要があると思います。
ただし、PRに関しては、精度はもはや問題ではありません。 ただし、計算コストが少し高くなります。 したがって、しきい値はおそらくPRのベンチマークによって決定する必要があります。
ベンチマークの精度はそれほど簡単ではありません。 難しいケースは均一に分散されないからです。
また、コーナーケースで検出されない場合は問題が発生する可能性があります。 通常、手頃なCPU制限内で数値精度を保証する必要があります。
しかし、他の場所で述べたように、10000000.01と10000000.00の単一の機能は、既知の問題のある方程式10000と10001をfp32で使用するときに、fp64で数値の不安定性を引き起こすのに十分なはずです。 1024の機能で、試してみてください
>>> import sklearn.metrics.pairwise as sk, scipy.spatial.distance as sp
>>> X = [[10000.01] * 1024, [10000.00] * 1024]
>>> print(sk.euclidean_distances(X,X), "\n", sp.cdist(X,X))
[[ 0. 0.31895195]
[ 0.31895195 0. ]]
[[ 0. 0.32]
[ 0.32 0. ]]
(これは0.19.1を使用していました)正しい距離は0.32です。
ご覧のとおり、数値の不安定性は、特徴の数とともに悪化する傾向があります(データがスパースでない限り)。 ここで、結果はFP64で2桁未満の精度になります。
13410はこの特定のケースを修正しません。 つまり、float64 +高次元。
ただし、float32では修正されています。
ただし、float64 + high dimの場合、精度の問題が発生する可能性は非常に低く、機械学習のユースケースには実際には当てはまらないため、そのままにしておくことにしました。
あなたの例では、X [0]とX [1]のノルムは320000.32と320000に等しく、距離は0.32、つまりノルムの1e-6倍です。 機械学習では、有効数字16桁(float64内)がすべて関連しているわけではありません。
ただし、float64 + high dimの場合、精度の問題が発生する可能性は非常に低く、機械学習のユースケースには実際には当てはまらないため、そのままにしておくことにしました。
私はこれについてもっと穏健だろう。 次元削減は、MLの通常の最初のステップです。 MDSはそのために使用でき、ユークリッド距離行列を多用します。
float64の場合の精度の向上を検討したい場合は、2つのfloatを使用して中間結果を表す方法があります。 scikit-learnの範囲を超え始めていると思いますが。
ftp://ftp.math.ethz.ch/users/wpp/CELL/qd.pdf
はっきりしませんでした。 高次元データが機械学習に適用されないと言っているのではありません。 float64で発生する種類の精度の問題には、距離が標準より6桁小さいポイントが関係していると言っています。 このような精度を持つことは、現実的な機械学習モデルでは意味がありません
機械学習では、有効数字16桁(float64内)がすべて関連しているわけではありません。
私は、これが一般的に真実であるとはまったく確信していません。
この例では、16桁のうち15桁の精度が失われています。 精度の半分を使用するかどうかは同意しますが、そのような関係はありません。 FP64からFP32へのダウンキャストによる損失は、測定精度のために許容できる場合があります。 また、コンシューマーグレードのGPUは、たとえばFP64よりもFP32の方がはるかに高速であり(場合によっては、FP32データとFP64アキュムレーターが許可されるようになりました)、ニューラルネットワークの推論ではint8が表示されることもあります。 しかし、それはどこにでも当てはまるわけではありません。
たとえば、k-meansクラスタリングでは、クラスターの平均が大幅に異なる(そして、事前に平均がわからない)という仮定があるため、ここでは精度が低下します。 クラスターが多数ある場合、それらの基準のいくつかは、それらの分離と比較して大きくなる可能性があります。
さらに、最初の最初の反復の後、距離のわずかな違いにより、あるポイントが別のクラスターに切り替わることがよくあります。 ここで精度が失われると、結果に影響を及ぼし、不安定になる可能性があります。
ここで、多くの変数を持つ時系列フラグメントのk-meansについて考えます。
データサイズが大きくなると、最近傍までの距離が小さくなると想定する必要があります。ノルムが0でない限り、最終的にはベクトルノルムよりも小さくなり、問題が発生します。 したがって、これはデータセットのサイズが大きくなるにつれてさらに深刻になる可能性があります。 次元の呪いは、最大距離と最小距離がますます類似することを示しています。 したがって、正しい最近傍ランキングを計算するには、高次元データで十分な精度が必要になる場合があります。 20newsデータセットでは、ゼロ以外の最小距離は約0.02です(標準はすべて1です)。 しかし、それはたった1万のインスタンスであり、かなり多様なコンテンツです。 ここで、データセットがほぼ重複した検出に関するものであると仮定します...
この「ありそうもない」ことがMLで発生するかどうかはわかりませんが、もちろん、すべての人に影響するわけではありません。
「機械学習では、有効数字16桁(float64内)がすべて関連しているわけではありません。」と言うとき、私は計算された距離について話しているのではなく、データXについて話しているのです。
機械学習では、データはメジャーから取得され、9桁目まで正確なメジャーはありません(素粒子物理学のメジャーはごくわずかです)。
したがって、 10000000.01と10000000.00では、Xの値の不確実性がはるかに大きい場合、0.01の距離をどのように重要視しますか?
KMeansの場合、最初に、精度の低下の大部分を克服する方法があります。 観測値xの最も近い中心を探している場合、距離計算にxのノルムを追加する必要はありません。これにより、ほとんどの場合、壊滅的なキャンセルが回避されます。
次に、ユークリッド距離に基づいてkmeansクラスターを作成します。 しかし、これがデータの正確な収集方法であるかどうかはわかりません。 実際、データがそのようにクラスター化される確率は0です。 Kmeansは、データがどのようにクラスター化されるかを推定し、2つのクラスターのフロンティアにあるポイントは、確実にどちらかに属しているとは見なされません。 2つのクラスターの同じ距離にある点のあなたの解釈は何ですか? 私の場合、2つのクラスターは1つのクラスターのみである必要があるか、KMeansはデータをクラスター化するのに最適なアルゴリズムではありません(または、kmeansでさえ、データがどのようにクラスター化されるかについてある程度良いアイデアを与えてくれますが、クラスターのフロンティアは関係ありません)。
「| b | ^ 2-2ab」のみを使用しても壊滅的なキャンセルはありませんが、違いを生む桁の精度が同じように失われます。 結果は、後で各距離にaのノルムを追加した場合と同じです。 距離がaのノルムよりもはるかに小さい場合、BLASハックなしで従来の方法で計算を行うことで回避できる、精度の低下が発生します。
したがって、実際にはこの方法で数値問題を克服することはできません!
K-meansは最適化問題です。 したがって、そのようなハッキングは、sklearnが他のツールよりも悪い解決策しか見つけられないことを意味する場合があります。 そして前に示したように、これも不安定性を引き起こす可能性があります。 最悪の場合、これによりsklearn kmeansがmax_iterまで同じ状態を繰り返し、改善が見られなくなる可能性があります(局所最適を見つけたい場合は、tol = 0と仮定)。これは理論上不可能です。
k-meansが収束するまで、2つのクラスターまでの距離が「同じ」ポイントについては多くを語ることはできません。 次の反復では、平均が移動し、差がはるかに大きくなり、問題になる可能性があります。
ノイズの多いデータではうまく機能しないため、私はk-meansの大ファンではありません。 しかし、そのようなケースをより適切に処理するバリエーションがあります。 しかし、それでも、それを使用する場合は、おそらく完全な品質を取得しようとし(そのため、私も常にtol=0使用します)、必要以上に悪化させないようにする必要があります。 適切な計算を行うのに十分安価です(前述のように、問題はデータサイズとともに悪化します。したがって、小さいデータの場合、実行時間が遅くなることは通常問題ではありません。大きいデータセットの場合、精度が重要になる可能性が高くなります)。
アプリケーションによっては、 10000000.01と10000000.00の違いが問題になる場合があります。 また、前に示したように、複数の機能を使用すると、問題が早期に発生します。 fp32が10000と10001で、単一の機能があり、100対101で100の機能があると思います。
述べたように、平均はあなたが失いたくない物理的な意味を持っているかもしれません。 ケルビン単位の温度のデータがある場合、それらを0:1でスケーリングしたり、中央に配置したりする必要はありません。 それはあなたの比率スケールを台無しにするでしょう。 たとえば、ある鉄鋼製品の冷却時の温度の時系列を比較し、冷却プロセスが鉄鋼製品の信頼性に影響を与えるかどうかを調べたい場合。 700 Kを超える温度が発生している可能性があり、クールダウンプロセスを分析する場合は、時系列に数百のデータポイントが含まれる可能性があります。 時系列の長さでわずか5桁の入力精度(0.01K)でも、数値の問題が発生する可能性があります。 結果が1〜2桁になる場合もあります。 この壊滅的な種類の影響がある場合、MLで精度が重要になることを除外できるとは思いません。 たとえば、16桁のうち10桁の精度を常に取得できることを保証できる場合は、これは異なります。 ここではそれを行うことはできません。最悪の場合、0桁になる可能性があります(それが壊滅的な理由です)。
機械学習では、データはメジャーから取得され、9桁目まで正確なメジャーはありません(素粒子物理学のメジャーはごくわずかです)。
現実の世界からの生の値がそのような正確さを持っていることはめったにありません、そうです。 しかし、MLはそのような入力に限定されません。 ルービックキューブのようなパズルのグラフにMDSを適用したり、パックマンをプレイしているRLエージェントの群れが見つけた成功した戦略をクラスタリングしたりするなど、数学の問題にMLを適用することをお勧めします。
情報の最初のソースが実世界である場合でも、ほとんどの桁をクラスタリングアルゴリズムに関連させる途中の処理が行われる可能性があります。 パラメータが実世界で統計的にサンプリングされた関数の最急降下法の結果のように。
なぜ私たちはまだこれについて議論しているのか、私は実際に疑問に思っています。 scikit-learnは、計算時間とのトレードオフの精度で最善を尽くすべきであることに私たちは皆同意していると思います。 また、現在の状態に満足できない場合は、プルリクエストを送信する必要があります。
「| b | ^ 2-2ab」のみを使用しても壊滅的なキャンセルはありませんが、違いを生む桁の精度が同じように失われます。 結果は、後で各距離にaのノルムを追加した場合と同じです。 距離がaのノルムよりもはるかに小さい場合、BLASハックなしで従来の方法で計算を行うことで回避できる、精度の低下が発生します。
したがって、実際にはこの方法で数値問題を克服することはできません!
精度は低下しますが、壊滅的なキャンセルを引き起こすことはできず(少なくとも、aとbが近い場合)、距離(距離ではない)の相対誤差が小さいままであることを示すことができます。
最も近い中心を見つけることだけに関心があるKMeansの場合、順序を正しく保つのに十分な精度があります。 最後に慣性が必要な場合は、正確な式を使用して、クラスターの中心までの各ポイントの距離を計算できます。
さらに、KMeansは凸最適化の問題ではないため、収束するまでtol = 0で実行させても、(kmeans ++初期化を使用しても)グローバル最小値から大きく外れる可能性のあるローカル最小値になります。 したがって、異なるinitと適度に少ない反復回数でkmeansを何度も実行したいと思います。 より良い極小値に到達する可能性が高くなります。 次に、収束するまで最適なものを再実行できます。
実際の距離と比較した相対誤差は任意に大きくなる可能性があるため、最近傍が間違って発生します。 たとえばtf-idfで| a |²= | b |²= 1の場合を考えてみましょう。 ベクトルが非常に近いと仮定します。 その場合、abも1に近くなり、この時点ですでに精度の多くが失われています。
上で書いたように、壊滅的なキャンセルがなくても、エラーはそこにあります。 8桁の精度を考慮してください。 実際の距離は0.000012345678であり、FP32と通常のユークリッド距離を使用して8桁で計算できます。 ただし、この式では、代わりに値ab = 0.99998765432を計算します。これは、FP32ではせいぜい約0.99998765に切り捨てられるため、この例では3桁の精度が不必要に失われます。 損失は壊滅的な場合と同じくらい大きいです。 距離が標準よりもはるかに小さい場合、このアプローチでは精度が任意に悪くなる可能性があります。
はい、kmeansは凸状ではありません。 しかし、精度が低すぎるために、少なくとも局所的な最適点を見つけて、行き詰まらないようにする必要があります(または、結果として生じるエラーが不規則に動作するために振動することさえありません)。 したがって、少なくとも、正常に動作するケースで複数回試行することで、グローバルなものを見つける機会が得られます。
この議論に感謝しますが、私たちが本当に必要としているのは、float64へのアップキャストを停止する前に行っていたものよりも悪くないソリューションです。 その意味で、 @ Celelibiのアップキャストソリューションで十分でした。 低次元で正確なソリューションを使用することは、以前の作業をさらに改善することです。
将来のバージョンに関して、高次元での正確な計算をいつ検討するかを効率的に検出する自信がありますか?
ベンチマークを実行して、乱数を使用したfloat64ケースの平均精度を評価しました。 3つのアルゴリズムを比較します: neumaier_sum((x-y)**2) 、 numpy.sum((x-y)**2) 、 X2 - 2*X.dot(Y.T) + Y2.T 。 比較する正確な結果は、256ビットの精度でmpmathを使用して取得されています。
XとYは、100個のサンプルと可変数の特徴があり、-2から2までの乱数で埋められます。
次のgifには、機能の数(1〜200)ごとに1つの画像があります。 各画像の各ドットは、 XとYの10000ペアのベクトルの1つ間のユークリッド距離の2乗の相対誤差を表します。 読みやすさのために、相対誤差に2 ^ 53を掛けます。これは、おおよそULP単位に対応します。
上記の曲線はおおよその分布です(カーネル密度推定を使用)。

グラフは読みやすさのために6ULPでカットされていることに注意してください。 最悪のケースではなく、平均的なケースを示しています。 展開された式の誤差はかなり大きくなる可能性があります。
この結果の私の分析では、平均して、展開された式の相対誤差は、いくつかの機能で非常に大きくなる可能性がありますが、すぐに差とnumpysumの相対誤差に似たものになります。 しきい値は5〜10個の機能です。
私は現在、拡張された式のエラーの上限と病理学的例を見つけようとしています。
@ kno10の懸念は、私たちがしばしば
ポイントはランダムに分布していませんが、互いに近くにあるか、ユニットを持っています
ノルム。
確かに、私は実際に、それは完全なBSはないことを確信する必要がありました。 ^^
上記のコメントを完了するには、式x²+y²-2abの相対誤差に制限がないようです。 私の分析が間違っていない限り、 xとyが互いに近い場合、相対誤差は最大2^(52*2)ます。 少なくとも理論的には。 実際には、私が見つけた最悪のケースは、 2^52+1相対誤差です。
>>> a, b = (0xfffffec4d6282+1) * 2.0**(511-52), 0xfffffec4d6282 * 2.0**(511-52)
>>> a, b
(6.703903473040778e+153, 6.7039034730407766e+153)
>>> exactdiff = (a-b)**2
>>> exactdiff
2.2158278651204453e+276
>>> computeddiff = a**2 + b**2 - 2*a*b
>>> computeddiff
-9.9792015476736e+291
>>> abs((computeddiff - exactdiff) / exactdiff)
4503599627370497.0
>>> bin(int(abs((computeddiff - exactdiff) / exactdiff)))
'0b10000000000000000000000000000000000000000000000000001'
結果の符号を反転すると、実際にはそれが真実に近づくでしょう。 これは私が見つけた最も劇的な例ですが、実際にはaとbの値の指数を変更しても、相対誤差は変わりません。
>>> a, b = (0xfffffec4d6282+1) * 2.0**(-52), 0xfffffec4d6282 * 2.0**(-52)
>>> a, b
(0.9999999266202912, 0.999999926620291)
>>> exactdiff = (a-b)**2
>>> computeddiff = a**2 + b**2 - 2*a*b
>>> abs((computeddiff - exactdiff) / exactdiff)
4503599627370497.0
ULPのヒストグラムプロットは、ULP内のエラー分布を持つ上記のアニメーションよりも理にかなっていると思います。 したがって、0ULPエラーと1ULPエラーは「可能な限り良好」です。 2 sqrtのため、ULPは避けられない可能性があります。 より大きなエラーは、調査する価値があると思います。
正確さが大きい限り、 (computed - exact) / exactが妥当です。 しかし、正確な値について数値的な課題が発生すると、これは非常に不安定になります。 このような場合、代わりに(computed-exact)/normを使用する価値があります。つまり、導出された距離と比較するのではなく、入力データと比較した距離計算の精度を確認します。
1 ULPだけが異なる2つの1次元値があり、2ULPの誤差が大きいように見える場合があります。 しかし、すでに入力データの解像度に達しているため、結果は非常に不安定です。
複数の次元を使用すると、入力データの解像度が高くなる可能性があることに注意してください。
タイプ(1, 1e-16)と(1, 2e-16)入力データを検討してください。 たとえば、入力データに定数属性がある場合、たとえばMNISTの白いピクセルです。
差分ベースの方程式ではこれで問題ありませんが、ドットバージョンは問題になりますね。 そのため、これを研究するには一次元実験では不十分な場合があります。
ULPのヒストグラムプロットは、ULP内のエラー分布を持つ上記のアニメーションよりも理にかなっていると思います。
あなたがそれをどのように表現したかわかりません。 機能の数ごとおよびアルゴリズムごとに1つのヒストグラムがあります。 3Dプロットやアニメーション以外にできることはあまりありません。
正確さが大きい限り、
(computed - exact) / exactが妥当です。 しかし、正確な値について数値的な課題が発生すると、これは非常に不安定になります。
この文脈で不安定とはどういう意味かわかりません。 正確な値は、正確にするために必要なものを使用して計算する必要があります。
(そういえば、正確に丸められた結果と比較するのではなく、プロットでも任意精度で相対誤差を計算する必要がありました。プロットを更新すると、奇妙な波が消えました。)
このような場合、代わりに
(computed-exact)/normを使用する価値があります。つまり、導出された距離と比較するのではなく、入力データと比較した距離計算の精度を確認します。
私があなたの考えを正しく理解しているなら、あなたはむしろ絶対誤差を入力データの大きさと比較したいと思います。 入力の大きさの集約された尺度としてベクトルノルムを使用します。 一方、標準の相対誤差は、正確な結果の大きさと比較します。
このメトリックを使用して、アルゴリズムの欠陥の程度を把握しようとしていると思います。 しかし、実際にはいくつかの理由で特に有用ではないようです。
- 結果の何桁が不正確であるかは実際にはわかりません。
- 実際、ほとんどのアルゴリズムのスコアは1e-15未満です。 拡張された式(ドットベースのアルゴリズム)でさえ、5 ULP(入力)のようなもので囲まれたスコアを持ちます(概算、私は完全な証明を書きませんでした)。
- また、両方のメトリックは絶対誤差
computed - exact再スケーリングされたバージョンであるため、同じ入力で評価された場合、アルゴリズムは同じ順序でランク付けされます。
したがって、これは通常の相対誤差と同じですが、値の解釈があまり役に立たない(IMO)だけです。
タイプ
(1, 1e-16)と(1, 2e-16)入力データを検討してください。 たとえば、入力データに定数属性がある場合、たとえばMNISTの白いピクセルです。
差分ベースの方程式ではこれで問題ありませんが、ドットバージョンは問題になりますね。 そのため、これを研究するには一次元実験では不十分な場合があります。
ドットベースのアルゴリズムの相対誤差は1になります。これは、誤差が正確な結果と同じくらい大きいため、結果の桁が正しくないことを意味します。 また、メトリックの値は1e-16これは、ベクトルノルムのスケールに対して、16桁目のみがオフであることを意味します。
この例で何を表示しようとしているのかわかりません。
float64を使用したeuclidean_distancesの精度についてまだ懸念がある場合は、ここに100のコメントがあるため、この議論を新しい問題に要約する方がよいでしょう。
最も参考になるコメント
現実の世界からの生の値がそのような正確さを持っていることはめったにありません、そうです。 しかし、MLはそのような入力に限定されません。 ルービックキューブのようなパズルのグラフにMDSを適用したり、パックマンをプレイしているRLエージェントの群れが見つけた成功した戦略をクラスタリングしたりするなど、数学の問題にMLを適用することをお勧めします。
情報の最初のソースが実世界である場合でも、ほとんどの桁をクラスタリングアルゴリズムに関連させる途中の処理が行われる可能性があります。 パラメータが実世界で統計的にサンプリングされた関数の最急降下法の結果のように。
なぜ私たちはまだこれについて議論しているのか、私は実際に疑問に思っています。 scikit-learnは、計算時間とのトレードオフの精度で最善を尽くすべきであることに私たちは皆同意していると思います。 また、現在の状態に満足できない場合は、プルリクエストを送信する必要があります。