Data.table: fread mit großem csv (44 GB) benötigt viel RAM in der neuesten data.table dev-Version
Hi,
Hardware und Software:
Server : Dell R930 4-Intel Xeon E7-8870 v3 2.1GHz,45M Cache,9.6GT/s QPI,Turbo,HT,18C/36T und 1TB RAM
Betriebssystem : Redhat 7.1
R-Version : 3.3.2
data.table Version : 1.10.5 gebaut 2017-03-21
Ich lade eine CSV-Datei (44 GB, 872505 Zeilen x 12785 Spalten). Es lädt sehr schnell, in 1,30 Minuten mit 144 Kernen (72 Kerne von den 4 Prozessoren mit aktiviertem Hyperthreading, um es zu einer 144-Kerne-Box zu machen).
Das Hauptproblem besteht darin, dass beim Laden des DT die verwendete Speichermenge im Verhältnis zur Größe der CSV-Datei erheblich zunimmt. In diesem Fall verwendet die 44 GB csv (gespeichert mit fwrite, gespeichert mit saveRDS und Compress=FALSE eine Datei von 84 GB) ~ 356 GB RAM.
Hier ist die Ausgabe mit "verbose=TRUE"
_Zuweisung von 12785 Spaltenslots (12785 - 0 fallengelassen)
madvise sequentiell: ok
Lesen von Daten mit 1440 Sprungpunkten und 144 Threads
95,7% von 858881 geschätzten Zeilen lesen
872505 Zeilen x 12785 Spalten aus einer 43,772-GB-Datei in 1 Minute 33,736 Sekunden Wanduhrzeit lesen (beeinflusst durch andere laufende Apps)
0,000s ( 0%) Speicherzuordnung
0,070s ( 0%) Sep-, ncol- und Header-Erkennung
26,227s (28 %) Spaltentyperkennung mit 34832 Abtastzeilen von 1440 Sprungpunkten
0,614 s ( 1 %) Zuweisung von 3683116 Zeilen x 12785 Spalten (350,838 GB) im RAM
0.000s ( 0%) Madvise sequentiell
66,825s (71 %) Daten lesen
93.736s Gesamt_
Es zeigt ein ähnliches Problem, das manchmal beim Arbeiten mit dem parallelen Paket auftritt, bei dem eine rsession pro Kern gestartet wird, wenn Funktionen wie "mclapply" verwendet werden. Sehen Sie sich die erstellten/aufgeführten Rsessions in diesem Screenshot an:

wenn ich "rm(DT)" tue, geht der RAM in den Anfangszustand zurück und die "Rsessions" werden entfernt.
Schon versucht, zB "setDTthreads(20)" und immer noch die gleiche Menge an RAM verwenden.
Wenn die Datei übrigens mit der nicht parallelen Version von "fread" geladen wird, beträgt die Speicherzuweisung nur bis zu ~106 GB.
Guillermo
Alle 30 Kommentare
Dies ist die Ausgabe der nicht parallelen Fread-Implementierung (data.table 1.10.5 IN DEVELOPMENT build 2017-02-09)

Und ich habe die Menge des verwendeten Speichers doppelt überprüft und es geht einfach auf 84 GB.
Guillermo
Ja, du hast Recht. Danke für den tollen Bericht. Der geschätzte nrow sieht ungefähr richtig aus (858.881 vs. 872.505), aber dann ist die Zuteilung 4,2x größer (3.683.116) und weit daneben. Ich habe die Berechnung verbessert und der ausführlichen Ausgabe mehr Details hinzugefügt. Warte aber erst mal mit dem erneuten Testen, bis noch ein paar Dinge erledigt sind.
Ok bitte erneut testen - sollte jetzt behoben sein.
Ich habe gerade data.table dev installiert:
data.table 1.10.5 IN ENTWICKLUNG gebaut 2017-03-27 02:50:31 UTC
Das erste, was ich bekam, als ich versuchte, dieselbe 44-GB-Datei zu lesen, war diese Nachricht:
DT <- fread('dt.daily.4km.csv')
Fehler: protect(): Schutzstapelüberlauf
Dann führe ich einfach den gleichen Befehl erneut aus und fing an, gut zu funktionieren. Diese Version verwendet jedoch keinen Multi-Core-Modus. Das Laden dauert ~ 25 Minuten, genau wie vor der fread-parallelen Version.
Guillermo
Ich habe alle r-Sitzungen geschlossen und einen Test erneut ausgeführt, und ich erhalte eine Fehlermeldung:
Der geschätzte Spaltentyp war für 34711745 Werte in 508 Spalten nicht ausreichend. Verwenden Sie colClasses, um diese Spaltenklassen manuell festzulegen.
Siehe unten, ich erhalte mehrere Meldungen bezüglich "erratene Ganzzahl, enthält aber <<0....>>
_Lesen Sie 872505 Zeilen x 12785 Spalten aus der 43,772 GB-Datei in 15: 27,024 Uhr Wanduhrzeit (kann durch alle anderen geöffneten Apps verlangsamt werden, auch wenn sie scheinbar inaktiv sind)
Spalte 171 ('D_19810618') hat 'Ganzzahl' erraten, enthält aber <<2.23000001907349>>
Spalte 347 ('D_19811211') hat 'Ganzzahl' erraten, enthält aber <<1.02999997138977>>
Spalte 348 ('D_19811212') hat 'Ganzzahl' erraten, enthält aber <<3.75>>_
Wow - Ihre Datei testet wirklich die Randfälle. Groß. In Zukunft bitte mit verbose=TRUE ausführen und die vollständige Ausgabe bereitstellen. Aber mit den Informationen, die Sie in diesem Fall gemacht haben, kann ich sehen, wo das Problem tatsächlich liegt. Für jede Spalte für jeden Thread (in diesem Fall über 12.000 Spalten) wird ein Puffer erstellt. Jeder ist derzeit separat GESCHÜTZT. Es gibt einen Weg, das zu vermeiden - wird tun. Die Meldungen zum Typ Erraten sind richtig. Bedeuten Ihnen diese 508 Spalten etwas und Sie stimmen zu, dass sie numerisch sein sollten? Sie können einen Spaltenbereich wie folgt an colClasses : colClasses=list("numeric"=11:518)
Aus welchem Feld stammen diese Daten? Erstellst du die Datei? Es fühlt sich an, als wäre es in ein Breitformat verdreht worden, während die normale Best Practice darin besteht, im Langformat zu schreiben (und es auch im Speicher zu behalten). Normalerweise würde ich erwarten, dass die Namen der 508 Spalten wie "D_19810618" als Werte in einer Spalte angezeigt werden, nicht als Spalten selbst. Deshalb frage ich, ob Sie die Datei erstellen und sie im Langformat erstellen können. Wenn nicht, schlagen Sie demjenigen vor, der die Datei erstellt, dass er es besser machen kann. Ich vermute, dass Sie Operationen über Spalten anwenden, vielleicht mit .SD und .SDcols . Es ist wirklich viel besser im langen Format und keyby= die Spalte, die die Werte wie "D_19810618" enthält.
Aber ich werde trotzdem versuchen, fread im Umgang mit jeder Eingabe so gut wie möglich zu machen – sogar sehr große Dateien mit über 12.000 Spalten.
Ich hoffe, dass andere Leute testen und überhaupt keine Probleme mit ihren Dateien finden!
_ Bedeuten Ihnen diese 508 Spalten etwas und Sie stimmen zu, dass sie numerisch sein sollten?
Diese Tabelle enthält Zeitreihen nach Zeilen. IDx,IDy, Time1_value, Time2_value, Time3_value... und alle Time N _value-Spalten enthalten nur numerische Werte. Wenn ich colClasses verwende, muss ich dies für 12783 tun: list("numeric"=2:12783). Ich werde das versuchen.
_Aus welchem Feld stammen diese Daten?_
Geodaten. Ich mache euklidische Distanzsuchen innerhalb von DT mit IDx & IDy. Ich vermute, je mehr Zeilen, desto langsamer die Suche, oder?
Im Moment geht es ziemlich schnell (Breitformat). Ich habe eine Karte, auf der ein Benutzer auf ein Gebiet klicken kann und dann wird eine Zeitreihe in einer CSV-Datei vom nächstgelegenen Ort (mit verfügbaren Daten) zum angegebenen Klick generiert. Ich werde es stattdessen mit einem langen Format implementieren.
Ich melde mich mit einigen Ergebnissen zurück.
OK gut. Sie brauchen list("numeric"=2:12783) iiuc nicht, da es nur Hilfe bei 508 Spalten benötigt. Oh - ich verstehe - die 508 sind dann wohl über die Säulen verstreut (sie sind keine zusammenhängenden Säulen)?
Nein - data.table ist fast nie schneller, wenn es breit ist! Lang ist fast immer schneller und bequemer. Haben Sie roll="nearest" gesehen und ausprobiert? Wie geht es dir jetzt? Bitte zeigen Sie den Code, damit wir ihn verstehen können. Mit ziemlicher Sicherheit ist das lange Format besser, aber wir benötigen möglicherweise einige Verbesserungen für 2D-Nächstes. Bitte zeigen Sie auch die Zeiten an. Wenn Sie "ziemlich schnell" sagen, stellt sich heraus, dass die Leute sehr unterschiedliche Vorstellungen davon haben, was "ziemlich schnell" ist.
Schmelzen dieser Tabelle erreicht die Grenze von 2^31. Ich erhalte die Fehlermeldung: "Vektoren mit negativer Länge sind nicht zulässig".
Ich werde auf die Quelle zurückkommen, um zu sehen, ob ich sie im Langformat generieren kann.
# Read Data
DT <- fread('dt.daily.4km.csv', showProgress = FALSE)
# Add two columns with truncated values of x and y (these are geog. coords.)
DT[,y_tr:=trunc(y)]
DT[,x_tr:=trunc(x)]
# For using on plotting (x-axis values)
xaxis<-seq.Date(as.Date("1981-01-01"),as.Date("2015-12-31"), "day")
# subset by truncated coordinates to avoid full-table search. Now searches
# will happen in a smaller subset
DT2 <- DT[y_tr==trunc(y_clicked) & x_tr==trunc(x_clicked),]
# Add distance from each point in the data.table to the provided location, "gdist" is from
# Imap package for euclidean distance.
DT2[,DIST:=gdist(lat.1 = DT2$y,
lon.1 = DT2$x,
lat.2 = y_clicked,
lon.2 = x_clicked, units="miles")]
# Get the minimum distance
minDist <- min(DT2[,DIST])
# Get the y-axis values
yt <- transpose(DT2[DIST==minDist,3:(ncol(DT2)-3)])$V1`
# Ready to plot xaxis vs yt
...
...
Ich habe die Anwendung nicht auf einem öffentlichen Server. Im Wesentlichen kann der Benutzer auf eine Karte klicken und dann diese Koordinaten erfassen und die obige Suche durchführen, die Zeitreihen abrufen und ein Diagramm erstellen.
Einen weiteren Stapelüberlauf für eine sehr große Anzahl von Spalten gefunden und behoben: https://github.com/Rdatatable/data.table/commit/d0469e670961dcdea115d433c0f2dce596d65906. Haben Sie dieses Problem Nummer zu markieren, in der msg begehen.
Oh. Das ist ein Argument. 872505 Zeilen * 12780 Spalten sind 11 Milliarden Zeilen. Mein Vorschlag, ein langes Format zu verwenden, funktioniert also nicht für Sie, da dies > 2 ^ 31 ist. Entschuldigung - das hätte ich bemerken sollen. Wir müssen nur in den sauren Apfel beißen und gehen dann > 2^31. Bleiben wir in der Zwischenzeit beim Großformat, das Sie arbeiten, und ich werde das festnageln.
Bitte versuche es erneut. Die Speicherauslastung sollte wieder normal sein und die 508 von 12.785 Spalten mit Ausnahmen vom Typ "Out-of-Sample" automatisch neu gelesen werden. Um die Zeit für die automatische Wiederholung zu vermeiden, können Sie colClasses festlegen.
Wenn es nicht behoben ist, fügen Sie bitte die vollständige ausführliche Ausgabe ein. Daumen drücken!
Okay...
Eine Zusammenfassung der Ergebnisse mit dem neuesten data.table dev: data.table 1.10.5 IN DEVELOPMENT gebaut 2017-03-29 16:17:01 UTC
Vier Hauptpunkte sind zu erwähnen:
- fread hat gut geklappt, die Datei in der Diskussion zu lesen.
- Das Lesen dauerte ~ 5,5 Minuten im Vergleich zur vorherigen Version, die ~ 1,3 Minuten dauerte.
- Diese Version erhöht die RAM-Zuweisung nicht wie die vorherige Version, die ich getestet habe.
- Kerne scheinen "weniger aktiv" zu sein (siehe Screenshot unten).
Einige Kommentare/Fragen:
1.
Der Prozentsatz der Nutzung in den Kernen ist nicht mehr so aktiv wie zuvor, siehe Screenshot:
In der vorherigen Version von fread betrug die Aktivität der Kerne immer ~ 90-80%. In dieser Version blieben etwa 2-3 % pro Kern übrig, wie in der Abbildung oben gezeigt.
Ich verstehe nicht, warum Fread Bumps von Integer zu Numerisch macht: zB
Column 1489 ("D_19850126") bumped from 'integer' to 'numeric' due to <<0.949999988079071>> somewhere between row 6041 and row 24473
Ich habe diese Zeilen doppelt überprüft und es scheint in Ordnung zu sein. Warum wird als Integer erkannt und muss auf „numerisch“ umgestellt werden, wenn es bereits numerisch ist (siehe Zusammenfassung für die unten vorgeschlagenen Zeilen)? oder verstehe ich diese Zeile falsch? Dies geschieht für 508 Zeilen. Machen die NAs Ärger?
summary(DT[6041:24473,.(D_19850126)])
D_19850126
Min. :0.750
1st Qu.:0.887
Median :0.945
Mean :0.966
3rd Qu.:1.045
Max. :1.210
NA's :18393
Unten einige ausführliche aus dem Test.
DT<-fread('dt.daily.4km.ver032917.csv', verbose=TRUE)
AUSGANG
Parameter na.strings == <<NA>>
None of the 0 na.strings are numeric (such as '-9999').Input contains no \n. Taking this to be a filename to open
File opened, filesize is 43.772296 GB.
Memory mapping ... ok
Detected eol as \n only (no \r afterwards), the UNIX and Mac standard.
Positioned on line 1 starting: <<x,y,D_19810101,D_19810102,D_19810103,D_19810104,D_19810105,D_19810106,D_19810107,D_19810108,D_19810109,D_19810110,D_19810111,D_19810112,D_19810113,
...
...
All the fields on line 1 are character fields. Treating as the column names.
Number of sampling jump points = 11 because 1281788 startSize * 10 NJUMPS * 2 = 25635760 <= -244636240 bytes from line 2 to eof
Type codes (jump 00) : 441111111111111111111111111111111111111111111111111111111111111111111111111111111111111111...1111111111 Quote rule 0
Type codes (jump 01) : 444422222222222222242444424444442222222222424444424442224424222244222222222242422222224422...4442244422 Quote rule 0
Type codes (jump 02) : 444422222222222222242444424444442422222242424444424442224424222244222222222242424222424424...4442444442 Quote rule 0
Type codes (jump 03) : 444422222222222224242444424444444422222244424444424442224424222244222222222244424442424424...4442444442 Quote rule 0
Type codes (jump 04) : 444444244422222224442444424444444444242244444444444444424444442244444222222244424442444444...4444444444 Quote rule 0
Type codes (jump 05) : 444444244422222224442444424444444444242244444444444444424444442244444222222244424442444444...4444444444 Quote rule 0
Type codes (jump 06) : 444444444422222224442444424444444444242244444444444444424444444444444244444444424442444444...4444444444 Quote rule 0
Type codes (jump 07) : 444444444422222224442444424444444444242244444444444444444444444444444244444444424444444444...4444444444 Quote rule 0
Type codes (jump 08) : 444444444442222224444444424444444444242244444444444444444444444444444444444444424444444444...4444444444 Quote rule 0
Type codes (jump 09) : 444444444442222424444444424444444444244444444444444444444444444444444444444444424444444444...4444444444 Quote rule 0
Type codes (jump 10) : 444444444442222424444444424444444444244444444444444444444444444444444444444444424444444444...4444444444 Quote rule 0
=====
Sampled 305 rows (handled \n inside quoted fields) at 11 jump points including middle and very end
Bytes from first data row on line 2 to the end of last row: 47000004016
Line length: mean=45578.20 sd=33428.37 min=12815 max=108497
Estimated nrow: 47000004016 / 45578.20 = 1031195
Initial alloc = 2062390 rows (1031195 + 100%) using bytes/max(mean-2*sd,min) clamped between [1.1*estn, 2.0*estn]
=====
Type codes (colClasses) : 444444444442222424444444424444444444244444444444444444444444444444444444444444424444444444...4444444444
Type codes (drop|select): 444444444442222424444444424444444444244444444444444444444444444444444444444444424444444444...4444444444
Allocating 12785 column slots (12785 - 0 dropped)
Reading 44928 chunks of 0.998MB (22 rows) using 144 threads
Read 872505 rows x 12785 columns from 43.772GB file in 05:26.908 wall clock time (can be slowed down by any other open apps even if seemingly idle)
Final type counts
0 : drop
0 : logical
0 : integer
0 : integer64
12785 : numeric
0 : character
Rereading 508 columns due to out-of-sample type exceptions.
Column 171 ("D_19810618") bumped from 'integer' to 'numeric' due to <<2.23000001907349>> somewhere between row 6041 and row 24473
Column 347 ("D_19811211") bumped from 'integer' to 'numeric' due to <<1.02999997138977>> somewhere between row 6041 and row 24473
Column 348 ("D_19811212") bumped from 'integer' to 'numeric' due to <<3.75>> somewhere between row 6041 and row 24473
Column 643 ("D_19821003") bumped from 'integer' to 'numeric' due to <<1.04999995231628>> somewhere between row 6041 and row 24473
Column 1066 ("D_19831130") bumped from 'integer' to 'numeric' due to <<1.46000003814697>> somewhere between row 6041 and row 24473
Column 1102 ("D_19840105") bumped from 'integer' to 'numeric' due to <<0.959999978542328>> somewhere between row 6041 and row 24473
Column 1124 ("D_19840127") bumped from 'integer' to 'numeric' due to <<0.620000004768372>> somewhere between row 6041 and row 24473
Column 1130 ("D_19840202") bumped from 'integer' to 'numeric' due to <<0.540000021457672>> somewhere between row 6041 and row 24473
Column 1489 ("D_19850126") bumped from 'integer' to 'numeric' due to <<0.949999988079071>> somewhere between row 6041 and row 24473
Column 1508 ("D_19850214") bumped from 'integer' to 'numeric' due to <<0.360000014305115>> somewhere between row 6041 and row 24473
...
...
Reread 872505 rows x 508 columns in 05:29.167
Read 872505 rows. Exactly what was estimated and allocated up front
Thread buffers were grown 0 times (if all 144 threads each grew once, this figure would be 144)
=============================
0.000s ( 0%) Memory map
0.093s ( 0%) sep, ncol and header detection
0.186s ( 0%) Column type detection using 305 sample rows from 44928 jump points
0.600s ( 0%) Allocation of 872505 rows x 12785 cols (192.552GB) plus 1.721GB of temporary buffers
326.029s ( 50%) Reading data
329.167s ( 50%) Rereading 508 columns due to out-of-sample type exceptions
656.075s Total
Diese letzte Zusammenfassung hat viel Zeit in Anspruch genommen (~6 Minuten). Wenn wir diese ausführliche Zusammenfassung mitzählen, dauerte der ganze Fread ~ 11 Minuten. Es liest diese 508-Spalten erneut, ich kann sogar die Meldung "Neulesen von 508-Spalten aufgrund von Typausnahmen außerhalb der Stichprobe" sehen, ohne das Verbose=TRUE zu verwenden.
Schau es dir ohne "verbose=TRUE" an
ptm<-proc.time()
DT<-fread('dt.daily.4km.ver032917.csv')
Read 872505 rows x 12785 columns from 43.772GB file in 05:26.647 wall clock time (can be slowed down by any other open apps even if seemingly idle)
Rereading 508 columns due to out-of-sample type exceptions.
Reread 872505 rows x 508 columns in 05:30.276
proc.time() - ptm
user system elapsed
2113.100 85.919 657.870
fwrite test
Es funktioniert gut. Wirklich schnell. Ich dachte, dass das Schreiben heutzutage langsamer ist als das Lesen.
fwrite(DT,'dt.daily.4km.ver032917.csv', verbose=TRUE)
No list columns are present. Setting sep2='' otherwise quote='auto' would quote fields containing sep2.
maxLineLen=151187 from sample. Found in 1.890s
Writing column names ... done in 0.000s
Writing 872505 rows in 32315 batches of 27 rows (each buffer size 8MB, showProgress=1, nth=144) ...
done (actual nth=144, anyBufferGrown=no, maxBuffUsed=46%)
Das "Reread" dauert ziemlich lange. Es ist, als würde man die Datei zweimal lesen.
Exzellent! Danke für alle Infos.
- Ja, du hast Recht, es sollte nicht erneut gelesen werden. Sie haben
colClasses=list("numeric"=1:12785)daher sollte die Ausgabezeile, die mitType code (colClasses)beginnt, alle den Wert 4 haben. Wird den fehlenden Test korrigieren und hinzufügen. - Auf 5,5m vs 1,3m ist das interessant. Der Puffer jedes Threads beträgt derzeit 1 MB. Die Idee ist, klein zu sein, um in den Cache zu passen. Aber in Ihrem Fall 1 MB / 12785 Spalten = 82 Byte. Es ist also 50x zu ineffizient im Cache mit dieser Wahl. Ich vermute jedenfalls. Ohne deine Tests wäre mir das nie eingefallen. Als es mit einer Geschwindigkeit von 1,3 m arbeitete, hatte es nicht diese Größe von 1 MB.
- Warum es nur 305 Zeilen abgetastet hat, ist auch sehr seltsam. Es sollte 1.000 Proben haben. Wenn das behoben ist, erkennt es möglicherweise die 508-Spalten, ohne dass Sie es müssen. Die Stichprobe nimmt keine Zeit in Anspruch (0,186s), sodass die Stichprobengröße erhöht werden könnte.
Können Sie bitte die Ausgabe des Unix-Befehls lscpu einfügen. Dies wird uns Ihre Cache-Größen sagen und ich kann von dort aus denken. Ich werde buffMB als Parameter für fread bereitstellen, damit Sie sehen können, ob es das ist. Wenn ja, kann ich mir eine bessere Berechnung einfallen lassen.
Mein Fehler in einem Punkt meines vorherigen Posts. Das colClasses=list("numeric"=1:12785) funktioniert einwandfrei. Wenn ich "colClasses" nicht angebe, wird "Rereading" durchgeführt. Entschuldigung für die Verwirrung.
Eine Sache, die mir aufgefallen ist, ist, dass, wenn ich die "colClasses" nicht angebe, die Tabelle mit NAs erstellt wird und RAM angezeigt wird, als ob das DT normal geladen wurde (~ 106 MB im RAM).
Hier ist die lscpu- Ausgabe:
Architecture: x86_64
CPU op-mode(s): 32-bit, 64-bit
Byte Order: Little Endian
CPU(s): 144
On-line CPU(s) list: 0-143
Thread(s) per core: 2
Core(s) per socket: 18
Socket(s): 4
NUMA node(s): 4
Vendor ID: GenuineIntel
CPU family: 6
Model: 63
Model name: Intel(R) Xeon(R) CPU E7-8870 v3 @ 2.10GHz
Stepping: 4
CPU MHz: 2898.328
BogoMIPS: 4195.66
Virtualization: VT-x
L1d cache: 32K
L1i cache: 32K
L2 cache: 256K
L3 cache: 46080K
NUMA node0 CPU(s): 0,4,8,12,16,20,24,28,32,36,40,44,48,52,56,60,64,68,72,76,80,84,88,92,96,100,104,108,112,116,120,124,128,132,136,140
NUMA node1 CPU(s): 1,5,9,13,17,21,25,29,33,37,41,45,49,53,57,61,65,69,73,77,81,85,89,93,97,101,105,109,113,117,121,125,129,133,137,141
NUMA node2 CPU(s): 2,6,10,14,18,22,26,30,34,38,42,46,50,54,58,62,66,70,74,78,82,86,90,94,98,102,106,110,114,118,122,126,130,134,138,142
NUMA node3 CPU(s): 3,7,11,15,19,23,27,31,35,39,43,47,51,55,59,63,67,71,75,79,83,87,91,95,99,103,107,111,115,119,123,127,131,135,139,143
OK habe es. Vielen Dank.
Wenn Sie Ihre ersten Kommentare noch einmal lesen, wäre es sinnvoller, wenn "Bumping von Integer auf Double" statt "Bumping von Integer auf Numerisch" steht?
Was meinst du mit 'mit NA's erstellt'? Die ganze Tabelle ist voller NAs, nur die 508 Spalten?
Was ist 'DT wurde normal geladen (~106 MB im RAM)'. Es ist eine 44-GB-Datei, also wie können 106 MB normal sein?
Nun, in dieser Datei habe ich nur reelle Zahlen und NAs. Ich habe keine ganzzahligen Werte darin. Wann werfen Sie die Meldung "Bumping from datatypeA to datatypeB"?
Genau das meine ich. In der aktuellen Version von data.table, die ich installiert habe, lädt es das DT, wenn ich die colClasses weglasse, aber voller NAs.
DT[!is.na(D_19821001),]erzeugt 0 Datensätze und wenn ich die Tabelle mit colClasses lade und die gleiche Filterung durchführe, werden die Datensätze tatsächlich angezeigt.Nun, diese Datei ist 47 GB als csv auf der Festplatte, aber sobald Sie sie in R geladen haben, benötigt sie mehr als das Doppelte an RAM ... Hat nichts mit der Genauigkeit der Werte zu tun, wenn sie einmal geladen ist, ist die Speicherzuweisung wirklich Zahlen verursachen diesen Anstieg?
Vielleicht noch ein Tippfehler: 106MB hätten 106GB sein sollen.
Ich folge nicht dem NA-Aspekt. Aber ein paar Fixes unterwegs und dann nochmal frisch versuchen...
Okay - bitte versuchen Sie es erneut. Die Schätzungsstichprobe hat sich auf 10.000 erhöht (es wird interessant sein zu sehen, wie lange das dauert) und die Puffergrößen haben jetzt ein Minimum.
Möglicherweise müssen Sie mindestens 30 Minuten warten, bis die Drat-Paketdatei hochgestuft wird.
Entschuldigung, es war "GB" statt "MB". Ich hatte nicht genug Kaffee.
Ich hole mir das Update und teste es.
Testen mit der neuesten data.table-Entwicklung: data.table 1.10.5 IN DEVELOPMENT build 2017-03-30 16:31:45 UTC :
Zusammenfassung:
Es arbeitet schnell (1,43 Minuten zum Lesen der CSV) und die Speicherzuweisung funktioniert auch gut, ich denke, die RAM-Zuweisung erhöht sich nicht wie zuvor. Ein 44 GB csv auf der Festplatte bedeutet nach dem Laden ~112 (+ 37 GB Temp. Puffer) GB RAM. Hängt das mit dem Datentyp der Werte in der Datei zusammen?
Prüfung 1 :
Ohne colClasses=list("numeric"=1:12785)
DT<-fread('dt.daily.4km.csv', verbose=TRUE)
Parameter na.strings == <<NA>>
None of the 1 na.strings are numeric (such as '-9999').
Input contains no \n. Taking this to be a filename to open
File opened, filesize is 43.772296 GB.
Memory mapping ... ok
Detected eol as \n only (no \r afterwards), the UNIX and Mac standard.
Positioned on line 1 starting: <<x,y,D_19810101,D_19810102,D_19810103,D_19810104,D_19810105,D_19810106,D_19810107,
D_19810108,D_19810109,D_19810110,D_19810111,D_19810112,
...
All the fields on line 1 are character fields. Treating as the column names.
Number of sampling jump points = 101 because 47000004016 bytes from row 1 to eof / (2 * 1281788 jump0size) == 18333
Type codes (jump 000) : 441111111111111111111111111111111111111111111111111111111111111111111111111111111111111111...1111111111 Quote rule 0
Type codes (jump 001) : 444222422222222224442444444444442222444444444444444444442444444444444222224444444444444444...4444444442 Quote rule 0
Type codes (jump 002) : 444222444444222244442444444444444422444444444444444444442444444444444222224444444444444444...4444444442 Quote rule 0
...
Type codes (jump 034) : 444444444444444444444444444444444444444444444444444444444444444444444444444444444444444444...4444444444 Quote rule 0
Type codes (jump 100) : 444444444444444444444444444444444444444444444444444444444444444444444444444444444444444444...4444444444 Quote rule 0
=====
Sampled 10049 rows (handled \n inside quoted fields) at 101 jump points including middle and very end
Bytes from first data row on line 2 to the end of last row: 47000004016
Line length: mean=79727.22 sd=32260.00 min=12804 max=153029
Estimated nrow: 47000004016 / 79727.22 = 589511
Initial alloc = 1179022 rows (589511 + 100%) using bytes/max(mean-2*sd,min) clamped between [1.1*estn, 2.0*estn]
=====
Type codes (colClasses) : 444444444444444444444444444444444444444444444444444444444444444444444444444444444444444444...4444444444
Type codes (drop|select) : 444444444444444444444444444444444444444444444444444444444444444444444444444444444444444444...4444444444
Allocating 12785 column slots (12785 - 0 dropped)
Reading 432 chunks of 103.756MB (1364 rows) using 144 threads
Read 872505 rows x 12785 columns from 43.772GB file in 02:17.726 wall clock time (can be slowed down by any other open apps even if seemingly idle)
Final type counts
0 : drop
0 : logical
0 : integer
0 : integer64
12785 : double
0 : character
Thread buffers were grown 67 times (if all 144 threads each grew once, this figure would be 144)
=============================
0.000s ( 0%) Memory map
0.099s ( 0%) sep, ncol and header detection
11.057s ( 8%) Column type detection using 10049 sample rows
0.899s ( 1%) Allocation of 872505 rows x 12785 cols (112.309GB) plus 37.433GB of temporary buffers
125.671s ( 91%) Reading data
0.000s ( 0%) Rereading 0 columns due to out-of-sample type exceptions
137.726s Total
Prüfung 2 :
Jetzt mit colClasses=list("numeric"=1:12785)
Das Timing wurde um einige Sekunden verbessert...
DT<-fread('dt.daily.4km.csv', colClasses=list("numeric"=1:12785), verbose=TRUE)
Allocating 12785 column slots (12785 - 0 dropped)
Reading 432 chunks of 103.756MB (1364 rows) using 144 threads
Read 872505 rows x 12785 columns from 43.772GB file in 01:43.028 wall clock time (can be slowed down by any other open apps even if seemingly idle)
Final type counts
0 : drop
0 : logical
0 : integer
0 : integer64
12785 : double
0 : character
Thread buffers were grown 67 times (if all 144 threads each grew once, this figure would be 144)
=============================
0.000s ( 0%) Memory map
0.092s ( 0%) sep, ncol and header detection
11.009s ( 11%) Column type detection using 10049 sample rows
0.332s ( 0%) Allocation of 872505 rows x 12785 cols (112.309GB) plus 37.433GB of temporary buffers
91.595s ( 89%) Reading data
0.000s ( 0%) Rereading 0 columns due to out-of-sample type exceptions
103.028s Total
Ok toll - wir sind dabei. Die korrigierte Stichprobengröße plus Erhöhung auf 100 Zeilen an 100 Punkten (10.000 Stichprobenzeilen) reichte damals aus, um die Typen richtig zu erraten - super. Da es 12.875 Spalten gibt und die Zeilenlänge in der 44-GB-Datei durchschnittlich 80.000 Zeichen beträgt, dauerte die Abtastung 11 Sekunden. Aber diese Zeit hat sich gelohnt, weil es ein erneutes Lesen vermieden hat, das weitere 90er Jahre gedauert hätte. Dann bleiben wir dabei.
Ich denke, das zweite Mal ist schneller, nur weil es das zweite Mal war und Ihr Betriebssystem aufgewärmt und die Datei zwischengespeichert hat. Alles andere, was auf der Box läuft, wirkt sich auf die Zeitangaben der Wanduhr aus. Dies wird durch Ausführen von 3 identischen aufeinanderfolgenden Durchläufen des ersten Tests behoben. Dann nur eine Sache ändern und wieder 3 identische aufeinanderfolgende Läufe laufen lassen. Bei einer Größe von 44 GB werden Sie eine große natürliche Varianz sehen. 3 Läufe reichen normalerweise aus, um eine Schlussfolgerung zu ziehen, aber es kann eine schwarze Kunst sein.
Ja, die 112 GB im Speicher gegenüber 44 GB auf der Festplatte sind zum Teil darauf zurückzuführen, dass die Daten im Speicher größer sind, da alle Spalten vom Typ double sind; es gibt keine speicherinterne Komprimierung in R und es gibt ziemlich viele NA-Werte, die in dieser CSV-Datei keinen Platz beanspruchen (nur ",," ), sondern 8 Byte im Speicher. Es sollte jedoch 83 GB und nicht 112 GB sein (872505 Zeilen x 12785 Spalten x 8 Byte Doubles / 1024^3 = 83 GB). Diese 112 GB wurden basierend auf dem Mittelwert und der Standardabweichung der Leitungslängen zugewiesen. Basierend auf der durchschnittlichen Zeilenlänge schätzte sie 589.511 Zeilen, was zu niedrig gewesen wäre. Die Leitungslängenvarianz war so hoch, dass die Klemmung bei +100% zum Tragen kam. 58.9511 * 2 = 1.179.022 * 12785 * 8 / 1024^3 = 112 GB. Am Ende fand er, dass 872.505 in der Datei ist. Aber es gibt nicht den freien Speicherplatz frei. Ich werde das beheben. (TODO1)
Wenn Sie den Spaltentyp für alle Spalten angegeben haben, wurden jedoch trotzdem Stichproben erstellt. Es sollte die Stichprobenerhebung überspringen, wenn der Benutzer jede Spalte angegeben hat. (TODO2)
Da alle Ihre Daten doppelt sind, werden 11 Milliarden Aufrufe der C-Bibliotheksfunktion strtod() durchgeführt. Die seit langem angestrebte Spezialisierung dieser Funktion sollte diese Datei theoretisch deutlich beschleunigen. (GETAN)
Danke dafür, tolle Erklärung. Lassen Sie es mich wissen, wenn Sie möchten, dass ich etwas anderes mit dieser Datei teste. Ich arbeite an einem anderen Datensatz, der eher im Langformat ist, mit ~ 21 Millionen Zeilen x 1432 Spalten.
NAME NROW NCOL MB
[1,] DT 21,812,625 1,432 238,310
Fügen Sie diesem einen zusätzlichen Datenpunkt hinzu. 89G .tsv , die maximale Speichernutzung während des Ladens beträgt ~180G. Ich denke, dies wird erwartet, da es viele NA und doppelte gibt.
Gerne teste ich das auch.
Ubuntu 16.04 64bit / Linux 4.4.0-71-generic
R version 3.3.2 (2016-10-31)
data.table 1.10.5 IN DEVELOPMENT built 2017-04-04 14:27:46 UTC
Architecture: x86_64
CPU op-mode(s): 32-bit, 64-bit
Byte Order: Little Endian
CPU(s): 64
On-line CPU(s) list: 0-63
Thread(s) per core: 2
Core(s) per socket: 16
Socket(s): 2
NUMA node(s): 2
Vendor ID: GenuineIntel
CPU family: 6
Model: 79
Model name: Intel(R) Xeon(R) CPU E5-2686 v4 @ 2.30GHz
Stepping: 1
CPU MHz: 2699.984
CPU max MHz: 3000.0000
CPU min MHz: 1200.0000
BogoMIPS: 4660.70
Hypervisor vendor: Xen
Virtualization type: full
L1d cache: 32K
L1i cache: 32K
L2 cache: 256K
L3 cache: 46080K
NUMA node0 CPU(s): 0-15,32-47
NUMA node1 CPU(s): 16-31,48-63
Flags: fpu vme de pse tsc msr pae mce cx8 apic sep mtrr pge mca cmov pat pse36 clflush mmx fxsr sse sse2 ht syscall nx pdpe1gb rdtscp lm constant_tsc rep_good nopl xtopology nonstop_tsc aperfmperf eagerfpu pni pclmulqdq monitor est ssse3 fma cx16 pcid sse4_1 sse4_2 x2apic movbe popcnt tsc_deadline_timer aes xsave avx f16c rdrand hypervisor lahf_lm abm fsgsbase bmi1 hle avx2 smep bmi2 erms invpcid rtm xsaveopt ida
Parameter na.strings == <<NA>>
None of the 1 na.strings are numeric (such as '-9999').
Input contains no \n. Taking this to be a filename to open
File opened, filesize is 88.603947 GB.
Memory mapping ... ok
Detected eol as \n only (no \r afterwards), the UNIX and Mac standard.
Positioned on line 1 starting: <<allele prediction_uuid sample_>>
Detecting sep ...
sep=='\t' with 101 lines of 76 fields using quote rule 0
Detected 76 columns on line 1. This line is either column names or first data row (first 30 chars): <<allele prediction_uuid sample_>>
All the fields on line 1 are character fields. Treating as the column names.
Number of sampling jump points = 101 because 95137762779 bytes from row 1 to eof / (2 * 24414 jump0size) == 1948426
Type codes (jump 000) : 5555542444111145424441111444111111111111111111111111111111111111111111111111 Quote rule 0
Type codes (jump 009) : 5555542444114445424441144444111111111111111111111111111111111111111111111111 Quote rule 0
Type codes (jump 042) : 5555542444444445424444444444111111111111111111111111111111111111111111111111 Quote rule 0
Type codes (jump 048) : 5555544444444445444444444444225225522555545111111111111111111111111111111111 Quote rule 0
Type codes (jump 083) : 5555544444444445444444444444225225522555545254452454411154454452454411154455 Quote rule 0
Type codes (jump 085) : 5555544444444445444444444444225225522555545254452454454454454452454454454455 Quote rule 0
Type codes (jump 100) : 5555544444444445444444444444225225522555545254452454454454454452454454454455 Quote rule 0
=====
Sampled 10028 rows (handled \n inside quoted fields) at 101 jump points including middle and very end
Bytes from first data row on line 2 to the end of last row: 95137762779
Line length: mean=465.06 sd=250.27 min=198 max=929
Estimated nrow: 95137762779 / 465.06 = 204571280
Initial alloc = 409142560 rows (204571280 + 100%) using bytes/max(mean-2*sd,min) clamped between [1.1*estn, 2.0*estn]
=====
Type codes (colClasses) : 5555544444444445444444444444225225522555545254452454454454454452454454454455
Type codes (drop|select) : 5555544444444445444444444444225225522555545254452454454454454452454454454455
Allocating 76 column slots (76 - 0 dropped)
Reading 90752 chunks of 1.000MB (2254 rows) using 64 threads
Wenn es hilft, hier sind die Ergebnisse einer sehr langen Datenbank: 419.124.196 x 42 (~2^34) mit einer Kopfzeile und übergebenen colClasses.
> library(data.table)
data.table 1.10.5 IN DEVELOPMENT built 2017-09-27 17:12:56 UTC; travis
The fastest way to learn (by data.table authors): https://www.datacamp.com/courses/data-analysis-the-data-table-way
Documentation: ?data.table, example(data.table) and browseVignettes("data.table")
Release notes, videos and slides: http://r-datatable.com
> CC <- c(rep('integer', 2), rep('character', 3),
+ rep('numeric', 2), rep('integer', 3),
+ rep('character', 2), 'integer', 'character', 'integer',
+ rep('character', 4), rep('numeric', 11), 'character',
+ 'numeric', 'character', rep('numeric', 2),
+ rep('integer', 3), rep('numeric', 2), 'integer',
+ 'numeric')
> P <- fread('XXXX.csv', colClasses = CC, header = TRUE, verbose = TRUE)
Input contains no \n. Taking this to be a filename to open
[01] Check arguments
Using 40 threads (omp_get_max_threads()=40, nth=40)
NAstrings = [<<NA>>]
None of the NAstrings look like numbers.
show progress = 1
0/1 column will be read as boolean
[02] Opening the file
Opening file XXXXcsv
File opened, size = 51.71GB (55521868868 bytes).
Memory mapping ... ok
[03] Detect and skip BOM
[04] Arrange mmap to be \0 terminated
\r-only line endings are not allowed because \n is found in the data
[05] Skipping initial rows if needed
Positioned on line 1 starting: <<X,X,X,X>>
[06] Detect separator, quoting rule, and ncolumns
Detecting sep ...
sep=',' with 100 lines of 42 fields using quote rule 0
Detected 42 columns on line 1. This line is either column names or first data row. Line starts as: <<X,X,X,X>>
Quote rule picked = 0
fill=false and the most number of columns found is 42
[07] Detect column types, good nrow estimate and whether first row is column names
'header' changed by user from 'auto' to true
Number of sampling jump points = 101 because (55521868866 bytes from row 1 to eof) / (2 * 13006 jump0size) == 2134471
Type codes (jump 000) : 5161010775551055105101111111111111110110771117717 Quote rule 0
Type codes (jump 022) : 5561010775551055105101111111111111110110771117717 Quote rule 0
Type codes (jump 030) : 5561010775551055105101010107517171151110110771117717 Quote rule 0
Type codes (jump 037) : 5561010775551055105101010107517771171110110771117717 Quote rule 0
Type codes (jump 073) : 5561010775551055105101010107517771177110110771117717 Quote rule 0
Type codes (jump 093) : 5561010775551055105101010107717771177110110771117717 Quote rule 0
Type codes (jump 100) : 5561010775551055105101010107717771177110110771117717 Quote rule 0
=====
Sampled 10049 rows (handled \n inside quoted fields) at 101 jump points
Bytes from first data row on line 1 to the end of last row: 55521868866
Line length: mean=132.68 sd=6.00 min=118 max=425
Estimated number of rows: 55521868866 / 132.68 = 418453923
Initial alloc = 460299315 rows (418453923 + 9%) using bytes/max(mean-2*sd,min) clamped between [1.1*estn, 2.0*estn]
=====
[08] Assign column names
[09] Apply user overrides on column types
After 11 type and 0 drop user overrides : 551010107755510105105101010107777777777710710775557757
[10] Allocate memory for the datatable
Allocating 42 column slots (42 - 0 dropped) with 460299315 rows
[11] Read the data
jumps=[0..52960), chunk_size=1048373, total_size=55521868441
Read 98%. ETA 00:00
[12] Finalizing the datatable
Read 419124195 rows x 42 columns from 51.71GB (55521868868 bytes) file in 13:42.935 wall clock time
Thread buffers were grown 0 times (if all 40 threads each grew once, this figure would be 40)
Final type counts
0 : drop
0 : bool8
0 : bool8
0 : bool8
0 : bool8
11 : int32
0 : int64
19 : float64
0 : float64
0 : float64
12 : string
=============================
0.000s ( 0%) Memory map 51.709GB file
0.016s ( 0%) sep=',' ncol=42 and header detection
0.016s ( 0%) Column type detection using 10049 sample rows
188.153s ( 23%) Allocation of 419124195 rows x 42 cols (125.177GB)
634.751s ( 77%) Reading 52960 chunks of 1.000MB (7901 rows) using 40 threads
= 0.121s ( 0%) Finding first non-embedded \n after each jump
+ 17.036s ( 2%) Parse to row-major thread buffers
+ 616.184s ( 75%) Transpose
+ 1.410s ( 0%) Waiting
0.000s ( 0%) Rereading 0 columns due to out-of-sample type exceptions
822.935s Total
> memory.size()
[1] 134270.3
> rm(P)
> gc()
used (Mb) gc trigger (Mb) max used (Mb)
Ncells 585532 31.3 5489235 293.2 6461124 345.1
Vcells 1508139082 11506.2 20046000758 152938.9 25028331901 190951.1
> memory.size()
[1] 87.56
> P <- fread('XXXX.csv', colClasses = CC, header = TRUE, verbose = TRUE)
Input contains no \n. Taking this to be a filename to open
[01] Check arguments
Using 40 threads (omp_get_max_threads()=40, nth=40)
NAstrings = [<<NA>>]
None of the NAstrings look like numbers.
show progress = 1
0/1 column will be read as boolean
[02] Opening the file
Opening file XXXX.csv
File opened, size = 51.71GB (55521868868 bytes).
Memory mapping ... ok
[03] Detect and skip BOM
[04] Arrange mmap to be \0 terminated
\r-only line endings are not allowed because \n is found in the data
[05] Skipping initial rows if needed
Positioned on line 1 starting: <<X,X,X,X>>
[06] Detect separator, quoting rule, and ncolumns
Detecting sep ...
sep=',' with 100 lines of 42 fields using quote rule 0
Detected 42 columns on line 1. This line is either column names or first data row. Line starts as: <<X,X,X,X>>
Quote rule picked = 0
fill=false and the most number of columns found is 42
[07] Detect column types, good nrow estimate and whether first row is column names
'header' changed by user from 'auto' to true
Number of sampling jump points = 101 because (55521868866 bytes from row 1 to eof) / (2 * 13006 jump0size) == 2134471
Type codes (jump 000) : 5161010775551055105101111111111111110110771117717 Quote rule 0
Type codes (jump 022) : 5561010775551055105101111111111111110110771117717 Quote rule 0
Type codes (jump 030) : 5561010775551055105101010107517171151110110771117717 Quote rule 0
Type codes (jump 037) : 5561010775551055105101010107517771171110110771117717 Quote rule 0
Type codes (jump 073) : 5561010775551055105101010107517771177110110771117717 Quote rule 0
Type codes (jump 093) : 5561010775551055105101010107717771177110110771117717 Quote rule 0
Type codes (jump 100) : 5561010775551055105101010107717771177110110771117717 Quote rule 0
=====
Sampled 10049 rows (handled \n inside quoted fields) at 101 jump points
Bytes from first data row on line 1 to the end of last row: 55521868866
Line length: mean=132.68 sd=6.00 min=118 max=425
Estimated number of rows: 55521868866 / 132.68 = 418453923
Initial alloc = 460299315 rows (418453923 + 9%) using bytes/max(mean-2*sd,min) clamped between [1.1*estn, 2.0*estn]
=====
[08] Assign column names
[09] Apply user overrides on column types
After 11 type and 0 drop user overrides : 551010107755510105105101010107777777777710710775557757
[10] Allocate memory for the datatable
Allocating 42 column slots (42 - 0 dropped) with 460299315 rows
[11] Read the data
jumps=[0..52960), chunk_size=1048373, total_size=55521868441
Read 98%. ETA 00:00
[12] Finalizing the datatable
Read 419124195 rows x 42 columns from 51.71GB (55521868868 bytes) file in 05:04.910 wall clock time
Thread buffers were grown 0 times (if all 40 threads each grew once, this figure would be 40)
Final type counts
0 : drop
0 : bool8
0 : bool8
0 : bool8
0 : bool8
11 : int32
0 : int64
19 : float64
0 : float64
0 : float64
12 : string
=============================
0.000s ( 0%) Memory map 51.709GB file
0.031s ( 0%) sep=',' ncol=42 and header detection
0.000s ( 0%) Column type detection using 10049 sample rows
28.437s ( 9%) Allocation of 419124195 rows x 42 cols (125.177GB)
276.442s ( 91%) Reading 52960 chunks of 1.000MB (7901 rows) using 40 threads
= 0.017s ( 0%) Finding first non-embedded \n after each jump
+ 12.941s ( 4%) Parse to row-major thread buffers
+ 262.989s ( 86%) Transpose
+ 0.495s ( 0%) Waiting
0.000s ( 0%) Rereading 0 columns due to out-of-sample type exceptions
304.910s Total
> memory.size()
[1] 157049.7
> sessionInfo()
R version 3.4.2 beta (2017-09-17 r73296)
Platform: x86_64-w64-mingw32/x64 (64-bit)
Running under: Windows Server >= 2012 x64 (build 9200)
Matrix products: default
locale:
[1] LC_COLLATE=English_United States.1252 LC_CTYPE=English_United States.1252
[3] LC_MONETARY=English_United States.1252 LC_NUMERIC=C
[5] LC_TIME=English_United States.1252
attached base packages:
[1] stats graphics grDevices utils datasets methods base
other attached packages:
[1] data.table_1.10.5
loaded via a namespace (and not attached):
[1] compiler_3.4.2 tools_3.4.2
Ein paar Anmerkungen. Ich hätte vorgeschlagen, [09] vor [07] zu setzen, damit es keinen Grund zum Überprüfen gibt, wenn colClasses übergeben werden. Außerdem zeigte Windows nach jedem Durchlauf etwa 160 GB an. memory.size() führt wahrscheinlich eine Reinigung durch. Bei 532 GB RAM auf diesem Server kann das Speichercaching mit der Geschwindigkeitssteigerung beim zweiten Durchlauf zu tun haben. Ich hoffe, das hilft.
Test an einer breiten "realen" Tabelle (Krankenhausdaten): 30 Mio. Zeilen × 125 Spalten v readr '1.2.0' und read.csv 3.4.3.
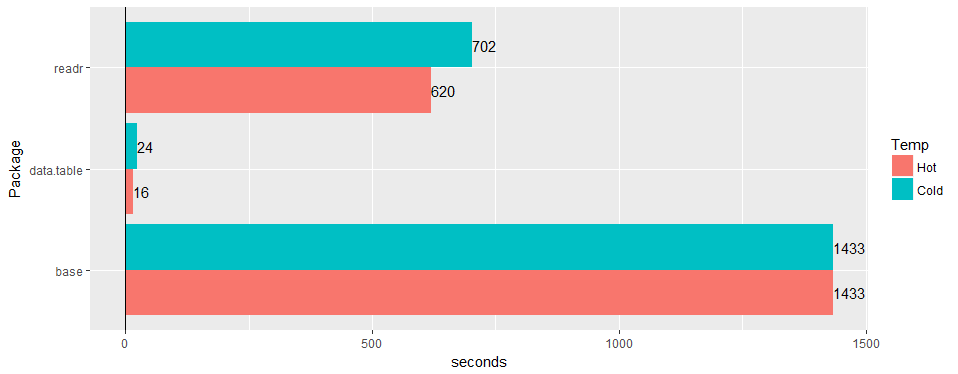
Gibt es eine Chance zu bestätigen, dass das Problem am 1.11.4 noch gültig ist? oder Code, um Beispieldaten zu erzeugen.
Das ist soweit ich weiß alles gelöst. @geponce bitte aktualisieren, wenn nicht.
TODO1 oben jetzt als #3024 eingereicht
TODO2 oben jetzt als #3025 eingereicht
Hilfreichster Kommentar
Test an einer breiten "realen" Tabelle (Krankenhausdaten): 30 Mio. Zeilen × 125 Spalten v readr '1.2.0' und
read.csv3.4.3.