Data.table: fread avec un grand csv (44 Go) prend beaucoup de RAM dans la dernière version de data.table dev
Salut,
Matériel et logiciel:
Serveur : Dell R930 4-Intel Xeon E7-8870 v3 2.1GHz,45M Cache,9.6GT/s QPI,Turbo,HT,18C/36T et 1To en RAM
OS : Redhat 7.1
Version R : 3.3.2
version data.table : 1.10.5 construit 2017-03-21
Je charge un fichier csv (44 Go, 872505 lignes x 12785 cols). Il se charge très rapidement, en 1,30 minutes en utilisant 144 cœurs (72 cœurs des 4 processeurs avec l'hyperthreading activé pour en faire une boîte à 144 cœurs).
Le problème principal est que lorsque le DT est chargé, la quantité de mémoire utilisée augmente considérablement par rapport à la taille du fichier csv. Dans ce cas, le csv de 44 Go (enregistré avec fwrite, enregistré avec saveRDS et compress=FALSE crée un fichier de 84 Go) utilise ~ 356 Go de RAM.
Voici le résultat en utilisant "verbose=TRUE"
_Allocation de 12785 emplacements de colonne (12785 - 0 abandonné)
madvise séquentiel : ok
Lecture de données avec 1440 points de saut et 144 threads
Lire 95,7% des 858881 lignes estimées
Lire 872505 lignes x 12785 colonnes à partir d'un fichier de 43,772 Go en 1 minute 33,736 secondes de temps d'horloge murale (affecté par d'autres applications en cours d'exécution)
0.000s ( 0%) Carte mémoire
0.070s ( 0%) sep, ncol et détection d'en-tête
26,227s (28 %) Détection du type de colonne à l'aide de 34832 lignes d'échantillons à partir de 1440 points de saut
0,614s ( 1%) Allocation de 3683116 lignes x 12785 cols (350,838 Go) en RAM
0.000s ( 0%) madvise séquentiel
66,825s (71%) Lecture des données
93.736s au total_
Il montre un problème similaire qui se pose parfois lors de l'utilisation du package parallèle, où une session est lancée par cœur lors de l'utilisation de fonctions telles que "mclapply". Voir les Rsessions créées/répertoriées dans cette capture d'écran :

si je fais "rm(DT)", la RAM revient à l'état initial et les "Rsessions" sont supprimées.
Déjà essayé, par exemple "setDTthreads(20)" et utilisant toujours la même quantité de RAM.
Soit dit en passant, si le fichier est chargé avec la version non parallèle de "fread", l'allocation de mémoire n'atteint que ~ 106 Go.
Guillermo
Tous les 30 commentaires
Ceci est le résultat de l'implémentation de fread non parallèle (data.table 1.10.5 IN DEVELOPMENT construit 2017-02-09)

Et j'ai vérifié deux fois la quantité de mémoire utilisée et elle monte à 84 Go.
Guillermo
Oui tu as raison. Merci pour le super reportage. Le nombre estimé semble à peu près correct (858 881 contre 872 505) mais l'allocation est alors 4,2 fois plus importante que cela (3 683 116) et bien loin. J'ai amélioré le calcul et ajouté plus de détails à la sortie détaillée. Attendez de retester pour l'instant jusqu'à ce que quelques autres choses aient été faites.
Ok s'il vous plaît retestez - devrait être corrigé maintenant.
Je viens d'installer data.table dev :
data.table 1.10.5 EN DÉVELOPPEMENT construit 2017-03-27 02:50:31 UTC
La première chose que j'ai reçue lorsque j'ai essayé de lire le même fichier de 44 Go était ce message :
DT <- fread('dt.daily.4km.csv')
Erreur : protect() : débordement de la pile de protection
Ensuite, je viens de réexécuter la même commande et j'ai commencé à bien fonctionner. Cependant, cette version n'utilise pas de mode multicœur. Le chargement prend environ 25 minutes, comme avant de mettre la version fread-parallel.
Guillermo
J'ai fermé toutes les r-sessions et ré-exécuté un test et j'obtiens une erreur :
Le type de colonne deviné était insuffisant pour 34711745 valeurs dans 508 colonnes. Utilisez colClasses pour définir ces classes de colonnes manuellement.
Voir ci-dessous, je reçois plusieurs messages concernant "entier deviné mais contient <<0....>>
_Lire 872505 lignes x 12785 colonnes à partir d'un fichier de 43,772 Go en 15:27.024 heure d'horloge murale (peut être ralenti par toute autre application ouverte même si apparemment inactive)
La colonne 171 ('D_19810618') a deviné 'entier' mais contient <<2.23000001907349>>
La colonne 347 ('D_19811211') a deviné 'entier' mais contient <<1.02999997138977>>
La colonne 348 ('D_19811212') a deviné 'entier' mais contient <<3.75>>_
Wow - votre fichier teste vraiment les cas extrêmes. Super. À l'avenir, veuillez exécuter avec verbose=TRUE et fournir la sortie complète. Mais avec les informations que vous avez fournies dans ce cas, je peux voir quel est le problème en réalité. Il y a un tampon créé pour chaque colonne pour chaque thread (dans ce cas, plus de 12.000 colonnes). Chacun est actuellement PROTÉGÉ séparément. Il existe un moyen d'éviter cela - fera l'affaire. Les messages sur le type devinant sont corrects. Ces 508 colonnes signifient-elles quelque chose pour vous et vous acceptez qu'elles soient numériques ? Vous pouvez transmettre une plage de colonnes à colClasses comme ceci : colClasses=list("numeric"=11:518)
De quel domaine proviennent ces données ? Créez-vous le fichier ? On a l'impression qu'il a été tordu au format large où la meilleure pratique normale est d'écrire (et de le garder en mémoire aussi) au format long. Je m'attendrais normalement à voir les 508 noms de colonnes tels que "D_19810618" en tant que valeurs dans une colonne, et non en tant que colonnes elles-mêmes. C'est pourquoi je vous demande si vous créez le fichier et pouvez-vous le créer au format long. Si ce n'est pas le cas, suggérez à celui qui crée le fichier qu'il peut le faire mieux. Je suppose que vous appliquez des opérations via des colonnes en utilisant peut-être .SD et .SDcols . C'est vraiment bien mieux en format long et keyby= la colonne contenant les valeurs comme "D_19810618".
Mais je vais quand même essayer de rendre fread aussi bon que possible en traitant n'importe quelle entrée - même des fichiers très volumineux de plus de 12 000 colonnes.
J'espère que d'autres personnes testent et ne trouvent aucun problème sur leurs fichiers !
_ Ces 508 colonnes vous disent-elles quelque chose et vous acceptez qu'elles soient numériques ?
Ce tableau contient des séries chronologiques par ligne. IDx,IDy, Time1_value, Time2_value, Time3_value... et toutes les colonnes Time N _value ne contiennent que des valeurs numériques. Si j'utilise colClasses, je vais devoir le faire pour 12783 : list("numeric"=2:12783). Je vais essayer ça.
_De quel champ proviennent ces données ?_
Données géospatiales. Je fais des recherches à distance euclidienne dans DT en utilisant IDx et IDy. Je suppose que plus il y a de lignes, plus les recherches sont lentes, n'est-ce pas ?
En ce moment, c'est assez rapide (format large). J'ai une carte, où un utilisateur peut cliquer sur une zone, puis une série temporelle est générée dans un fichier csv à partir de l'emplacement le plus proche (avec des données disponibles) jusqu'au clic donné. Je vais l'implémenter avec un format long à la place.
Je reviendrai avec quelques résultats.
OK bien. Vous n'avez pas besoin de list("numeric"=2:12783) iiuc car il n'a besoin d'aide que pour 508 colonnes. Oh - je vois - les 508 sont dispersés dans les colonnes, je suppose alors (ce ne sont pas un ensemble de colonnes contigu) ?
Non - data.table n'est presque jamais plus rapide lorsqu'il est large ! Long est presque toujours plus rapide et plus pratique. Avez-vous vu et essayé roll="nearest" ? Comment fais-tu maintenant? Veuillez montrer le code afin que nous puissions comprendre. Le format long est presque certainement meilleur, mais nous aurons peut-être besoin de quelques améliorations pour la 2D la plus proche. Veuillez également indiquer les horaires. Lorsque vous dites "assez vite", il s'avère que les gens ont des idées très différentes sur ce qu'est "assez vite".
faire fondre cette table atteint la limite 2^31. J'obtiens l'erreur : "les vecteurs de longueur négative ne sont pas autorisés".
Je vais revenir à la source pour voir si je peux le générer au format long.
# Read Data
DT <- fread('dt.daily.4km.csv', showProgress = FALSE)
# Add two columns with truncated values of x and y (these are geog. coords.)
DT[,y_tr:=trunc(y)]
DT[,x_tr:=trunc(x)]
# For using on plotting (x-axis values)
xaxis<-seq.Date(as.Date("1981-01-01"),as.Date("2015-12-31"), "day")
# subset by truncated coordinates to avoid full-table search. Now searches
# will happen in a smaller subset
DT2 <- DT[y_tr==trunc(y_clicked) & x_tr==trunc(x_clicked),]
# Add distance from each point in the data.table to the provided location, "gdist" is from
# Imap package for euclidean distance.
DT2[,DIST:=gdist(lat.1 = DT2$y,
lon.1 = DT2$x,
lat.2 = y_clicked,
lon.2 = x_clicked, units="miles")]
# Get the minimum distance
minDist <- min(DT2[,DIST])
# Get the y-axis values
yt <- transpose(DT2[DIST==minDist,3:(ncol(DT2)-3)])$V1`
# Ready to plot xaxis vs yt
...
...
Je n'ai pas l'application sur un serveur public. Essentiellement, l'utilisateur peut cliquer sur une carte, puis je capture ces coordonnées et effectue la recherche ci-dessus, obtient la série temporelle et crée un tracé.
Trouvé et corrigé un autre débordement de pile pour un très grand nombre de colonnes : https://github.com/Rdatatable/data.table/commit/d0469e670961dcdea115d433c0f2dce596d65906. J'ai oublié de marquer ce numéro de problème dans le message de validation.
Oh. C'est un point. 872505 rangs * 12780 cols soit 11 milliards de rangs. Donc, ma suggestion d'utiliser un format long ne fonctionnera pas pour vous car c'est > 2^31. Désolé, j'aurais dû le remarquer. Nous devrons juste mordre la balle et aller > 2^31 alors. En attendant, restons-en au format large que vous utilisez et je vais le résoudre.
Veuillez réessayer. L'utilisation de la mémoire devrait revenir à la normale et il devrait relire automatiquement les 508 colonnes sur 12 785 avec des exceptions de type hors échantillon. Pour éviter le délai de réexécution automatique, vous pouvez définir colClasses .
Si ce n'est pas corrigé, veuillez coller la sortie détaillée complète. Doigts croisés!
D'accord...
Un résumé des résultats avec les derniers data.table dev : data.table 1.10.5 EN DÉVELOPPEMENT construit 2017-03-29 16:17:01 UTC
Quatre points principaux à mentionner :
- fread a bien fonctionné en lisant le fichier dans la discussion.
- La lecture a pris environ 5,5 minutes par rapport à la version précédente qui prenait environ 1,3 minutes.
- Cette version n'augmente pas l'allocation de RAM comme la version précédente que je testais.
- Les cœurs semblent être "moins actifs" (voir capture d'écran ci-dessous).
Quelques commentaires/questions :
1.
Le pourcentage d'utilisation dans les cœurs n'est pas aussi actif qu'avant, voir capture d'écran :
Dans la version précédente de fread, l'activité des cœurs était toujours d'environ 90 à 80 %. Dans cette version, il restait environ 2 à 3 % de chaque cœur, comme indiqué dans l'image ci-dessus.
Je ne comprends pas pourquoi fread fait des sauts d'entier à numérique: par exemple
Column 1489 ("D_19850126") bumped from 'integer' to 'numeric' due to <<0.949999988079071>> somewhere between row 6041 and row 24473
J'ai vérifié ces lignes et cela semble ok. Pourquoi est-il détecté comme un nombre entier et doit-il être « remplacé » par « numérique » s'il est déjà numérique (voir le résumé pour les lignes suggérées ci-dessous) ? ou est-ce que je comprends mal cette ligne? Cela se produit pour 508 lignes. Est-ce que les NA causent des problèmes ?
summary(DT[6041:24473,.(D_19850126)])
D_19850126
Min. :0.750
1st Qu.:0.887
Median :0.945
Mean :0.966
3rd Qu.:1.045
Max. :1.210
NA's :18393
Ci-dessous un peu verbeux du test.
DT<-fread('dt.daily.4km.ver032917.csv', verbose=TRUE)
SORTIR
Parameter na.strings == <<NA>>
None of the 0 na.strings are numeric (such as '-9999').Input contains no \n. Taking this to be a filename to open
File opened, filesize is 43.772296 GB.
Memory mapping ... ok
Detected eol as \n only (no \r afterwards), the UNIX and Mac standard.
Positioned on line 1 starting: <<x,y,D_19810101,D_19810102,D_19810103,D_19810104,D_19810105,D_19810106,D_19810107,D_19810108,D_19810109,D_19810110,D_19810111,D_19810112,D_19810113,
...
...
All the fields on line 1 are character fields. Treating as the column names.
Number of sampling jump points = 11 because 1281788 startSize * 10 NJUMPS * 2 = 25635760 <= -244636240 bytes from line 2 to eof
Type codes (jump 00) : 441111111111111111111111111111111111111111111111111111111111111111111111111111111111111111...1111111111 Quote rule 0
Type codes (jump 01) : 444422222222222222242444424444442222222222424444424442224424222244222222222242422222224422...4442244422 Quote rule 0
Type codes (jump 02) : 444422222222222222242444424444442422222242424444424442224424222244222222222242424222424424...4442444442 Quote rule 0
Type codes (jump 03) : 444422222222222224242444424444444422222244424444424442224424222244222222222244424442424424...4442444442 Quote rule 0
Type codes (jump 04) : 444444244422222224442444424444444444242244444444444444424444442244444222222244424442444444...4444444444 Quote rule 0
Type codes (jump 05) : 444444244422222224442444424444444444242244444444444444424444442244444222222244424442444444...4444444444 Quote rule 0
Type codes (jump 06) : 444444444422222224442444424444444444242244444444444444424444444444444244444444424442444444...4444444444 Quote rule 0
Type codes (jump 07) : 444444444422222224442444424444444444242244444444444444444444444444444244444444424444444444...4444444444 Quote rule 0
Type codes (jump 08) : 444444444442222224444444424444444444242244444444444444444444444444444444444444424444444444...4444444444 Quote rule 0
Type codes (jump 09) : 444444444442222424444444424444444444244444444444444444444444444444444444444444424444444444...4444444444 Quote rule 0
Type codes (jump 10) : 444444444442222424444444424444444444244444444444444444444444444444444444444444424444444444...4444444444 Quote rule 0
=====
Sampled 305 rows (handled \n inside quoted fields) at 11 jump points including middle and very end
Bytes from first data row on line 2 to the end of last row: 47000004016
Line length: mean=45578.20 sd=33428.37 min=12815 max=108497
Estimated nrow: 47000004016 / 45578.20 = 1031195
Initial alloc = 2062390 rows (1031195 + 100%) using bytes/max(mean-2*sd,min) clamped between [1.1*estn, 2.0*estn]
=====
Type codes (colClasses) : 444444444442222424444444424444444444244444444444444444444444444444444444444444424444444444...4444444444
Type codes (drop|select): 444444444442222424444444424444444444244444444444444444444444444444444444444444424444444444...4444444444
Allocating 12785 column slots (12785 - 0 dropped)
Reading 44928 chunks of 0.998MB (22 rows) using 144 threads
Read 872505 rows x 12785 columns from 43.772GB file in 05:26.908 wall clock time (can be slowed down by any other open apps even if seemingly idle)
Final type counts
0 : drop
0 : logical
0 : integer
0 : integer64
12785 : numeric
0 : character
Rereading 508 columns due to out-of-sample type exceptions.
Column 171 ("D_19810618") bumped from 'integer' to 'numeric' due to <<2.23000001907349>> somewhere between row 6041 and row 24473
Column 347 ("D_19811211") bumped from 'integer' to 'numeric' due to <<1.02999997138977>> somewhere between row 6041 and row 24473
Column 348 ("D_19811212") bumped from 'integer' to 'numeric' due to <<3.75>> somewhere between row 6041 and row 24473
Column 643 ("D_19821003") bumped from 'integer' to 'numeric' due to <<1.04999995231628>> somewhere between row 6041 and row 24473
Column 1066 ("D_19831130") bumped from 'integer' to 'numeric' due to <<1.46000003814697>> somewhere between row 6041 and row 24473
Column 1102 ("D_19840105") bumped from 'integer' to 'numeric' due to <<0.959999978542328>> somewhere between row 6041 and row 24473
Column 1124 ("D_19840127") bumped from 'integer' to 'numeric' due to <<0.620000004768372>> somewhere between row 6041 and row 24473
Column 1130 ("D_19840202") bumped from 'integer' to 'numeric' due to <<0.540000021457672>> somewhere between row 6041 and row 24473
Column 1489 ("D_19850126") bumped from 'integer' to 'numeric' due to <<0.949999988079071>> somewhere between row 6041 and row 24473
Column 1508 ("D_19850214") bumped from 'integer' to 'numeric' due to <<0.360000014305115>> somewhere between row 6041 and row 24473
...
...
Reread 872505 rows x 508 columns in 05:29.167
Read 872505 rows. Exactly what was estimated and allocated up front
Thread buffers were grown 0 times (if all 144 threads each grew once, this figure would be 144)
=============================
0.000s ( 0%) Memory map
0.093s ( 0%) sep, ncol and header detection
0.186s ( 0%) Column type detection using 305 sample rows from 44928 jump points
0.600s ( 0%) Allocation of 872505 rows x 12785 cols (192.552GB) plus 1.721GB of temporary buffers
326.029s ( 50%) Reading data
329.167s ( 50%) Rereading 508 columns due to out-of-sample type exceptions
656.075s Total
Ce dernier résumé a pris beaucoup de temps pour le terminer (~6 minutes). Si nous comptons ce résumé verbeux, l'ensemble de la lecture a pris environ 11 minutes. Il relit ces 508 colonnes, je peux même voir le message "Relecture de 508 colonnes en raison d'exceptions de type hors échantillon" sans utiliser le verbeux=TRUE.
Vérifiez-le sans "verbose=TRUE"
ptm<-proc.time()
DT<-fread('dt.daily.4km.ver032917.csv')
Read 872505 rows x 12785 columns from 43.772GB file in 05:26.647 wall clock time (can be slowed down by any other open apps even if seemingly idle)
Rereading 508 columns due to out-of-sample type exceptions.
Reread 872505 rows x 508 columns in 05:30.276
proc.time() - ptm
user system elapsed
2113.100 85.919 657.870
test d'écriture
Cela fonctionne bien. Très rapide. Je pensais qu'écrire était plus lent que lire de nos jours.
fwrite(DT,'dt.daily.4km.ver032917.csv', verbose=TRUE)
No list columns are present. Setting sep2='' otherwise quote='auto' would quote fields containing sep2.
maxLineLen=151187 from sample. Found in 1.890s
Writing column names ... done in 0.000s
Writing 872505 rows in 32315 batches of 27 rows (each buffer size 8MB, showProgress=1, nth=144) ...
done (actual nth=144, anyBufferGrown=no, maxBuffUsed=46%)
La "Relecture" prend pas mal de temps. C'est comme lire le fichier deux fois.
Excellent! Merci pour toutes ces informations.
- Oui tu as raison, il ne faut pas relire. Vous avez réussi
colClasses=list("numeric"=1:12785)donc la ligne de sortie commençant parType code (colClasses)devrait avoir la valeur 4. Corrigera et ajoutera le test manquant. - Sur 5,5m contre 1,3m, c'est intéressant. La mémoire tampon de chaque thread est actuellement de 1 Mo. L'idée étant d'être petit pour tenir dans le cache. Mais dans votre cas 1 Mo / 12785 cols = 82 octets. Il est donc 50x trop inefficace en cache avec ce choix. Je suppose, en tout cas. Je n'y aurais jamais pensé sans tes tests. Lorsqu'il fonctionnait à une vitesse de 1,3 m, il n'avait pas cette taille de 1 Mo.
- Pourquoi il n'a échantillonné que 305 lignes est également très étrange. Il aurait dû en échantillonner 1 000. Si cela est corrigé, il détectera peut-être les 508 colonnes sans que vous ayez à le faire. L'échantillon ne prend pas de temps (0,186 s), la taille de l'échantillon peut donc être augmentée.
Pouvez-vous coller la sortie de la commande unix lscpu s'il vous plaît. Cela nous indiquera la taille de votre cache et je pourrai réfléchir à partir de là. Je vais fournir buffMB comme paramètre à fread afin que vous puissiez voir si c'est ça. Je peux faire un meilleur calcul si c'est le cas.
Mon erreur sur un point de mon post précédent. Le colClasses=list("numeric"=1:12785) fonctionne bien. Si je ne spécifie pas "colClasses", il effectue la "Relecture". Désolé pour la confusion.
Une chose que j'ai remarquée est que si je ne spécifie pas les "colClasses", la table est créée avec des NA et la RAM apparaît comme si le DT était chargé normalement (~ 106 Mo en RAM).
Voici la sortie lscpu :
Architecture: x86_64
CPU op-mode(s): 32-bit, 64-bit
Byte Order: Little Endian
CPU(s): 144
On-line CPU(s) list: 0-143
Thread(s) per core: 2
Core(s) per socket: 18
Socket(s): 4
NUMA node(s): 4
Vendor ID: GenuineIntel
CPU family: 6
Model: 63
Model name: Intel(R) Xeon(R) CPU E7-8870 v3 @ 2.10GHz
Stepping: 4
CPU MHz: 2898.328
BogoMIPS: 4195.66
Virtualization: VT-x
L1d cache: 32K
L1i cache: 32K
L2 cache: 256K
L3 cache: 46080K
NUMA node0 CPU(s): 0,4,8,12,16,20,24,28,32,36,40,44,48,52,56,60,64,68,72,76,80,84,88,92,96,100,104,108,112,116,120,124,128,132,136,140
NUMA node1 CPU(s): 1,5,9,13,17,21,25,29,33,37,41,45,49,53,57,61,65,69,73,77,81,85,89,93,97,101,105,109,113,117,121,125,129,133,137,141
NUMA node2 CPU(s): 2,6,10,14,18,22,26,30,34,38,42,46,50,54,58,62,66,70,74,78,82,86,90,94,98,102,106,110,114,118,122,126,130,134,138,142
NUMA node3 CPU(s): 3,7,11,15,19,23,27,31,35,39,43,47,51,55,59,63,67,71,75,79,83,87,91,95,99,103,107,111,115,119,123,127,131,135,139,143
Ok, j'ai compris. Merci.
En relisant vos premiers commentaires, cela aurait-il plus de sens s'il disait « passer de l'entier au double » plutôt que « passer de l'entier au numérique » ?
Qu'entendez-vous par « créé avec les NA » ? Toute la table est pleine de NA, juste les 508 colonnes ?
Qu'est-ce que 'DT a été chargé normalement (~106 Mo de RAM)'. C'est un fichier de 44 Go, alors comment 106 Mo peut-il être normal ?
Eh bien, dans ce fichier, je n'ai que des nombres réels et des NA. Je n'ai pas de valeurs entières dedans. Quand lancez-vous ce message, "passer du type de données A au type de données B" ?
C'est exactement ce que je veux dire. Dans la version actuelle de data.table que j'ai installée, si j'omet les colClasses, il charge le DT mais plein de NA.
DT[!is.na(D_19821001),]produit 0 enregistrement et si je charge la table avec colClasses et fais le même filtrage, il affiche en fait les enregistrements.Bon, ce fichier fait 47 Go en csv sur disque, mais une fois qu'on le charge dans R, il faut plus du double en RAM... N'est pas lié à la précision des valeurs, une fois chargé l'allocation de mémoire pour de vrai les chiffres causent cette augmentation?
Peut-être une autre faute de frappe alors : 106 Mo aurait dû être 106 Go.
Je ne suis pas l'aspect NA. Mais quelques corrections en cours de route, puis réessayez à nouveau...
D'accord, veuillez réessayer. L'échantillon de devinettes est passé à 10 000 (il sera intéressant de voir combien de temps cela prendra) et les tailles de tampon ont maintenant un minimum imposé.
Vous devrez peut-être attendre au moins 30 minutes pour que le fichier du package drat soit promu.
Désolé, c'était "Go" au lieu de "Mo". Je n'avais pas assez de café.
Je vais chercher la mise à jour et la tester.
Test avec le dernier dev. data.table :
Sommaire:
Cela fonctionne rapidement (1,43 minute pour lire le csv) et l'allocation de mémoire fonctionne bien aussi, je suppose que l'allocation de RAM n'augmente pas comme avant. Un csv de 44 Go sur le disque se traduit par ~ 112 (+ 37 Go de tampons temporaires) Go de RAM une fois chargé. Est-ce lié au type de données des valeurs dans le fichier ?
Essai 1 :
Sans utiliser colClasses=list("numeric"=1:12785)
DT<-fread('dt.daily.4km.csv', verbose=TRUE)
Parameter na.strings == <<NA>>
None of the 1 na.strings are numeric (such as '-9999').
Input contains no \n. Taking this to be a filename to open
File opened, filesize is 43.772296 GB.
Memory mapping ... ok
Detected eol as \n only (no \r afterwards), the UNIX and Mac standard.
Positioned on line 1 starting: <<x,y,D_19810101,D_19810102,D_19810103,D_19810104,D_19810105,D_19810106,D_19810107,
D_19810108,D_19810109,D_19810110,D_19810111,D_19810112,
...
All the fields on line 1 are character fields. Treating as the column names.
Number of sampling jump points = 101 because 47000004016 bytes from row 1 to eof / (2 * 1281788 jump0size) == 18333
Type codes (jump 000) : 441111111111111111111111111111111111111111111111111111111111111111111111111111111111111111...1111111111 Quote rule 0
Type codes (jump 001) : 444222422222222224442444444444442222444444444444444444442444444444444222224444444444444444...4444444442 Quote rule 0
Type codes (jump 002) : 444222444444222244442444444444444422444444444444444444442444444444444222224444444444444444...4444444442 Quote rule 0
...
Type codes (jump 034) : 444444444444444444444444444444444444444444444444444444444444444444444444444444444444444444...4444444444 Quote rule 0
Type codes (jump 100) : 444444444444444444444444444444444444444444444444444444444444444444444444444444444444444444...4444444444 Quote rule 0
=====
Sampled 10049 rows (handled \n inside quoted fields) at 101 jump points including middle and very end
Bytes from first data row on line 2 to the end of last row: 47000004016
Line length: mean=79727.22 sd=32260.00 min=12804 max=153029
Estimated nrow: 47000004016 / 79727.22 = 589511
Initial alloc = 1179022 rows (589511 + 100%) using bytes/max(mean-2*sd,min) clamped between [1.1*estn, 2.0*estn]
=====
Type codes (colClasses) : 444444444444444444444444444444444444444444444444444444444444444444444444444444444444444444...4444444444
Type codes (drop|select) : 444444444444444444444444444444444444444444444444444444444444444444444444444444444444444444...4444444444
Allocating 12785 column slots (12785 - 0 dropped)
Reading 432 chunks of 103.756MB (1364 rows) using 144 threads
Read 872505 rows x 12785 columns from 43.772GB file in 02:17.726 wall clock time (can be slowed down by any other open apps even if seemingly idle)
Final type counts
0 : drop
0 : logical
0 : integer
0 : integer64
12785 : double
0 : character
Thread buffers were grown 67 times (if all 144 threads each grew once, this figure would be 144)
=============================
0.000s ( 0%) Memory map
0.099s ( 0%) sep, ncol and header detection
11.057s ( 8%) Column type detection using 10049 sample rows
0.899s ( 1%) Allocation of 872505 rows x 12785 cols (112.309GB) plus 37.433GB of temporary buffers
125.671s ( 91%) Reading data
0.000s ( 0%) Rereading 0 columns due to out-of-sample type exceptions
137.726s Total
Essai 2 :
Maintenant en utilisant colClasses=list("numeric"=1:12785)
Le timing a été amélioré de quelques secondes...
DT<-fread('dt.daily.4km.csv', colClasses=list("numeric"=1:12785), verbose=TRUE)
Allocating 12785 column slots (12785 - 0 dropped)
Reading 432 chunks of 103.756MB (1364 rows) using 144 threads
Read 872505 rows x 12785 columns from 43.772GB file in 01:43.028 wall clock time (can be slowed down by any other open apps even if seemingly idle)
Final type counts
0 : drop
0 : logical
0 : integer
0 : integer64
12785 : double
0 : character
Thread buffers were grown 67 times (if all 144 threads each grew once, this figure would be 144)
=============================
0.000s ( 0%) Memory map
0.092s ( 0%) sep, ncol and header detection
11.009s ( 11%) Column type detection using 10049 sample rows
0.332s ( 0%) Allocation of 872505 rows x 12785 cols (112.309GB) plus 37.433GB of temporary buffers
91.595s ( 89%) Reading data
0.000s ( 0%) Rereading 0 columns due to out-of-sample type exceptions
103.028s Total
Ok super - nous y arrivons. La taille de l'échantillon corrigé plus son augmentation à 100 lignes à 100 points (10 000 lignes d'échantillon) était suffisante pour deviner les types correctement alors - super. Comme il y a 12 875 colonnes et que la longueur de ligne est en moyenne de 80 000 caractères dans le fichier de 44 Go, il a fallu 11 secondes pour échantillonner. Mais ce temps en valait la peine car il évitait une relecture qui aurait pris 90s supplémentaires. Nous nous en tiendrons à ce moment-là.
Je pense que la 2ème fois est plus rapide simplement parce que c'était la 2ème fois et que votre système d'exploitation s'est réchauffé et a mis en cache le fichier. Tout ce qui s'exécute sur la boîte affectera les horaires de l'horloge murale. Cela est résolu en exécutant 3 exécutions consécutives identiques du 1er test. Puis en changeant une seule chose et en exécutant à nouveau 3 courses identiques consécutives. Avec une taille de 44 Go, vous verrez beaucoup de variance naturelle. 3 runs suffisent généralement pour conclure mais cela peut être un art noir.
Oui, les 112 Go de mémoire contre 44 Go de disque sont dus en partie au fait que les données sont plus volumineuses en mémoire car toutes les colonnes sont de type double ; il n'y a pas de compression en mémoire dans R et il y a pas mal de valeurs NA qui ne prennent pas de place dans ce CSV (juste ",," ) mais 8 octets en mémoire. Cependant, il devrait être de 83 Go et non de 112 Go (872505 lignes x 12785 cols x 8 octets doubles / 1024^3 = 83 Go). Ces 112 Go sont ce qui a été alloué en fonction de la moyenne et de l'écart type des longueurs de ligne. Sur la base de la longueur moyenne des lignes, il a estimé qu'il s'agirait de 589 511 lignes, ce qui aurait été trop faible. La variance de la longueur de ligne était si élevée que la pince est entrée en vigueur à +100 %. 58 9511 * 2 = 1 179 022 * 12785 * 8 / 1024^3 = 112 Go. En fin de compte, il a constaté que 872 505 sont dans le dossier. Mais cela ne libère pas l'espace libre. Je vais arranger ça. (À FAIRE1)
Lorsque vous avez spécifié le type de colonne pour toutes les colonnes, il a quand même été échantillonné. Il doit ignorer l'échantillonnage lorsque l'utilisateur a spécifié chaque colonne. (À FAIRE2)
Étant donné que toutes vos données sont doubles, elles effectuent 11 milliards d'appels à la fonction de bibliothèque C strtod(). La spécialisation longtemps souhaitée de cette fonction devrait en théorie permettre une accélération significative de ce dossier. (TERMINÉ)
Merci pour ça, super explication. Faites-moi savoir si vous voulez que je teste autre chose avec ce fichier. Je travaille sur un autre ensemble de données qui est davantage au format long, avec environ 21 millions de lignes x 1432 colonnes.
NAME NROW NCOL MB
[1,] DT 21,812,625 1,432 238,310
Ajout d'un point de données supplémentaire à cela. 89G .tsv , l'utilisation maximale de la mémoire pendant le chargement est d'environ 180G. Je pense que c'est à prévoir car il y a beaucoup de NA et de doubles.
Je suis également heureux de tester à ce sujet.
Ubuntu 16.04 64bit / Linux 4.4.0-71-generic
R version 3.3.2 (2016-10-31)
data.table 1.10.5 IN DEVELOPMENT built 2017-04-04 14:27:46 UTC
Architecture: x86_64
CPU op-mode(s): 32-bit, 64-bit
Byte Order: Little Endian
CPU(s): 64
On-line CPU(s) list: 0-63
Thread(s) per core: 2
Core(s) per socket: 16
Socket(s): 2
NUMA node(s): 2
Vendor ID: GenuineIntel
CPU family: 6
Model: 79
Model name: Intel(R) Xeon(R) CPU E5-2686 v4 @ 2.30GHz
Stepping: 1
CPU MHz: 2699.984
CPU max MHz: 3000.0000
CPU min MHz: 1200.0000
BogoMIPS: 4660.70
Hypervisor vendor: Xen
Virtualization type: full
L1d cache: 32K
L1i cache: 32K
L2 cache: 256K
L3 cache: 46080K
NUMA node0 CPU(s): 0-15,32-47
NUMA node1 CPU(s): 16-31,48-63
Flags: fpu vme de pse tsc msr pae mce cx8 apic sep mtrr pge mca cmov pat pse36 clflush mmx fxsr sse sse2 ht syscall nx pdpe1gb rdtscp lm constant_tsc rep_good nopl xtopology nonstop_tsc aperfmperf eagerfpu pni pclmulqdq monitor est ssse3 fma cx16 pcid sse4_1 sse4_2 x2apic movbe popcnt tsc_deadline_timer aes xsave avx f16c rdrand hypervisor lahf_lm abm fsgsbase bmi1 hle avx2 smep bmi2 erms invpcid rtm xsaveopt ida
Parameter na.strings == <<NA>>
None of the 1 na.strings are numeric (such as '-9999').
Input contains no \n. Taking this to be a filename to open
File opened, filesize is 88.603947 GB.
Memory mapping ... ok
Detected eol as \n only (no \r afterwards), the UNIX and Mac standard.
Positioned on line 1 starting: <<allele prediction_uuid sample_>>
Detecting sep ...
sep=='\t' with 101 lines of 76 fields using quote rule 0
Detected 76 columns on line 1. This line is either column names or first data row (first 30 chars): <<allele prediction_uuid sample_>>
All the fields on line 1 are character fields. Treating as the column names.
Number of sampling jump points = 101 because 95137762779 bytes from row 1 to eof / (2 * 24414 jump0size) == 1948426
Type codes (jump 000) : 5555542444111145424441111444111111111111111111111111111111111111111111111111 Quote rule 0
Type codes (jump 009) : 5555542444114445424441144444111111111111111111111111111111111111111111111111 Quote rule 0
Type codes (jump 042) : 5555542444444445424444444444111111111111111111111111111111111111111111111111 Quote rule 0
Type codes (jump 048) : 5555544444444445444444444444225225522555545111111111111111111111111111111111 Quote rule 0
Type codes (jump 083) : 5555544444444445444444444444225225522555545254452454411154454452454411154455 Quote rule 0
Type codes (jump 085) : 5555544444444445444444444444225225522555545254452454454454454452454454454455 Quote rule 0
Type codes (jump 100) : 5555544444444445444444444444225225522555545254452454454454454452454454454455 Quote rule 0
=====
Sampled 10028 rows (handled \n inside quoted fields) at 101 jump points including middle and very end
Bytes from first data row on line 2 to the end of last row: 95137762779
Line length: mean=465.06 sd=250.27 min=198 max=929
Estimated nrow: 95137762779 / 465.06 = 204571280
Initial alloc = 409142560 rows (204571280 + 100%) using bytes/max(mean-2*sd,min) clamped between [1.1*estn, 2.0*estn]
=====
Type codes (colClasses) : 5555544444444445444444444444225225522555545254452454454454454452454454454455
Type codes (drop|select) : 5555544444444445444444444444225225522555545254452454454454454452454454454455
Allocating 76 column slots (76 - 0 dropped)
Reading 90752 chunks of 1.000MB (2254 rows) using 64 threads
Si cela peut vous aider, voici les résultats sur une base de données très longue : 419 124 196 x 42 (~2^34) avec une ligne d'en-tête et colClasses transmis.
> library(data.table)
data.table 1.10.5 IN DEVELOPMENT built 2017-09-27 17:12:56 UTC; travis
The fastest way to learn (by data.table authors): https://www.datacamp.com/courses/data-analysis-the-data-table-way
Documentation: ?data.table, example(data.table) and browseVignettes("data.table")
Release notes, videos and slides: http://r-datatable.com
> CC <- c(rep('integer', 2), rep('character', 3),
+ rep('numeric', 2), rep('integer', 3),
+ rep('character', 2), 'integer', 'character', 'integer',
+ rep('character', 4), rep('numeric', 11), 'character',
+ 'numeric', 'character', rep('numeric', 2),
+ rep('integer', 3), rep('numeric', 2), 'integer',
+ 'numeric')
> P <- fread('XXXX.csv', colClasses = CC, header = TRUE, verbose = TRUE)
Input contains no \n. Taking this to be a filename to open
[01] Check arguments
Using 40 threads (omp_get_max_threads()=40, nth=40)
NAstrings = [<<NA>>]
None of the NAstrings look like numbers.
show progress = 1
0/1 column will be read as boolean
[02] Opening the file
Opening file XXXXcsv
File opened, size = 51.71GB (55521868868 bytes).
Memory mapping ... ok
[03] Detect and skip BOM
[04] Arrange mmap to be \0 terminated
\r-only line endings are not allowed because \n is found in the data
[05] Skipping initial rows if needed
Positioned on line 1 starting: <<X,X,X,X>>
[06] Detect separator, quoting rule, and ncolumns
Detecting sep ...
sep=',' with 100 lines of 42 fields using quote rule 0
Detected 42 columns on line 1. This line is either column names or first data row. Line starts as: <<X,X,X,X>>
Quote rule picked = 0
fill=false and the most number of columns found is 42
[07] Detect column types, good nrow estimate and whether first row is column names
'header' changed by user from 'auto' to true
Number of sampling jump points = 101 because (55521868866 bytes from row 1 to eof) / (2 * 13006 jump0size) == 2134471
Type codes (jump 000) : 5161010775551055105101111111111111110110771117717 Quote rule 0
Type codes (jump 022) : 5561010775551055105101111111111111110110771117717 Quote rule 0
Type codes (jump 030) : 5561010775551055105101010107517171151110110771117717 Quote rule 0
Type codes (jump 037) : 5561010775551055105101010107517771171110110771117717 Quote rule 0
Type codes (jump 073) : 5561010775551055105101010107517771177110110771117717 Quote rule 0
Type codes (jump 093) : 5561010775551055105101010107717771177110110771117717 Quote rule 0
Type codes (jump 100) : 5561010775551055105101010107717771177110110771117717 Quote rule 0
=====
Sampled 10049 rows (handled \n inside quoted fields) at 101 jump points
Bytes from first data row on line 1 to the end of last row: 55521868866
Line length: mean=132.68 sd=6.00 min=118 max=425
Estimated number of rows: 55521868866 / 132.68 = 418453923
Initial alloc = 460299315 rows (418453923 + 9%) using bytes/max(mean-2*sd,min) clamped between [1.1*estn, 2.0*estn]
=====
[08] Assign column names
[09] Apply user overrides on column types
After 11 type and 0 drop user overrides : 551010107755510105105101010107777777777710710775557757
[10] Allocate memory for the datatable
Allocating 42 column slots (42 - 0 dropped) with 460299315 rows
[11] Read the data
jumps=[0..52960), chunk_size=1048373, total_size=55521868441
Read 98%. ETA 00:00
[12] Finalizing the datatable
Read 419124195 rows x 42 columns from 51.71GB (55521868868 bytes) file in 13:42.935 wall clock time
Thread buffers were grown 0 times (if all 40 threads each grew once, this figure would be 40)
Final type counts
0 : drop
0 : bool8
0 : bool8
0 : bool8
0 : bool8
11 : int32
0 : int64
19 : float64
0 : float64
0 : float64
12 : string
=============================
0.000s ( 0%) Memory map 51.709GB file
0.016s ( 0%) sep=',' ncol=42 and header detection
0.016s ( 0%) Column type detection using 10049 sample rows
188.153s ( 23%) Allocation of 419124195 rows x 42 cols (125.177GB)
634.751s ( 77%) Reading 52960 chunks of 1.000MB (7901 rows) using 40 threads
= 0.121s ( 0%) Finding first non-embedded \n after each jump
+ 17.036s ( 2%) Parse to row-major thread buffers
+ 616.184s ( 75%) Transpose
+ 1.410s ( 0%) Waiting
0.000s ( 0%) Rereading 0 columns due to out-of-sample type exceptions
822.935s Total
> memory.size()
[1] 134270.3
> rm(P)
> gc()
used (Mb) gc trigger (Mb) max used (Mb)
Ncells 585532 31.3 5489235 293.2 6461124 345.1
Vcells 1508139082 11506.2 20046000758 152938.9 25028331901 190951.1
> memory.size()
[1] 87.56
> P <- fread('XXXX.csv', colClasses = CC, header = TRUE, verbose = TRUE)
Input contains no \n. Taking this to be a filename to open
[01] Check arguments
Using 40 threads (omp_get_max_threads()=40, nth=40)
NAstrings = [<<NA>>]
None of the NAstrings look like numbers.
show progress = 1
0/1 column will be read as boolean
[02] Opening the file
Opening file XXXX.csv
File opened, size = 51.71GB (55521868868 bytes).
Memory mapping ... ok
[03] Detect and skip BOM
[04] Arrange mmap to be \0 terminated
\r-only line endings are not allowed because \n is found in the data
[05] Skipping initial rows if needed
Positioned on line 1 starting: <<X,X,X,X>>
[06] Detect separator, quoting rule, and ncolumns
Detecting sep ...
sep=',' with 100 lines of 42 fields using quote rule 0
Detected 42 columns on line 1. This line is either column names or first data row. Line starts as: <<X,X,X,X>>
Quote rule picked = 0
fill=false and the most number of columns found is 42
[07] Detect column types, good nrow estimate and whether first row is column names
'header' changed by user from 'auto' to true
Number of sampling jump points = 101 because (55521868866 bytes from row 1 to eof) / (2 * 13006 jump0size) == 2134471
Type codes (jump 000) : 5161010775551055105101111111111111110110771117717 Quote rule 0
Type codes (jump 022) : 5561010775551055105101111111111111110110771117717 Quote rule 0
Type codes (jump 030) : 5561010775551055105101010107517171151110110771117717 Quote rule 0
Type codes (jump 037) : 5561010775551055105101010107517771171110110771117717 Quote rule 0
Type codes (jump 073) : 5561010775551055105101010107517771177110110771117717 Quote rule 0
Type codes (jump 093) : 5561010775551055105101010107717771177110110771117717 Quote rule 0
Type codes (jump 100) : 5561010775551055105101010107717771177110110771117717 Quote rule 0
=====
Sampled 10049 rows (handled \n inside quoted fields) at 101 jump points
Bytes from first data row on line 1 to the end of last row: 55521868866
Line length: mean=132.68 sd=6.00 min=118 max=425
Estimated number of rows: 55521868866 / 132.68 = 418453923
Initial alloc = 460299315 rows (418453923 + 9%) using bytes/max(mean-2*sd,min) clamped between [1.1*estn, 2.0*estn]
=====
[08] Assign column names
[09] Apply user overrides on column types
After 11 type and 0 drop user overrides : 551010107755510105105101010107777777777710710775557757
[10] Allocate memory for the datatable
Allocating 42 column slots (42 - 0 dropped) with 460299315 rows
[11] Read the data
jumps=[0..52960), chunk_size=1048373, total_size=55521868441
Read 98%. ETA 00:00
[12] Finalizing the datatable
Read 419124195 rows x 42 columns from 51.71GB (55521868868 bytes) file in 05:04.910 wall clock time
Thread buffers were grown 0 times (if all 40 threads each grew once, this figure would be 40)
Final type counts
0 : drop
0 : bool8
0 : bool8
0 : bool8
0 : bool8
11 : int32
0 : int64
19 : float64
0 : float64
0 : float64
12 : string
=============================
0.000s ( 0%) Memory map 51.709GB file
0.031s ( 0%) sep=',' ncol=42 and header detection
0.000s ( 0%) Column type detection using 10049 sample rows
28.437s ( 9%) Allocation of 419124195 rows x 42 cols (125.177GB)
276.442s ( 91%) Reading 52960 chunks of 1.000MB (7901 rows) using 40 threads
= 0.017s ( 0%) Finding first non-embedded \n after each jump
+ 12.941s ( 4%) Parse to row-major thread buffers
+ 262.989s ( 86%) Transpose
+ 0.495s ( 0%) Waiting
0.000s ( 0%) Rereading 0 columns due to out-of-sample type exceptions
304.910s Total
> memory.size()
[1] 157049.7
> sessionInfo()
R version 3.4.2 beta (2017-09-17 r73296)
Platform: x86_64-w64-mingw32/x64 (64-bit)
Running under: Windows Server >= 2012 x64 (build 9200)
Matrix products: default
locale:
[1] LC_COLLATE=English_United States.1252 LC_CTYPE=English_United States.1252
[3] LC_MONETARY=English_United States.1252 LC_NUMERIC=C
[5] LC_TIME=English_United States.1252
attached base packages:
[1] stats graphics grDevices utils datasets methods base
other attached packages:
[1] data.table_1.10.5
loaded via a namespace (and not attached):
[1] compiler_3.4.2 tools_3.4.2
Quelques notes. J'aurais suggéré de mettre [09] avant [07] pour dire que si colClasses est passé, il n'y a aucune raison de vérifier. De plus, Windows affichait environ 160 Go d'utilisation après chaque exécution. memory.size() fait probablement un peu de nettoyage. Avec 532 Go de RAM sur ce serveur, la mise en cache mémoire peut avoir de quoi faire avec l'augmentation de la vitesse lors de la deuxième exécution. J'espère que cela pourra aider.
Test sur une large table "du monde réel" (données hospitalières): 30M lignes × 125 colonnes v readr '1.2.0' et read.csv 3.4.3.
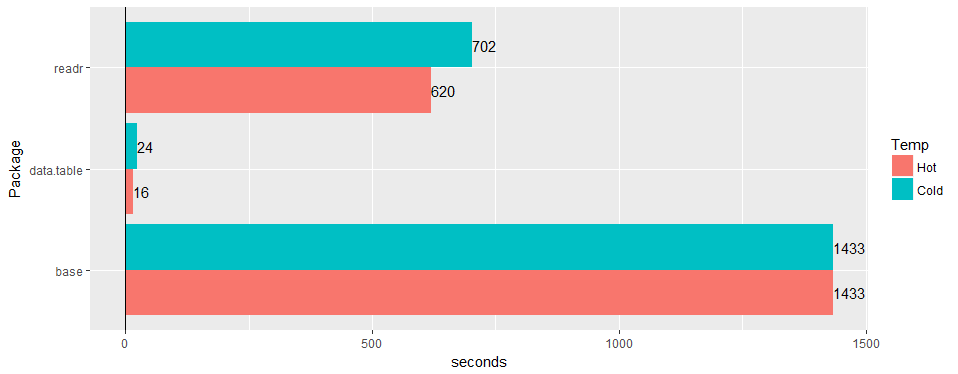
une chance de confirmer que le problème est toujours valide sur 1.11.4? ou du code pour produire des exemples de données.
Celui-ci est tout résolu pour autant que je sache. @geponce s'il vous plaît mettre à jour sinon.
TODO1 ci-dessus maintenant déposé sous le numéro 3024
TODO2 ci-dessus maintenant déposé sous le numéro 3025
Commentaire le plus utile
Test sur une large table "du monde réel" (données hospitalières): 30M lignes × 125 colonnes v readr '1.2.0' et
read.csv3.4.3.