Data.table: fread con gran csv (44 GB) requiere mucha RAM en la última versión de desarrollo de data.table
Hola,
Hardware y software:
Servidor : Dell R930 4-Intel Xeon E7-8870 v3 2.1GHz, 45M Cache, 9.6GT / s QPI, Turbo, HT, 18C / 36T y 1TB en RAM
SO : Redhat 7.1
Versión R : 3.3.2
versión de data.table : 1.10.5 construido 2017-03-21
Estoy cargando un archivo csv (44 GB, 872505 filas x 12785 columnas). Se carga muy rápido, en 1,30 minutos usando 144 núcleos (72 núcleos de los 4 procesadores con hyperthreading habilitado para convertirlo en una caja de 144 núcleos).
El problema principal es que cuando se carga el DT, la cantidad de memoria en uso aumenta significativamente en relación con el tamaño del archivo csv. En este caso, el csv de 44 GB (guardado con fwrite, guardado con saveRDS y compress = FALSE crea un archivo de 84 GB) está usando ~ 356 GB de RAM.
Aquí está la salida usando "verbose = TRUE"
_Asignación de 12785 espacios de columna (12785 - 0 eliminado)
secuencial madvise: ok
Lectura de datos con 1440 puntos de salto y 144 hilos
Leer 95,7% de 858881 filas estimadas
Leer 872505 filas x 12785 columnas de un archivo de 43,772 GB en 1 minuto 33,736 segundos de tiempo de reloj de pared (afectado por la ejecución de otras aplicaciones)
0.000s (0%) Mapa de memoria
0.070s (0%) sep, ncol y detección de encabezado
26.227s (28%) Detección de tipo de columna usando 34832 filas de muestra de 1440 puntos de salto
0,614 s (1%) Asignación de 3683116 filas x 12785 columnas (350,838 GB) en RAM
0.000s (0%) madvise secuencial
66.825 s (71%) Lectura de datos
93.736s Total_
Muestra un problema similar que a veces surge cuando se trabaja con el paquete paralelo, donde se lanza una rsesión por núcleo cuando se usan funciones como "mclapply". Vea las Rsessions creadas / enumeradas en esta captura de pantalla:

si hago "rm (DT)", la RAM vuelve al estado inicial y las "Rsessions" se eliminan.
Ya probé, por ejemplo, "setDTthreads (20)" y todavía usa la misma cantidad de RAM.
Por cierto, si el archivo se carga con la versión no paralela de "fread", la asignación de memoria solo llega a ~ 106 GB.
Guillermo
Todos 30 comentarios
Este es el resultado de la implementación de fread no paralela (tabla de datos 1.10.5 EN DESARROLLO construido 2017-02-09)

Y verifiqué dos veces la cantidad de memoria utilizada y solo sube a 84GB.
Guillermo
Sí tienes razón. Gracias por el gran informe. El nrow estimado parece correcto (858,881 vs 872,505) pero luego la asignación es 4.2 veces más grande que eso (3,683,116) y muy lejos. Mejoré el cálculo y agregué más detalles a la salida detallada. Sin embargo, no vuelva a realizar la prueba hasta que se hayan hecho algunas cosas más.
Ok, por favor vuelva a probar - debería ser arreglado ahora.
Acabo de instalar data.table dev:
data.table 1.10.5 EN DESARROLLO construido 2017-03-27 02:50:31 UTC
Lo primero que recibí cuando intenté leer el mismo archivo de 44 GB fue este mensaje:
DT <- fread ('dt.daily.4km.csv')
Error: proteger (): desbordamiento de la pila de protección
Luego volví a ejecutar el mismo comando y comencé a trabajar bien. Sin embargo, esta versión no utiliza un modo multinúcleo. La carga está tardando ~ 25 minutos, igual que antes de poner la versión fread-paralela.
Guillermo
Cerré todas las r-sesiones y volví a ejecutar una prueba y recibo un error:
El tipo de columna adivinado fue insuficiente para 34711745 valores en 508 columnas. Utilice colClasses para configurar estas clases de columna manualmente.
Vea a continuación, recibo varios mensajes sobre "entero adivinado pero contiene << 0 .... >>
_Leer 872505 filas x 12785 columnas de un archivo de 43.772GB en 15: 27.024 tiempo de reloj de pared (puede ralentizarse con cualquier otra aplicación abierta incluso si aparentemente está inactiva)
La columna 171 ('D_19810618') supuso 'entero' pero contiene << 2.23000001907349 >>
La columna 347 ('D_19811211') supuso 'entero' pero contiene << 1.02999997138977 >>
La columna 348 ('D_19811212') supuso 'entero' pero contiene << 3,75 >> _
Vaya, su archivo realmente está probando los casos extremos. Excelente. En el futuro, ejecute verbose=TRUE y proporcione el resultado completo. Pero con la información que proporcionó en este caso, puedo ver cuál es el problema en realidad. Hay un búfer creado para cada columna para cada hilo (en este caso, más de 12.000 columnas). Cada uno está PROTEGIDO por separado actualmente. Hay una forma de evitar eso: servirá. Los mensajes sobre el tipo de adivinación son correctos. ¿Esas 508 columnas significan algo para usted y está de acuerdo en que deben ser numéricas? Puede pasar un rango de columnas a colClasses así: colClasses=list("numeric"=11:518)
¿De qué campo son estos datos? ¿Estás creando el archivo? Parece que se ha torcido a un formato amplio, donde la mejor práctica normal es escribir (y mantenerlo en la memoria también) en formato largo. Normalmente, esperaría ver los nombres de 508 columnas como "D_19810618" como valores en una columna, no como columnas en sí mismas. Por eso le pregunto si está creando el archivo y si puede crearlo en formato largo. Si no es así, sugiera a quien esté creando el archivo que puede hacerlo mejor. Supongo que está aplicando operaciones a través de columnas, quizás usando .SD y .SDcols . Es mucho mejor en formato largo y keyby= la columna que contiene valores como "D_19810618".
Pero aún intentaré hacer que fread sea lo mejor posible al tratar con cualquier entrada, incluso archivos muy anchos de más de 12,000 columnas.
¡Espero que otras personas estén probando y no encuentren ningún problema en sus archivos!
_ ¿Esas 508 columnas significan algo para ti y estás de acuerdo en que deben ser numéricas?
Esta tabla contiene series de tiempo por fila. IDx, IDy, Time1_value, Time2_value, Time3_value ... y todas las columnas Time N _value contienen solo valores numéricos. Si utilizo colClasses, tendré que hacerlo para 12783: list ("numeric" = 2: 12783). Intentaré esto.
_¿De qué campo son estos datos? _
Datos geoespaciales. Estoy haciendo búsquedas a distancia euclidiana dentro de DT usando IDx e IDy. Supongo que cuantas más filas, más lentas serán las búsquedas, ¿verdad?
Ahora mismo es bastante rápido (formato ancho). Tengo un mapa, donde un usuario puede hacer clic en un área y luego se genera una serie de tiempo dentro de un archivo csv desde la ubicación más cercana (con datos disponibles) al clic dado. Lo implementaré con un formato largo en su lugar.
Volveré con algunos resultados.
Está bien. No necesita list("numeric"=2:12783) iiuc porque solo necesita ayuda con 508 columnas. Oh, ya veo, los 508 están dispersos a través de las columnas, supongo entonces (no son un conjunto contiguo de columnas).
No, ¡data.table casi nunca es más rápido cuando es ancho! Long es casi siempre más rápido y más conveniente. ¿Has visto y probado roll="nearest" ? ¿Cómo lo estás haciendo ahora? Muestra el código para que podamos entenderlo. Es casi seguro que el formato largo es mejor, pero es posible que necesitemos algunas mejoras para el 2D más cercano. Por favor, muestre también los tiempos. Cuando dices "bastante rápido", resulta que la gente tiene ideas tremendamente diferentes sobre lo que es "bastante rápido".
derritiendo esta mesa alcanza el límite de 2 ^ 31. Recibo el error: "los vectores de longitud negativa no están permitidos".
Volveré a la fuente para ver si puedo generarlo en formato largo.
# Read Data
DT <- fread('dt.daily.4km.csv', showProgress = FALSE)
# Add two columns with truncated values of x and y (these are geog. coords.)
DT[,y_tr:=trunc(y)]
DT[,x_tr:=trunc(x)]
# For using on plotting (x-axis values)
xaxis<-seq.Date(as.Date("1981-01-01"),as.Date("2015-12-31"), "day")
# subset by truncated coordinates to avoid full-table search. Now searches
# will happen in a smaller subset
DT2 <- DT[y_tr==trunc(y_clicked) & x_tr==trunc(x_clicked),]
# Add distance from each point in the data.table to the provided location, "gdist" is from
# Imap package for euclidean distance.
DT2[,DIST:=gdist(lat.1 = DT2$y,
lon.1 = DT2$x,
lat.2 = y_clicked,
lon.2 = x_clicked, units="miles")]
# Get the minimum distance
minDist <- min(DT2[,DIST])
# Get the y-axis values
yt <- transpose(DT2[DIST==minDist,3:(ncol(DT2)-3)])$V1`
# Ready to plot xaxis vs yt
...
...
No tengo la aplicación en un servidor público. Esencialmente, el usuario puede hacer clic en un mapa y luego capturar esas coordenadas y realizar la búsqueda anterior, obtener la serie de tiempo y crear un gráfico.
Encontró y corrigió otro desbordamiento de pila para una gran cantidad de columnas: https://github.com/Rdatatable/data.table/commit/d0469e670961dcdea115d433c0f2dce596d65906. Olvidé etiquetar este número de problema en el mensaje de confirmación.
Oh. Ese es el punto. 872505 filas * 12780 columnas son 11 mil millones de filas. Entonces, mi sugerencia de usar un formato largo no funcionará para usted, ya que eso es> 2 ^ 31. Lo siento, debería haberlo visto. Tendremos que morder la bala e ir> 2 ^ 31 entonces. Mientras tanto, sigamos con el formato ancho que tiene funcionando y lo concretaré.
Inténtalo de nuevo. El uso de memoria debería volver a la normalidad y debería volver a leer automáticamente la 508 de 12,785 columnas con excepciones de tipo fuera de muestra. Para evitar el tiempo de repetición automática, puede configurar colClasses .
Si no se corrige, pegue el resultado completo y detallado. ¡Dedos cruzados!
OK...
Un resumen de resultados con la última data.table dev: data.table 1.10.5 EN DESARROLLO construido 2017-03-29 16:17:01 UTC
Cuatro puntos principales a mencionar:
- Fread funcionó bien leyendo el archivo en la discusión.
- La lectura tardó ~ 5,5 minutos en comparación con la versión anterior que tardó ~ 1,3 minutos.
- Esta versión no aumenta la asignación de RAM como la versión anterior que estaba probando.
- Los núcleos parecen estar "menos activos" (vea la captura de pantalla a continuación).
Algunos comentarios / preguntas:
1.
El porcentaje de uso en los núcleos no está tan activo como antes, vea la captura de pantalla:
En la versión anterior de fread, la actividad de los núcleos siempre fue ~ 90-80%. En esta versión se mantuvo alrededor de ~ 2-3% cada núcleo como se muestra en la imagen de arriba.
No entiendo por qué fread está haciendo saltos de entero a numérico: por ejemplo
Column 1489 ("D_19850126") bumped from 'integer' to 'numeric' due to <<0.949999988079071>> somewhere between row 6041 and row 24473
Revisé esas filas y parece estar bien. ¿Por qué se detecta como un número entero y es necesario 'pasar' a 'numérico' si ya es numérico (consulte el resumen de las filas sugeridas a continuación)? ¿O estoy entendiendo mal esta línea? Esto sucede para 508 líneas. ¿NA está causando problemas?
summary(DT[6041:24473,.(D_19850126)])
D_19850126
Min. :0.750
1st Qu.:0.887
Median :0.945
Mean :0.966
3rd Qu.:1.045
Max. :1.210
NA's :18393
A continuación, algunos detalles de la prueba.
DT<-fread('dt.daily.4km.ver032917.csv', verbose=TRUE)
PRODUCCIÓN
Parameter na.strings == <<NA>>
None of the 0 na.strings are numeric (such as '-9999').Input contains no \n. Taking this to be a filename to open
File opened, filesize is 43.772296 GB.
Memory mapping ... ok
Detected eol as \n only (no \r afterwards), the UNIX and Mac standard.
Positioned on line 1 starting: <<x,y,D_19810101,D_19810102,D_19810103,D_19810104,D_19810105,D_19810106,D_19810107,D_19810108,D_19810109,D_19810110,D_19810111,D_19810112,D_19810113,
...
...
All the fields on line 1 are character fields. Treating as the column names.
Number of sampling jump points = 11 because 1281788 startSize * 10 NJUMPS * 2 = 25635760 <= -244636240 bytes from line 2 to eof
Type codes (jump 00) : 441111111111111111111111111111111111111111111111111111111111111111111111111111111111111111...1111111111 Quote rule 0
Type codes (jump 01) : 444422222222222222242444424444442222222222424444424442224424222244222222222242422222224422...4442244422 Quote rule 0
Type codes (jump 02) : 444422222222222222242444424444442422222242424444424442224424222244222222222242424222424424...4442444442 Quote rule 0
Type codes (jump 03) : 444422222222222224242444424444444422222244424444424442224424222244222222222244424442424424...4442444442 Quote rule 0
Type codes (jump 04) : 444444244422222224442444424444444444242244444444444444424444442244444222222244424442444444...4444444444 Quote rule 0
Type codes (jump 05) : 444444244422222224442444424444444444242244444444444444424444442244444222222244424442444444...4444444444 Quote rule 0
Type codes (jump 06) : 444444444422222224442444424444444444242244444444444444424444444444444244444444424442444444...4444444444 Quote rule 0
Type codes (jump 07) : 444444444422222224442444424444444444242244444444444444444444444444444244444444424444444444...4444444444 Quote rule 0
Type codes (jump 08) : 444444444442222224444444424444444444242244444444444444444444444444444444444444424444444444...4444444444 Quote rule 0
Type codes (jump 09) : 444444444442222424444444424444444444244444444444444444444444444444444444444444424444444444...4444444444 Quote rule 0
Type codes (jump 10) : 444444444442222424444444424444444444244444444444444444444444444444444444444444424444444444...4444444444 Quote rule 0
=====
Sampled 305 rows (handled \n inside quoted fields) at 11 jump points including middle and very end
Bytes from first data row on line 2 to the end of last row: 47000004016
Line length: mean=45578.20 sd=33428.37 min=12815 max=108497
Estimated nrow: 47000004016 / 45578.20 = 1031195
Initial alloc = 2062390 rows (1031195 + 100%) using bytes/max(mean-2*sd,min) clamped between [1.1*estn, 2.0*estn]
=====
Type codes (colClasses) : 444444444442222424444444424444444444244444444444444444444444444444444444444444424444444444...4444444444
Type codes (drop|select): 444444444442222424444444424444444444244444444444444444444444444444444444444444424444444444...4444444444
Allocating 12785 column slots (12785 - 0 dropped)
Reading 44928 chunks of 0.998MB (22 rows) using 144 threads
Read 872505 rows x 12785 columns from 43.772GB file in 05:26.908 wall clock time (can be slowed down by any other open apps even if seemingly idle)
Final type counts
0 : drop
0 : logical
0 : integer
0 : integer64
12785 : numeric
0 : character
Rereading 508 columns due to out-of-sample type exceptions.
Column 171 ("D_19810618") bumped from 'integer' to 'numeric' due to <<2.23000001907349>> somewhere between row 6041 and row 24473
Column 347 ("D_19811211") bumped from 'integer' to 'numeric' due to <<1.02999997138977>> somewhere between row 6041 and row 24473
Column 348 ("D_19811212") bumped from 'integer' to 'numeric' due to <<3.75>> somewhere between row 6041 and row 24473
Column 643 ("D_19821003") bumped from 'integer' to 'numeric' due to <<1.04999995231628>> somewhere between row 6041 and row 24473
Column 1066 ("D_19831130") bumped from 'integer' to 'numeric' due to <<1.46000003814697>> somewhere between row 6041 and row 24473
Column 1102 ("D_19840105") bumped from 'integer' to 'numeric' due to <<0.959999978542328>> somewhere between row 6041 and row 24473
Column 1124 ("D_19840127") bumped from 'integer' to 'numeric' due to <<0.620000004768372>> somewhere between row 6041 and row 24473
Column 1130 ("D_19840202") bumped from 'integer' to 'numeric' due to <<0.540000021457672>> somewhere between row 6041 and row 24473
Column 1489 ("D_19850126") bumped from 'integer' to 'numeric' due to <<0.949999988079071>> somewhere between row 6041 and row 24473
Column 1508 ("D_19850214") bumped from 'integer' to 'numeric' due to <<0.360000014305115>> somewhere between row 6041 and row 24473
...
...
Reread 872505 rows x 508 columns in 05:29.167
Read 872505 rows. Exactly what was estimated and allocated up front
Thread buffers were grown 0 times (if all 144 threads each grew once, this figure would be 144)
=============================
0.000s ( 0%) Memory map
0.093s ( 0%) sep, ncol and header detection
0.186s ( 0%) Column type detection using 305 sample rows from 44928 jump points
0.600s ( 0%) Allocation of 872505 rows x 12785 cols (192.552GB) plus 1.721GB of temporary buffers
326.029s ( 50%) Reading data
329.167s ( 50%) Rereading 508 columns due to out-of-sample type exceptions
656.075s Total
Este último resumen tomó mucho tiempo para terminarlo (~ 6 minutos). Si contamos este detallado resumen, todo el proceso tomó ~ 11 minutos. Está releyendo esas 508 columnas, incluso puedo ver el mensaje "Releyendo 508 columnas debido a excepciones de tipo fuera de muestra" sin usar el verbose = TRUE.
Compruébalo sin "verbose = TRUE"
ptm<-proc.time()
DT<-fread('dt.daily.4km.ver032917.csv')
Read 872505 rows x 12785 columns from 43.772GB file in 05:26.647 wall clock time (can be slowed down by any other open apps even if seemingly idle)
Rereading 508 columns due to out-of-sample type exceptions.
Reread 872505 rows x 508 columns in 05:30.276
proc.time() - ptm
user system elapsed
2113.100 85.919 657.870
prueba de escritura
Está funcionando bien. Realmente rápido. Pensé que escribir era más lento que leer en estos días.
fwrite(DT,'dt.daily.4km.ver032917.csv', verbose=TRUE)
No list columns are present. Setting sep2='' otherwise quote='auto' would quote fields containing sep2.
maxLineLen=151187 from sample. Found in 1.890s
Writing column names ... done in 0.000s
Writing 872505 rows in 32315 batches of 27 rows (each buffer size 8MB, showProgress=1, nth=144) ...
done (actual nth=144, anyBufferGrown=no, maxBuffUsed=46%)
La "Relectura" está tomando bastante tiempo. Es como leer el archivo dos veces.
¡Excelente! Gracias por toda la información.
- Sí, tienes razón, no debería ser releído. Pasó
colClasses=list("numeric"=1:12785)por lo que la línea de salida que comienza conType code (colClasses)debe tener el valor 4. Arreglará y agregará la prueba faltante. - En 5,5 m frente a 1,3 m, eso es interesante. El búfer de cada hilo es actualmente de 1 MB. La idea es ser pequeño para caber en el caché. Pero en su caso 1MB / 12785 cols = 82 bytes. Por lo que es 50 veces más ineficaz en caché con esa elección. Supongo que de todos modos. Nunca hubiera pensado en eso sin tus pruebas. Cuando funcionaba a una velocidad de 1,3 m, no tenía ese tamaño de 1 MB.
- También es muy extraño por qué solo ha muestreado 305 líneas. Debería haber muestreado 1,000. Si se soluciona, tal vez detecte las columnas 508 sin que usted tenga que hacerlo. La muestra no está demorando (0,186 s), por lo que se podría aumentar el tamaño de la muestra.
¿Puede pegar la salida del comando lscpu unix por favor? Esto nos dirá los tamaños de su caché y puedo pensar a partir de ahí. Proporcionaré buffMB como parámetro a fread para que pueda ver si es así. Puedo hacer un mejor cálculo si es así.
Mi error en un punto de mi publicación anterior. El colClasses=list("numeric"=1:12785) está funcionando bien. Si no especifico "colClasses", hace la "Relectura". Perdón por la confusión.
Una cosa que noté es que si no especifico las "colClasses", la tabla se crea con NA y la RAM se muestra como si el DT se cargara normalmente (~ 106 MB en RAM).
Aquí está la salida de lscpu :
Architecture: x86_64
CPU op-mode(s): 32-bit, 64-bit
Byte Order: Little Endian
CPU(s): 144
On-line CPU(s) list: 0-143
Thread(s) per core: 2
Core(s) per socket: 18
Socket(s): 4
NUMA node(s): 4
Vendor ID: GenuineIntel
CPU family: 6
Model: 63
Model name: Intel(R) Xeon(R) CPU E7-8870 v3 @ 2.10GHz
Stepping: 4
CPU MHz: 2898.328
BogoMIPS: 4195.66
Virtualization: VT-x
L1d cache: 32K
L1i cache: 32K
L2 cache: 256K
L3 cache: 46080K
NUMA node0 CPU(s): 0,4,8,12,16,20,24,28,32,36,40,44,48,52,56,60,64,68,72,76,80,84,88,92,96,100,104,108,112,116,120,124,128,132,136,140
NUMA node1 CPU(s): 1,5,9,13,17,21,25,29,33,37,41,45,49,53,57,61,65,69,73,77,81,85,89,93,97,101,105,109,113,117,121,125,129,133,137,141
NUMA node2 CPU(s): 2,6,10,14,18,22,26,30,34,38,42,46,50,54,58,62,66,70,74,78,82,86,90,94,98,102,106,110,114,118,122,126,130,134,138,142
NUMA node3 CPU(s): 3,7,11,15,19,23,27,31,35,39,43,47,51,55,59,63,67,71,75,79,83,87,91,95,99,103,107,111,115,119,123,127,131,135,139,143
Ok lo tengo. Gracias.
Al volver a leer sus primeros comentarios, ¿tendría más sentido si dijera "pasando de entero a doble" en lugar de "pasando de entero a numérico"?
¿Qué quieres decir con "creado con NA's"? Toda la tabla está llena de NA, ¿solo las 508 columnas?
¿Qué es 'DT se cargó normalmente (~ 106 MB en RAM)'. Es un archivo de 44 GB, entonces, ¿cómo pueden 106 MB ser normales?
Bueno, en este archivo todo lo que tengo son números reales y NA. No tengo valores enteros. ¿Cuándo lanza ese mensaje, "pasando del tipo de datosA al tipo de datosB"?
Eso es exactamente lo que quiero decir. En la versión actual de data.table que instalé, si omito las colClasses, carga el DT pero lleno de NA.
DT[!is.na(D_19821001),]produce 0 registros y si cargo la tabla con colClasses y hago el mismo filtrado, en realidad muestra los registros.Bueno, este archivo tiene 47 GB como csv en disco, pero una vez que lo cargas en R, toma más del doble en RAM ... No está relacionado con la precisión de los valores, una vez que se carga la asignación de memoria real los números causan ese aumento?
Quizás otro error tipográfico entonces: 106 MB deberían haber sido 106 GB.
No sigo el aspecto de NA. Pero algunas correcciones en el camino y luego vuelve a intentarlo de nuevo ...
Ok, inténtalo de nuevo. La muestra de adivinanzas ha aumentado a 10,000 (será interesante ver cuánto tiempo toma) y los tamaños de búfer ahora tienen un mínimo impuesto.
Es posible que deba esperar al menos 30 minutos para que se promocione el archivo del paquete drat.
Lo siento, fue "GB" en lugar de "MB". No tomé suficiente café.
Conseguiré la actualización y la probaré.
Pruebas con la última data.table dev .: data.table 1.10.5 EN DESARROLLO construido 2017-03-30 16:31:45 UTC :
Resumen:
Funciona rápido (1,43 minutos para leer el csv) y la asignación de memoria también funciona bien, supongo, la asignación de RAM no aumenta como antes. Un csv de 44 GB en disco se traduce en ~ 112 (+ 37 GB de búfer de temperatura) GB en RAM una vez que se carga. ¿Está esto relacionado con el tipo de datos de los valores en el archivo?
Prueba 1 :
Sin usar colClasses=list("numeric"=1:12785)
DT<-fread('dt.daily.4km.csv', verbose=TRUE)
Parameter na.strings == <<NA>>
None of the 1 na.strings are numeric (such as '-9999').
Input contains no \n. Taking this to be a filename to open
File opened, filesize is 43.772296 GB.
Memory mapping ... ok
Detected eol as \n only (no \r afterwards), the UNIX and Mac standard.
Positioned on line 1 starting: <<x,y,D_19810101,D_19810102,D_19810103,D_19810104,D_19810105,D_19810106,D_19810107,
D_19810108,D_19810109,D_19810110,D_19810111,D_19810112,
...
All the fields on line 1 are character fields. Treating as the column names.
Number of sampling jump points = 101 because 47000004016 bytes from row 1 to eof / (2 * 1281788 jump0size) == 18333
Type codes (jump 000) : 441111111111111111111111111111111111111111111111111111111111111111111111111111111111111111...1111111111 Quote rule 0
Type codes (jump 001) : 444222422222222224442444444444442222444444444444444444442444444444444222224444444444444444...4444444442 Quote rule 0
Type codes (jump 002) : 444222444444222244442444444444444422444444444444444444442444444444444222224444444444444444...4444444442 Quote rule 0
...
Type codes (jump 034) : 444444444444444444444444444444444444444444444444444444444444444444444444444444444444444444...4444444444 Quote rule 0
Type codes (jump 100) : 444444444444444444444444444444444444444444444444444444444444444444444444444444444444444444...4444444444 Quote rule 0
=====
Sampled 10049 rows (handled \n inside quoted fields) at 101 jump points including middle and very end
Bytes from first data row on line 2 to the end of last row: 47000004016
Line length: mean=79727.22 sd=32260.00 min=12804 max=153029
Estimated nrow: 47000004016 / 79727.22 = 589511
Initial alloc = 1179022 rows (589511 + 100%) using bytes/max(mean-2*sd,min) clamped between [1.1*estn, 2.0*estn]
=====
Type codes (colClasses) : 444444444444444444444444444444444444444444444444444444444444444444444444444444444444444444...4444444444
Type codes (drop|select) : 444444444444444444444444444444444444444444444444444444444444444444444444444444444444444444...4444444444
Allocating 12785 column slots (12785 - 0 dropped)
Reading 432 chunks of 103.756MB (1364 rows) using 144 threads
Read 872505 rows x 12785 columns from 43.772GB file in 02:17.726 wall clock time (can be slowed down by any other open apps even if seemingly idle)
Final type counts
0 : drop
0 : logical
0 : integer
0 : integer64
12785 : double
0 : character
Thread buffers were grown 67 times (if all 144 threads each grew once, this figure would be 144)
=============================
0.000s ( 0%) Memory map
0.099s ( 0%) sep, ncol and header detection
11.057s ( 8%) Column type detection using 10049 sample rows
0.899s ( 1%) Allocation of 872505 rows x 12785 cols (112.309GB) plus 37.433GB of temporary buffers
125.671s ( 91%) Reading data
0.000s ( 0%) Rereading 0 columns due to out-of-sample type exceptions
137.726s Total
Prueba 2 :
Ahora usando colClasses=list("numeric"=1:12785)
El tiempo se mejoró en unos segundos ...
DT<-fread('dt.daily.4km.csv', colClasses=list("numeric"=1:12785), verbose=TRUE)
Allocating 12785 column slots (12785 - 0 dropped)
Reading 432 chunks of 103.756MB (1364 rows) using 144 threads
Read 872505 rows x 12785 columns from 43.772GB file in 01:43.028 wall clock time (can be slowed down by any other open apps even if seemingly idle)
Final type counts
0 : drop
0 : logical
0 : integer
0 : integer64
12785 : double
0 : character
Thread buffers were grown 67 times (if all 144 threads each grew once, this figure would be 144)
=============================
0.000s ( 0%) Memory map
0.092s ( 0%) sep, ncol and header detection
11.009s ( 11%) Column type detection using 10049 sample rows
0.332s ( 0%) Allocation of 872505 rows x 12785 cols (112.309GB) plus 37.433GB of temporary buffers
91.595s ( 89%) Reading data
0.000s ( 0%) Rereading 0 columns due to out-of-sample type exceptions
103.028s Total
Muy bien, estamos llegando. El tamaño de muestra corregido más el aumento a 100 líneas en 100 puntos (10,000 líneas de muestra) fue suficiente para adivinar los tipos correctamente en ese momento, genial. Dado que hay 12,875 columnas y la longitud de línea es un promedio de 80,000 caracteres en el archivo de 44 GB, se necesitaron 11 segundos para muestrear. Pero ese tiempo valió la pena porque evitó una nueva lectura que habría tomado 90 segundos más. Seguiremos con eso entonces.
Creo que la segunda vez es más rápida solo porque es la segunda vez y su sistema operativo se ha calentado y almacenado en caché el archivo. Cualquier otra cosa que se ejecute en la caja afectará los tiempos del reloj de pared. Eso se soluciona ejecutando 3 ejecuciones consecutivas idénticas de la primera prueba. Luego, cambiando solo una cosa y ejecutando 3 carreras consecutivas idénticas nuevamente. Con un tamaño de 44 GB, verá mucha variación natural. 3 carreras suelen ser suficientes para llegar a una conclusión, pero puede ser un arte negro.
Sí, los 112 GB de memoria frente a los 44 GB de disco se deben en parte a que los datos son más grandes en la memoria porque todas las columnas son de tipo doble; no hay compresión en memoria en R y hay bastantes valores NA que no ocupan espacio en este CSV (solo ",," ) pero 8 bytes en la memoria. Sin embargo, debería ser de 83 GB, no de 112 GB (872505 filas x 12785 columnas x 8 bytes dobles / 1024 ^ 3 = 83 GB). Ese 112 GB es lo que se asignó en función de la desviación media y estándar de las longitudes de línea. Con base en la longitud promedio de la línea, estimó que serían 589,511 filas, lo que habría sido demasiado bajo. La variación de la longitud de la línea fue tan alta que la abrazadera entró en vigor al + 100%. 58,9511 * 2 = 1,179,022 * 12785 * 8/1024 ^ 3 = 112GB. Al final, encontró que 872,505 están en el archivo. Pero no está liberando el espacio libre. Yo arreglaré eso. (TODO1)
Sin embargo, cuando especificó el tipo de columna para todas las columnas, aún se muestreó. Debería omitir el muestreo cuando el usuario haya especificado cada columna. (TODO2)
Dado que todos sus datos son el doble, está realizando 11 mil millones de llamadas a la función de biblioteca C strtod (). La largamente deseada especialización de esa función debería, en teoría, hacer una aceleración significativa para este archivo. (HECHO)
Gracias por eso, gran explicación. Avísame si quieres que pruebe algo más con este archivo. Estoy trabajando en otro conjunto de datos que tiene más formato largo, con ~ 21 millones de filas x 1432 columnas.
NAME NROW NCOL MB
[1,] DT 21,812,625 1,432 238,310
Añadiendo un punto de datos adicional a esto. 89G .tsv , el uso máximo de memoria durante la carga es ~ 180G. Creo que esto es lo esperado ya que hay muchos NA y el doble.
También estoy feliz de probar esto.
Ubuntu 16.04 64bit / Linux 4.4.0-71-generic
R version 3.3.2 (2016-10-31)
data.table 1.10.5 IN DEVELOPMENT built 2017-04-04 14:27:46 UTC
Architecture: x86_64
CPU op-mode(s): 32-bit, 64-bit
Byte Order: Little Endian
CPU(s): 64
On-line CPU(s) list: 0-63
Thread(s) per core: 2
Core(s) per socket: 16
Socket(s): 2
NUMA node(s): 2
Vendor ID: GenuineIntel
CPU family: 6
Model: 79
Model name: Intel(R) Xeon(R) CPU E5-2686 v4 @ 2.30GHz
Stepping: 1
CPU MHz: 2699.984
CPU max MHz: 3000.0000
CPU min MHz: 1200.0000
BogoMIPS: 4660.70
Hypervisor vendor: Xen
Virtualization type: full
L1d cache: 32K
L1i cache: 32K
L2 cache: 256K
L3 cache: 46080K
NUMA node0 CPU(s): 0-15,32-47
NUMA node1 CPU(s): 16-31,48-63
Flags: fpu vme de pse tsc msr pae mce cx8 apic sep mtrr pge mca cmov pat pse36 clflush mmx fxsr sse sse2 ht syscall nx pdpe1gb rdtscp lm constant_tsc rep_good nopl xtopology nonstop_tsc aperfmperf eagerfpu pni pclmulqdq monitor est ssse3 fma cx16 pcid sse4_1 sse4_2 x2apic movbe popcnt tsc_deadline_timer aes xsave avx f16c rdrand hypervisor lahf_lm abm fsgsbase bmi1 hle avx2 smep bmi2 erms invpcid rtm xsaveopt ida
Parameter na.strings == <<NA>>
None of the 1 na.strings are numeric (such as '-9999').
Input contains no \n. Taking this to be a filename to open
File opened, filesize is 88.603947 GB.
Memory mapping ... ok
Detected eol as \n only (no \r afterwards), the UNIX and Mac standard.
Positioned on line 1 starting: <<allele prediction_uuid sample_>>
Detecting sep ...
sep=='\t' with 101 lines of 76 fields using quote rule 0
Detected 76 columns on line 1. This line is either column names or first data row (first 30 chars): <<allele prediction_uuid sample_>>
All the fields on line 1 are character fields. Treating as the column names.
Number of sampling jump points = 101 because 95137762779 bytes from row 1 to eof / (2 * 24414 jump0size) == 1948426
Type codes (jump 000) : 5555542444111145424441111444111111111111111111111111111111111111111111111111 Quote rule 0
Type codes (jump 009) : 5555542444114445424441144444111111111111111111111111111111111111111111111111 Quote rule 0
Type codes (jump 042) : 5555542444444445424444444444111111111111111111111111111111111111111111111111 Quote rule 0
Type codes (jump 048) : 5555544444444445444444444444225225522555545111111111111111111111111111111111 Quote rule 0
Type codes (jump 083) : 5555544444444445444444444444225225522555545254452454411154454452454411154455 Quote rule 0
Type codes (jump 085) : 5555544444444445444444444444225225522555545254452454454454454452454454454455 Quote rule 0
Type codes (jump 100) : 5555544444444445444444444444225225522555545254452454454454454452454454454455 Quote rule 0
=====
Sampled 10028 rows (handled \n inside quoted fields) at 101 jump points including middle and very end
Bytes from first data row on line 2 to the end of last row: 95137762779
Line length: mean=465.06 sd=250.27 min=198 max=929
Estimated nrow: 95137762779 / 465.06 = 204571280
Initial alloc = 409142560 rows (204571280 + 100%) using bytes/max(mean-2*sd,min) clamped between [1.1*estn, 2.0*estn]
=====
Type codes (colClasses) : 5555544444444445444444444444225225522555545254452454454454454452454454454455
Type codes (drop|select) : 5555544444444445444444444444225225522555545254452454454454454452454454454455
Allocating 76 column slots (76 - 0 dropped)
Reading 90752 chunks of 1.000MB (2254 rows) using 64 threads
Si ayuda, aquí hay resultados en una base de datos muy larga: 419,124,196 x 42 (~ 2 ^ 34) con una fila de encabezado y colClasses pasadas.
> library(data.table)
data.table 1.10.5 IN DEVELOPMENT built 2017-09-27 17:12:56 UTC; travis
The fastest way to learn (by data.table authors): https://www.datacamp.com/courses/data-analysis-the-data-table-way
Documentation: ?data.table, example(data.table) and browseVignettes("data.table")
Release notes, videos and slides: http://r-datatable.com
> CC <- c(rep('integer', 2), rep('character', 3),
+ rep('numeric', 2), rep('integer', 3),
+ rep('character', 2), 'integer', 'character', 'integer',
+ rep('character', 4), rep('numeric', 11), 'character',
+ 'numeric', 'character', rep('numeric', 2),
+ rep('integer', 3), rep('numeric', 2), 'integer',
+ 'numeric')
> P <- fread('XXXX.csv', colClasses = CC, header = TRUE, verbose = TRUE)
Input contains no \n. Taking this to be a filename to open
[01] Check arguments
Using 40 threads (omp_get_max_threads()=40, nth=40)
NAstrings = [<<NA>>]
None of the NAstrings look like numbers.
show progress = 1
0/1 column will be read as boolean
[02] Opening the file
Opening file XXXXcsv
File opened, size = 51.71GB (55521868868 bytes).
Memory mapping ... ok
[03] Detect and skip BOM
[04] Arrange mmap to be \0 terminated
\r-only line endings are not allowed because \n is found in the data
[05] Skipping initial rows if needed
Positioned on line 1 starting: <<X,X,X,X>>
[06] Detect separator, quoting rule, and ncolumns
Detecting sep ...
sep=',' with 100 lines of 42 fields using quote rule 0
Detected 42 columns on line 1. This line is either column names or first data row. Line starts as: <<X,X,X,X>>
Quote rule picked = 0
fill=false and the most number of columns found is 42
[07] Detect column types, good nrow estimate and whether first row is column names
'header' changed by user from 'auto' to true
Number of sampling jump points = 101 because (55521868866 bytes from row 1 to eof) / (2 * 13006 jump0size) == 2134471
Type codes (jump 000) : 5161010775551055105101111111111111110110771117717 Quote rule 0
Type codes (jump 022) : 5561010775551055105101111111111111110110771117717 Quote rule 0
Type codes (jump 030) : 5561010775551055105101010107517171151110110771117717 Quote rule 0
Type codes (jump 037) : 5561010775551055105101010107517771171110110771117717 Quote rule 0
Type codes (jump 073) : 5561010775551055105101010107517771177110110771117717 Quote rule 0
Type codes (jump 093) : 5561010775551055105101010107717771177110110771117717 Quote rule 0
Type codes (jump 100) : 5561010775551055105101010107717771177110110771117717 Quote rule 0
=====
Sampled 10049 rows (handled \n inside quoted fields) at 101 jump points
Bytes from first data row on line 1 to the end of last row: 55521868866
Line length: mean=132.68 sd=6.00 min=118 max=425
Estimated number of rows: 55521868866 / 132.68 = 418453923
Initial alloc = 460299315 rows (418453923 + 9%) using bytes/max(mean-2*sd,min) clamped between [1.1*estn, 2.0*estn]
=====
[08] Assign column names
[09] Apply user overrides on column types
After 11 type and 0 drop user overrides : 551010107755510105105101010107777777777710710775557757
[10] Allocate memory for the datatable
Allocating 42 column slots (42 - 0 dropped) with 460299315 rows
[11] Read the data
jumps=[0..52960), chunk_size=1048373, total_size=55521868441
Read 98%. ETA 00:00
[12] Finalizing the datatable
Read 419124195 rows x 42 columns from 51.71GB (55521868868 bytes) file in 13:42.935 wall clock time
Thread buffers were grown 0 times (if all 40 threads each grew once, this figure would be 40)
Final type counts
0 : drop
0 : bool8
0 : bool8
0 : bool8
0 : bool8
11 : int32
0 : int64
19 : float64
0 : float64
0 : float64
12 : string
=============================
0.000s ( 0%) Memory map 51.709GB file
0.016s ( 0%) sep=',' ncol=42 and header detection
0.016s ( 0%) Column type detection using 10049 sample rows
188.153s ( 23%) Allocation of 419124195 rows x 42 cols (125.177GB)
634.751s ( 77%) Reading 52960 chunks of 1.000MB (7901 rows) using 40 threads
= 0.121s ( 0%) Finding first non-embedded \n after each jump
+ 17.036s ( 2%) Parse to row-major thread buffers
+ 616.184s ( 75%) Transpose
+ 1.410s ( 0%) Waiting
0.000s ( 0%) Rereading 0 columns due to out-of-sample type exceptions
822.935s Total
> memory.size()
[1] 134270.3
> rm(P)
> gc()
used (Mb) gc trigger (Mb) max used (Mb)
Ncells 585532 31.3 5489235 293.2 6461124 345.1
Vcells 1508139082 11506.2 20046000758 152938.9 25028331901 190951.1
> memory.size()
[1] 87.56
> P <- fread('XXXX.csv', colClasses = CC, header = TRUE, verbose = TRUE)
Input contains no \n. Taking this to be a filename to open
[01] Check arguments
Using 40 threads (omp_get_max_threads()=40, nth=40)
NAstrings = [<<NA>>]
None of the NAstrings look like numbers.
show progress = 1
0/1 column will be read as boolean
[02] Opening the file
Opening file XXXX.csv
File opened, size = 51.71GB (55521868868 bytes).
Memory mapping ... ok
[03] Detect and skip BOM
[04] Arrange mmap to be \0 terminated
\r-only line endings are not allowed because \n is found in the data
[05] Skipping initial rows if needed
Positioned on line 1 starting: <<X,X,X,X>>
[06] Detect separator, quoting rule, and ncolumns
Detecting sep ...
sep=',' with 100 lines of 42 fields using quote rule 0
Detected 42 columns on line 1. This line is either column names or first data row. Line starts as: <<X,X,X,X>>
Quote rule picked = 0
fill=false and the most number of columns found is 42
[07] Detect column types, good nrow estimate and whether first row is column names
'header' changed by user from 'auto' to true
Number of sampling jump points = 101 because (55521868866 bytes from row 1 to eof) / (2 * 13006 jump0size) == 2134471
Type codes (jump 000) : 5161010775551055105101111111111111110110771117717 Quote rule 0
Type codes (jump 022) : 5561010775551055105101111111111111110110771117717 Quote rule 0
Type codes (jump 030) : 5561010775551055105101010107517171151110110771117717 Quote rule 0
Type codes (jump 037) : 5561010775551055105101010107517771171110110771117717 Quote rule 0
Type codes (jump 073) : 5561010775551055105101010107517771177110110771117717 Quote rule 0
Type codes (jump 093) : 5561010775551055105101010107717771177110110771117717 Quote rule 0
Type codes (jump 100) : 5561010775551055105101010107717771177110110771117717 Quote rule 0
=====
Sampled 10049 rows (handled \n inside quoted fields) at 101 jump points
Bytes from first data row on line 1 to the end of last row: 55521868866
Line length: mean=132.68 sd=6.00 min=118 max=425
Estimated number of rows: 55521868866 / 132.68 = 418453923
Initial alloc = 460299315 rows (418453923 + 9%) using bytes/max(mean-2*sd,min) clamped between [1.1*estn, 2.0*estn]
=====
[08] Assign column names
[09] Apply user overrides on column types
After 11 type and 0 drop user overrides : 551010107755510105105101010107777777777710710775557757
[10] Allocate memory for the datatable
Allocating 42 column slots (42 - 0 dropped) with 460299315 rows
[11] Read the data
jumps=[0..52960), chunk_size=1048373, total_size=55521868441
Read 98%. ETA 00:00
[12] Finalizing the datatable
Read 419124195 rows x 42 columns from 51.71GB (55521868868 bytes) file in 05:04.910 wall clock time
Thread buffers were grown 0 times (if all 40 threads each grew once, this figure would be 40)
Final type counts
0 : drop
0 : bool8
0 : bool8
0 : bool8
0 : bool8
11 : int32
0 : int64
19 : float64
0 : float64
0 : float64
12 : string
=============================
0.000s ( 0%) Memory map 51.709GB file
0.031s ( 0%) sep=',' ncol=42 and header detection
0.000s ( 0%) Column type detection using 10049 sample rows
28.437s ( 9%) Allocation of 419124195 rows x 42 cols (125.177GB)
276.442s ( 91%) Reading 52960 chunks of 1.000MB (7901 rows) using 40 threads
= 0.017s ( 0%) Finding first non-embedded \n after each jump
+ 12.941s ( 4%) Parse to row-major thread buffers
+ 262.989s ( 86%) Transpose
+ 0.495s ( 0%) Waiting
0.000s ( 0%) Rereading 0 columns due to out-of-sample type exceptions
304.910s Total
> memory.size()
[1] 157049.7
> sessionInfo()
R version 3.4.2 beta (2017-09-17 r73296)
Platform: x86_64-w64-mingw32/x64 (64-bit)
Running under: Windows Server >= 2012 x64 (build 9200)
Matrix products: default
locale:
[1] LC_COLLATE=English_United States.1252 LC_CTYPE=English_United States.1252
[3] LC_MONETARY=English_United States.1252 LC_NUMERIC=C
[5] LC_TIME=English_United States.1252
attached base packages:
[1] stats graphics grDevices utils datasets methods base
other attached packages:
[1] data.table_1.10.5
loaded via a namespace (and not attached):
[1] compiler_3.4.2 tools_3.4.2
Un par de notas. Yo habría sugerido poner [09] antes de [07] en el sentido de que si se pasan colClasses no hay razón para verificar. Además, Windows mostró alrededor de 160 GB en uso después de cada ejecución. memory.size () probablemente hace algo de limpieza. Con 532 GB de RAM en este servidor, el almacenamiento en caché de la memoria puede tener que ver con el aumento de velocidad en la segunda ejecución. Espero que ayude.
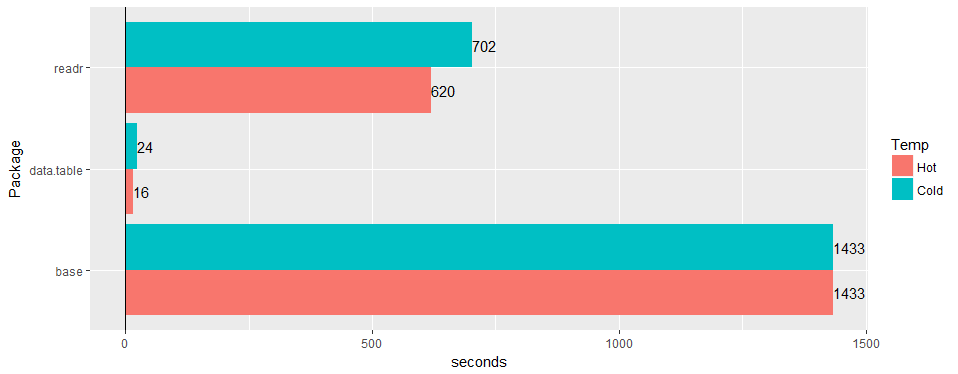
Pruebe en una amplia tabla del "mundo real" (datos del hospital): 30 millones de filas × 125 columnas v readr '1.2.0' y read.csv 3.4.3.
¿Alguna posibilidad de confirmar que el problema sigue siendo válido en 1.11.4? o código para producir datos de ejemplo.
Este está todo resuelto hasta donde yo sé. @geponce , actualice si no.
TODO1 anterior ahora archivado como # 3024
TODO2 anterior ahora archivado como # 3025
Comentario más útil
Pruebe en una amplia tabla del "mundo real" (datos del hospital): 30 millones de filas × 125 columnas v readr '1.2.0' y
read.csv3.4.3.