Xgboost: Vorhersage unterschiedlich in v1.2.1 und Master-Zweig
Ich verwende die freigegebene Version 1.2.1 für die Entwicklung aufgrund der einfachen Installation in Python und schreibe auch einen einfachen C-Code zur Vorhersage mit der c_api. Bei einer Verknüpfung mit v1.2.1 libxgboost.so ist die Differenz der Vorhersagen zwischen Python und c genau null. Bei einer Verknüpfung mit libxgboost.so aus dem Master-Zweig (Commit f3a425398 am 5. November 2020) gibt es jedoch einen Unterschied.
Ich möchte den C-Code in einem realen System mit dem Master-Zweig bereitstellen, da ich eine statische Bibliothek erstellen möchte, und jetzt behindert mich der Unterschied der Vorhersagen zwischen v1.2.1 und dem Master-Zweig.
Danke.
Alle 35 Kommentare
Können Sie ein Beispielprogramm posten, damit wir den Fehler reproduzieren können?
Sehen Sie auch unterschiedliche Vorhersagen aus der Masterversion des Python-Pakets und der C-API?
Es gibt einige Optimierungen am CPU-Prädiktor, die durch unterschiedliche Gleitkommafehler zu unterschiedlichen Ergebnissen führen können. Aber ja, hast du ein reproduzierbares Beispiel?
Sehen Sie auch unterschiedliche Vorhersagen aus der Masterversion des Python-Pakets und der C-API?
Ich vergleiche die v1.2.1 des Python-Pakets und den Master-Zweig der C-API.
@7starsea Können Sie auch die Ausgaben der Python- und C-API vergleichen, beide aus dem Master-Zweig? Das Problem könnte an der Verwendung von C-API-Funktionen in Ihrer Anwendung liegen.
@7starsea Können Sie auch die Ausgaben der Python- und C-API vergleichen, beide aus dem Master-Zweig? Das Problem könnte an der Verwendung von C-API-Funktionen in Ihrer Anwendung liegen.
Ich habe die Python- und C-API verglichen, beide von v1.2.1 und die Vorhersagen sind genau gleich.
@7starsea Verstanden . Wenn Sie sowohl Python- als auch C-Programme veröffentlichen, die von demselben Modell vorhersagen, können wir das Problem weiter beheben.
@hcho3 hier ist der
@7starsea Ich habe gerade dein Beispiel ausprobiert und folgende Ausgabe erhalten:
difference: [0. 0. 0. 0.] 0.0 0.0
Ich habe das neueste Commit von XGBoost verwendet (debeae2509d90ec1d3402a3a185fba7a25113ff1).
@7starsea Ich habe gerade dein Beispiel ausprobiert und folgende Ausgabe erhalten:
difference: [0. 0. 0. 0.] 0.0 0.0Ich habe das neueste Commit von XGBoost ( debeae2 ) verwendet.
Interessant, ist Ihre Python-Version v1.2.1?
Ich habe immer noch einen Unterschied zwischen Python-Version 1.2.1 und c-API, die mit XGBoost ( debeae2 ) verknüpft sind.
@7starsea Nein, ich habe XGBoost aus der neuesten Quelle kompiliert (commit debeae2509d90ec1d3402a3a185fba7a25113ff1), also ist es neuer als v1.2.1. Mein XGBoost Python-Paket gibt 1.3.0-SNAPSHOT für das Feld xgboost.__version__ aus.
@hcho3 Ich frage mich, ob XGBoost eine konsistente Vorhersage zwischen verschiedenen Versionen (zumindest aufeinanderfolgende Versionen) beibehalten sollte?
Erwartet auch die Veröffentlichung von v1.3.0.
(Anscheinend muss ich das Modell jetzt mit dem Master-Zweig trainieren)
Vielen Dank für Ihre Zeit.
@7starsea Wenn Sie ein gespeichertes Modell aus einer früheren Version laden, sollten Sie in der Lage sein, die konsistente Vorhersage zu erhalten.
Ich konnte das Problem mit deinem Skript nicht reproduzieren. Können Sie versuchen, ein Docker-Image oder ein VM-Image zu erstellen und es mit mir zu teilen?
@7starsea FYI, ich habe auch versucht, XGBoost 1.2.1 aus der Quelle zu erstellen, wie folgt:
git clone --recursive https://github.com/dmlc/xgboost -b release_1.2.0 xgb_source
cd xgb_source
mkdir build
cd build
cmake ..
make
cd ../python-package
python setup.py install
Die Ergebnisse zeigen wieder difference: [0. 0. 0. 0.] 0.0 0.0
Um den Unterschied zu sehen, benötigen Sie zwei Versionen von XGBoost, v1.2.1 für Python und
dtest = xgb.DMatrix(rx, missing=0.0)
y1 = m2.predict(dtest) # # internally using libxgboost.so v1.2.1
und einer für cpp
m1 = XgbShannonPredictor(fname)
y2 = m1.predict(rx2) # # internally using libxgboost.so from the master branch (debeae2)
Ich werde versuchen, ein Docker-Image zu erstellen (das für mich neu ist).
Lass mich mal sehen.
Ich werde versuchen, ein Docker-Image zu erstellen (das für mich neu ist).
Nicht nötig.
Tatsächlich konnte ich das Problem reproduzieren. Es stellt sich heraus, dass die Entwicklerversion von XGBoost eine andere Vorhersage liefert als XGBoost 1.2.0. Und das Problem ist einfach zu reproduzieren; keine Notwendigkeit, die C-API zu verwenden.
Reproduzierbares Beispiel (EDIT: Setzen des Zufalls-Seeds):
import numpy as np
import xgboost as xgb
rng = np.random.default_rng(seed=2020)
rx = rng.standard_normal(size=(100, 127 + 7 + 1))
rx = rx.astype(np.float32, order='C')
m2 = xgb.Booster({'nthread': '4'}) # init model
m2.load_model('xgb.model.bin') # load data
dtest = xgb.DMatrix(rx, missing=0.0)
y1 = m2.predict(dtest)
print(xgb.__version__)
print(y1)
Ausgabe aus 1.2.0:
1.2.0
[ 0.00698659 -0.00211251 0.00180039 -0.00016004 0.00526169 0.00801963
0.00016755 0.00226218 0.00276762 0.00408182 0.00303206 0.00291929
0.01101092 0.0068329 0.00145864 0.00326979 0.00572816 0.01019934
0.00074345 0.00784767 0.00173795 -0.00219297 0.0060181 0.00606489
0.00447372 0.00103396 0.00932363 0.00230178 0.00389203 0.00151157
0.0034163 0.00821933 0.006686 0.00630778 0.00331488 0.00775066
0.00443819 0.01030204 0.00924486 0.00645933 0.00777653 0.00231206
0.00457835 0.00390425 0.00947028 0.00410065 0.00220913 0.00292507
0.00637993 0.00796807 0.00140873 0.00887537 0.00496858 0.01049942
0.00908098 0.00332722 0.00799242 0.00228494 0.00463879 0.00213429
0.00729388 0.01049232 0.00790522 0.01269361 -0.00425893 0.00256333
0.00859573 0.00472835 0.00077197 0.00191873 0.01546788 0.0014475
0.00888193 0.00648022 0.00115797 0.00351191 0.00580138 0.00614035
0.00632426 0.00408354 0.00346044 -0.00034332 0.00599384 0.00302595
0.00657633 0.01086903 0.00625807 0.00096565 0.00061804 0.00038511
0.00523874 0.00633043 0.00379965 0.00302553 -0.00123322 0.00153473
0.00725579 0.00836438 0.01295918 0.00737873]
Notiz. Das Ausführen des Skripts mit XGBoost 1.0.0 und 1.1.0 führt zur identischen Ausgabe wie 1.2.0.
Ausgabe der Entwicklerversion (c5645180a6afb9d3d771165e681985fe3522adf6)
1.3.0-SNAPSHOT
[ 0.00698666 -0.00211278 0.00180034 -0.00016027 0.00526194 0.00801962
0.00016758 0.00226211 0.00276773 0.00408198 0.00303223 0.00291933
0.01101091 0.00683288 0.00145871 0.00326988 0.00572827 0.01019943
0.00074329 0.00784767 0.00173803 -0.00219286 0.00601804 0.00606472
0.00447388 0.00103391 0.00932358 0.00230171 0.003892 0.00151177
0.00341637 0.00821943 0.00668607 0.00630774 0.00331502 0.00775074
0.0044381 0.01030211 0.00924495 0.00645958 0.00777672 0.00231205
0.00457842 0.00390424 0.00947046 0.00410091 0.0022092 0.00292498
0.00638005 0.00796804 0.00140869 0.00887531 0.00496863 0.01049942
0.00908096 0.00332738 0.00799218 0.00228496 0.004639 0.00213413
0.00729368 0.01049243 0.00790528 0.01269368 -0.00425872 0.00256319
0.00859569 0.00472848 0.0007721 0.00191874 0.01546813 0.00144742
0.00888212 0.00648021 0.00115819 0.00351191 0.00580168 0.00614044
0.00632418 0.0040833 0.00346038 -0.00034315 0.00599405 0.00302578
0.0065765 0.01086897 0.00625799 0.00096572 0.00061766 0.00038494
0.00523901 0.00633054 0.00379964 0.00302567 -0.00123339 0.00153471
0.00725584 0.00836433 0.01295913 0.00737863]
@hcho3 Willst du das untersuchen? Bei Bedarf kann ich beim Halbieren helfen.
Warte eine Sekunde, ich habe vergessen, den zufälligen Seed in meinem Repro zu setzen. Wie dumm von mir.
Ich habe mein Repro mit dem festen zufälligen Seed aktualisiert. Der Fehler besteht weiterhin. Ich habe versucht, das aktualisierte Repro mit XGBoost 1.0.0 und 1.1.0 auszuführen, und die Vorhersagen stimmen mit den Vorhersagen von XGBoost 1.2.0 überein.
Zusamenfassend:
Prediction from 1.0.0
== Prediction from 1.1.0
== Prediction from 1.2.0
!= Prediction from latest master
@trivialfis Ja, Ihre Hilfe wird geschätzt.
Markieren Sie dies als blockierend.
Habe es.
Zurückverfolgt zu a4ce0eae43f7e0e2f91566ef2360830b86b9fdcf . @ShvetsKS Möchten Sie einen Blick darauf werfen?
Zurückverfolgt zu a4ce0ea . @ShvetsKS Möchten Sie einen Blick darauf werfen?
Sicher. Könnten Sie beim Trainieren des Modells vom Python-Reproduzierer helfen:
m2.load_model('xgb.model.bin') # load data
Welche XGBoost-Version wird für das Training verwendet und welche Parameter sollten genau bereitgestellt werden?
@ShvetsKS Sie können die Modelldatei xgb.model.bin von https://github.com/7starsea/xgboost-testing erhalten. Das Modell wurde mit 1.0.0 trainiert.
@ShvetsKS Sie können die Modelldatei
xgb.model.binvon https://github.com/7starsea/xgboost-testing erhalten. Das Modell wurde mit 1.0.0 trainiert.
das Modell wurde tatsächlich mit 1.2.1 und Parametern trainiert
param = {'max_depth': 8, 'eta': 0.1, 'min_child_weight': 2, 'gamma': 1e-8, 'subsample': 0.6, 'nthread': 4}
Danke.
Anscheinend ist der kleine Unterschied auf die geänderte Reihenfolge der Gleitkommaoperationen zurückzuführen.
_Genauer Grund:_
Vor a4ce0ea inkrementieren wir alle Baumantworten in die lokale Variable psum (anfänglich gleich Null) und inkrementieren dann den entsprechenden Wert von out_preds .
In a4ce0ea erhöhen wir out_preds Werte direkt um jede Baumantwort.
Fix ist vorbereitet: https://github.com/dmlc/xgboost/pull/6384
@7starsea danke, dass du den Unterschied gefunden hast.
@hcho3 , @trivialfis Betrachten wir einen solchen Unterschied in Zukunft als kritisch? Scheint eine erhebliche Einschränkung zu sein, die es nicht erlaubt, die Reihenfolge von Gleitkommaoperationen für Rückschlüsse zu ändern. Aber für die Trainingsphase gibt es keine solche Anforderung, wie ich mich erinnere.
Halten wir einen solchen Unterschied in Zukunft für kritisch?
Normalerweise nein. Lassen Sie mich einen Blick auf Ihre Änderungen werfen. ;-)
@ShvetsKS Ich habe gerade nachgesehen und der Unterschied ist jetzt genau null. Vielen Dank, dass Sie den Vorhersageunterschied behoben haben.
@ShvetsKS
Halten wir einen solchen Unterschied in Zukunft für kritisch? Scheint eine erhebliche Einschränkung zu sein, die es nicht erlaubt, die Reihenfolge der Gleitkommaoperationen für Rückschlüsse zu ändern
Tatsächlich stimmen wir ( @RAMitchell , @trivialfis und ich) Ihnen hier zu. Die Vorgabe einer exakten Reproduzierbarkeit der Vorhersage wird unsere Fähigkeit, Änderungen vorzunehmen, stark beeinträchtigen. Gleitkommaarithmetik ist bekanntlich nicht assoziativ, daher wird die Summe einer Zahlenliste je nach Additionsreihenfolge geringfügig abweichen.
Ich habe ein Experiment durchgeführt, um zu quantifizieren, wie stark sich die Vorhersage zwischen XGBoost 1.2.0 und dem neuesten master Zweig ändert: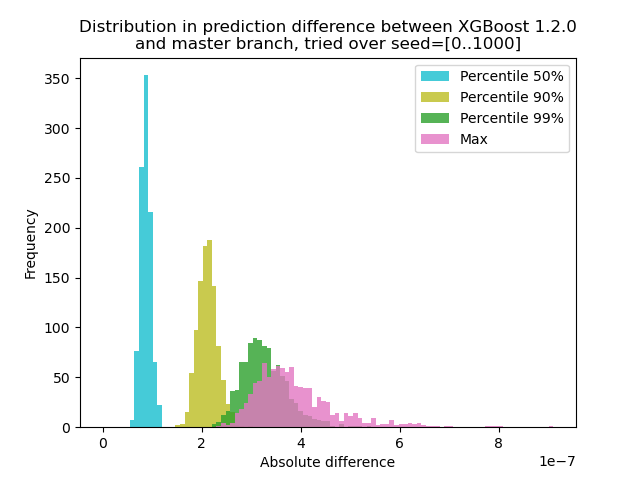
Ich habe Daten mit 1000 verschiedenen Zufalls-Seeds generiert und dann eine Vorhersage mit den 1000 Matrizen durchgeführt, wobei sowohl die Versionen 1.2.0 als auch der Master verwendet wurden. Die Vorhersageänderung ändert sich zwischen den Startpunkten geringfügig, aber
Skript zum Experiment
**test.py**: Generiere 1000 Matrizen mit verschiedenen zufälligen Startwerten und führe Vorhersagen für sie aus.
import numpy as np
import xgboost as xgb
import argparse
def main(args):
m2 = xgb.Booster({'nthread': '4'}) # init model
m2.load_model('xgb.model.bin') # load data
out = {}
for seed in range(1000):
rng = np.random.default_rng(seed=seed)
rx = rng.standard_normal(size=(100, 127 + 7 + 1))
rx = rx.astype(np.float32, order='C')
dtest = xgb.DMatrix(rx, missing=0.0)
out[str(seed)] = m2.predict(dtest)
np.savez(args.out_pred, **out)
if __name__ == '__main__':
parser = argparse.ArgumentParser()
parser.add_argument('--out-pred', type=str, required=True)
args = parser.parse_args()
main(args)
import numpy as np
import matplotlib.pyplot as plt
xgb120 = np.load('xgb120.npz')
xgblatest = np.load('xgblatest.npz')
percentile_pts = [50, 90, 99]
colors = ['tab:cyan', 'tab:olive', 'tab:green', 'tab:pink']
percentile = {}
for x in percentile_pts:
percentile[x] = []
percentile['max'] = []
for seed in range(1000):
diff = np.abs(xgb120[str(seed)] - xgblatest[str(seed)])
t = np.percentile(diff, percentile_pts)
for x, y in zip(percentile_pts, t):
percentile[x].append(y)
percentile['max'].append(np.max(diff))
bins = np.linspace(0, np.max(percentile['max']), 100)
idx = 0
for x in percentile_pts:
plt.hist(percentile[x], label=f'Percentile {x}%', bins=bins, alpha=0.8, color=colors[idx])
idx += 1
plt.hist(percentile['max'], label='Max', bins=bins, alpha=0.8, color=colors[idx])
plt.legend(loc='best')
plt.title('Distribution in prediction difference between XGBoost 1.2.0\nand master branch, tried over seed=[0..1000]')
plt.xlabel('Absolute difference')
plt.ylabel('Frequency')
plt.savefig('foobar.png', dpi=100)
Da hier das Problem darin besteht, dass + mit float keine Gruppe bildet, können wir mit entfernter Summe testen: Vorhersage auf einem einzelnen Baum. Das Ergebnis sollte genau das gleiche sein.
@trivialfis Tatsächlich, wenn ich ntree_limit=1 Argument zu m2.predict() hinzugefügt habe, verschwindet die Differenz auf 0.
Toll! Als nächstes ist also die Frage, wie wir es dokumentieren oder ob wir es dokumentieren sollen.
Lass mich darauf schlafen. Für den Moment genügt es zu sagen, dass dieses Problem nicht wirklich ein Fehler ist.
Hilfreichster Kommentar
@ShvetsKS
Tatsächlich stimmen wir ( @RAMitchell , @trivialfis und ich) Ihnen hier zu. Die Vorgabe einer exakten Reproduzierbarkeit der Vorhersage wird unsere Fähigkeit, Änderungen vorzunehmen, stark beeinträchtigen. Gleitkommaarithmetik ist bekanntlich nicht assoziativ, daher wird die Summe einer Zahlenliste je nach Additionsreihenfolge geringfügig abweichen.
Ich habe ein Experiment durchgeführt, um zu quantifizieren, wie stark sich die Vorhersage zwischen XGBoost 1.2.0 und dem neuesten

masterZweig ändert:Ich habe Daten mit 1000 verschiedenen Zufalls-Seeds generiert und dann eine Vorhersage mit den 1000 Matrizen durchgeführt, wobei sowohl die Versionen 1.2.0 als auch der Master verwendet wurden. Die Vorhersageänderung ändert sich zwischen den Startpunkten geringfügig, aber
Skript zum Experiment
**test.py**: Generiere 1000 Matrizen mit verschiedenen zufälligen Startwerten und führe Vorhersagen für sie aus.
Befehl: `python test.py --out-pred [out.npz]`. Stellen Sie sicher, dass Ihre Python-Umgebung die richtige Version von XGBoost hat. Nehmen wir an, `xgb120.npz` speichert das Ergebnis für XGBoost 1.2.0 und `xgblatest.npz` speichert das Ergebnis für den neuesten Master. **compare.py**: Erstellen Sie ein Histogrammdiagramm für den Vorhersageunterschied