Xgboost: prédiction différente dans la v1.2.1 et la branche master
J'utilise la version 1.2.1 publiée pour le développement en raison de son installation facile en python et j'écris également un code c simple pour la prédiction à l'aide de c_api. S'il est lié à v1.2.1 libxgboost.so, la différence des prédictions entre python et c est exactement zéro. Cependant, s'il est lié à libxgboost.so depuis la branche master (commit f3a425398 le 5 novembre 2020), il y a une différence.
J'aimerais déployer le code c dans un système réel en utilisant la branche master car je voudrais construire une bibliothèque statique, et maintenant la différence des prédictions entre la v1.2.1 et la branche master me gêne.
Merci.
Tous les 35 commentaires
Pouvez-vous poster un exemple de programme afin que nous puissions reproduire le bogue ?
De plus, voyez-vous des prédictions différentes de la version principale du package Python et de l'API C ?
Certaines optimisations sont effectuées sur le prédicteur du processeur, elles peuvent générer un résultat différent par une erreur en virgule flottante différente. Mais oui, avez-vous un exemple reproductible ?
De plus, voyez-vous des prédictions différentes de la version principale du package Python et de l'API C ?
Je compare la version 1.2.1 du package python et la branche principale de l'API C.
@7starsea Pouvez-vous également comparer les sorties des API Python et C, toutes deux de la branche master ? Le problème pourrait être la façon dont les fonctions de l'API C sont utilisées dans votre application.
@7starsea Pouvez-vous également comparer les sorties des API Python et C, toutes deux de la branche master ? Le problème pourrait être la façon dont les fonctions de l'API C sont utilisées dans votre application.
J'ai comparé python et c api, tous deux de la v1.2.1 et les prédictions sont exactement les mêmes.
@7starsea Je l'ai compris . Si vous publiez à la fois des programmes Python et C qui prédisent à partir du même modèle, nous serions en mesure de résoudre davantage le problème.
@hcho3 voici le code de test
@7starsea Je viens d'essayer votre exemple et j'ai obtenu le résultat suivant :
difference: [0. 0. 0. 0.] 0.0 0.0
J'ai utilisé le dernier commit de XGBoost (debeae2509d90ec1d3402a3a185fba7a25113ff1).
@7starsea Je viens d'essayer votre exemple et j'ai obtenu le résultat suivant :
difference: [0. 0. 0. 0.] 0.0 0.0J'ai utilisé le dernier commit de XGBoost ( debeae2 ).
Intéressant, votre python est-il en version 1.2.1 ?
J'ai encore une différence entre python-version 1.2.1 et c api lié à XGBoost ( debeae2 ).
@7starsea Non, j'ai compilé XGBoost à partir de la dernière source (commit debeae2509d90ec1d3402a3a185fba7a25113ff1), il est donc plus récent que la v1.2.1. Mon package Python XGBoost imprime 1.3.0-SNAPSHOT pour le champ xgboost.__version__ .
@hcho3 Je me demande si XGBoost doit conserver une prédiction cohérente entre différentes versions (au moins des versions consécutives) ?
Attendons également la sortie de la v1.3.0.
(il semble que j'ai besoin d'entraîner le modèle à l'aide de la branche master maintenant)
Merci pour votre temps.
@7starsea Si vous chargez un modèle enregistré à partir d'une version précédente, vous devriez pouvoir obtenir la prédiction cohérente.
Je n'ai pas pu reproduire le problème à l'aide de votre script. Pouvez-vous essayer de créer une image Docker ou une image de machine virtuelle et la partager avec moi ?
@7starsea Pour info , j'ai également essayé de créer XGBoost 1.2.1 à partir de la source, comme suit :
git clone --recursive https://github.com/dmlc/xgboost -b release_1.2.0 xgb_source
cd xgb_source
mkdir build
cd build
cmake ..
make
cd ../python-package
python setup.py install
Les résultats montrent à nouveau difference: [0. 0. 0. 0.] 0.0 0.0
Pour voir la différence, vous avez besoin de deux versions de XGBoost, v1.2.1 pour python et
dtest = xgb.DMatrix(rx, missing=0.0)
y1 = m2.predict(dtest) # # internally using libxgboost.so v1.2.1
et un pour cpp
m1 = XgbShannonPredictor(fname)
y2 = m1.predict(rx2) # # internally using libxgboost.so from the master branch (debeae2)
Je vais essayer de créer une image docker (ce qui est nouveau pour moi).
Laisse-moi regarder.
Je vais essayer de créer une image docker (ce qui est nouveau pour moi).
Pas nécessaire.
En fait, j'ai réussi à reproduire le problème. Il s'avère que la version de développement de XGBoost produit une prédiction différente de celle de XGBoost 1.2.0. Et le problème est simple à reproduire ; pas besoin d'utiliser l'API C.
Exemple reproductible (EDIT : réglage de la graine aléatoire) :
import numpy as np
import xgboost as xgb
rng = np.random.default_rng(seed=2020)
rx = rng.standard_normal(size=(100, 127 + 7 + 1))
rx = rx.astype(np.float32, order='C')
m2 = xgb.Booster({'nthread': '4'}) # init model
m2.load_model('xgb.model.bin') # load data
dtest = xgb.DMatrix(rx, missing=0.0)
y1 = m2.predict(dtest)
print(xgb.__version__)
print(y1)
Sortie de la 1.2.0 :
1.2.0
[ 0.00698659 -0.00211251 0.00180039 -0.00016004 0.00526169 0.00801963
0.00016755 0.00226218 0.00276762 0.00408182 0.00303206 0.00291929
0.01101092 0.0068329 0.00145864 0.00326979 0.00572816 0.01019934
0.00074345 0.00784767 0.00173795 -0.00219297 0.0060181 0.00606489
0.00447372 0.00103396 0.00932363 0.00230178 0.00389203 0.00151157
0.0034163 0.00821933 0.006686 0.00630778 0.00331488 0.00775066
0.00443819 0.01030204 0.00924486 0.00645933 0.00777653 0.00231206
0.00457835 0.00390425 0.00947028 0.00410065 0.00220913 0.00292507
0.00637993 0.00796807 0.00140873 0.00887537 0.00496858 0.01049942
0.00908098 0.00332722 0.00799242 0.00228494 0.00463879 0.00213429
0.00729388 0.01049232 0.00790522 0.01269361 -0.00425893 0.00256333
0.00859573 0.00472835 0.00077197 0.00191873 0.01546788 0.0014475
0.00888193 0.00648022 0.00115797 0.00351191 0.00580138 0.00614035
0.00632426 0.00408354 0.00346044 -0.00034332 0.00599384 0.00302595
0.00657633 0.01086903 0.00625807 0.00096565 0.00061804 0.00038511
0.00523874 0.00633043 0.00379965 0.00302553 -0.00123322 0.00153473
0.00725579 0.00836438 0.01295918 0.00737873]
Noter. l'exécution du script avec XGBoost 1.0.0 et 1.1.0 donne une sortie identique à 1.2.0.
Sortie de la version dev (c5645180a6afb9d3d771165e681985fe3522adf6)
1.3.0-SNAPSHOT
[ 0.00698666 -0.00211278 0.00180034 -0.00016027 0.00526194 0.00801962
0.00016758 0.00226211 0.00276773 0.00408198 0.00303223 0.00291933
0.01101091 0.00683288 0.00145871 0.00326988 0.00572827 0.01019943
0.00074329 0.00784767 0.00173803 -0.00219286 0.00601804 0.00606472
0.00447388 0.00103391 0.00932358 0.00230171 0.003892 0.00151177
0.00341637 0.00821943 0.00668607 0.00630774 0.00331502 0.00775074
0.0044381 0.01030211 0.00924495 0.00645958 0.00777672 0.00231205
0.00457842 0.00390424 0.00947046 0.00410091 0.0022092 0.00292498
0.00638005 0.00796804 0.00140869 0.00887531 0.00496863 0.01049942
0.00908096 0.00332738 0.00799218 0.00228496 0.004639 0.00213413
0.00729368 0.01049243 0.00790528 0.01269368 -0.00425872 0.00256319
0.00859569 0.00472848 0.0007721 0.00191874 0.01546813 0.00144742
0.00888212 0.00648021 0.00115819 0.00351191 0.00580168 0.00614044
0.00632418 0.0040833 0.00346038 -0.00034315 0.00599405 0.00302578
0.0065765 0.01086897 0.00625799 0.00096572 0.00061766 0.00038494
0.00523901 0.00633054 0.00379964 0.00302567 -0.00123339 0.00153471
0.00725584 0.00836433 0.01295913 0.00737863]
@hcho3 Voulez-vous vous
Attendez une seconde, j'ai oublié de définir la graine aléatoire dans ma reproduction. Que je suis bête.
J'ai mis à jour ma reproduction avec la graine aléatoire fixe. Le bug persiste toujours. J'ai essayé d'exécuter la reproduction mise à jour avec XGBoost 1.0.0 et 1.1.0, et les prédictions correspondent à celles de XGBoost 1.2.0.
En bref:
Prediction from 1.0.0
== Prediction from 1.1.0
== Prediction from 1.2.0
!= Prediction from latest master
@trivialfis Oui, votre aide sera appréciée.
Marquer ceci comme bloquant.
J'ai compris.
Tracé jusqu'à a4ce0eae43f7e0e2f91566ef2360830b86b9fdcf . @ShvetsKS Voulez-vous jeter un œil ?
Tracé jusqu'à a4ce0ea . @ShvetsKS Voulez-vous jeter un œil ?
Sûr. Pourriez-vous nous aider à entraîner le modèle à partir du reproducteur Python :
m2.load_model('xgb.model.bin') # load data
Quelle version de XGBoost est utilisée pour l'entraînement et quels paramètres doivent être fournis exactement ?
@ShvetsKS Vous pouvez obtenir le fichier modèle xgb.model.bin sur https://github.com/7starsea/xgboost-testing. Le modèle a été entraîné avec 1.0.0.
@ShvetsKS Vous pouvez obtenir le fichier modèle
xgb.model.binsur https://github.com/7starsea/xgboost-testing. Le modèle a été entraîné avec 1.0.0.
le modèle a en fait été entraîné avec 1.2.1 et les paramètres
param = {'max_depth': 8, 'eta': 0.1, 'min_child_weight': 2, 'gamma': 1e-8, 'subsample': 0.6, 'nthread': 4}
Merci.
Il semble que la petite différence soit due à la modification de la séquence d'opérations en virgule flottante.
_Raison exacte :_
Avant a4ce0ea, nous incrémentons toutes les réponses des arbres dans la variable locale psum (initialement égale à zéro) puis incrémentons la valeur appropriée à partir de out_preds .
Dans a4ce0ea, nous incrémentons les valeurs out_preds directement par chaque réponse d'arbre.
Le correctif est préparé : https://github.com/dmlc/xgboost/pull/6384
@7starsea merci d'avoir trouvé la différence, pourriez-vous vérifier le correctif ci-dessus ?
@hcho3 , @trivialfis Considérons -nous une telle différence comme critique à l'avenir ? Il semble que ce soit une restriction importante qui ne permet pas de modifier la séquence d'opérations en virgule flottante pour l'inférence. Mais pour le stade de la formation, il n'y a pas de telle exigence si je me souviens bien.
Considérons-nous une telle différence comme critique à l'avenir ?
Généralement non. Laissez-moi jeter un œil à vos modifications. ;-)
@ShvetsKS Je viens de vérifier et la différence est exactement nulle maintenant. Merci d'avoir corrigé la différence de prédiction.
@ShvetsKS
Considérons-nous une telle différence comme critique à l'avenir ? Il semble que ce soit une restriction importante qui ne permet pas de modifier la séquence d'opérations en virgule flottante pour l'inférence
En effet, nous ( @RAMitchell , @trivialfis , et moi) sommes d'accord avec vous ici. Obliger la reproductibilité exacte de la prédiction entravera gravement notre capacité à apporter des changements. L'arithmétique à virgule flottante est notoirement non associative, donc la somme d'une liste de nombres sera légèrement différente selon l'ordre d'addition.
J'ai mené une expérience pour quantifier à quel point la prédiction change entre XGBoost 1.2.0 et la dernière branche master :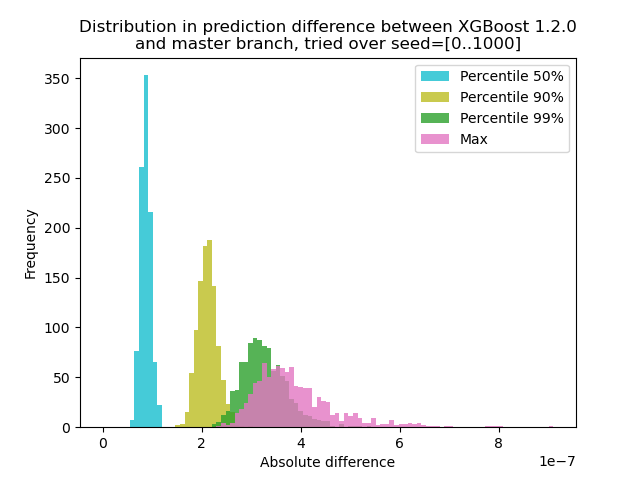
J'ai généré des données avec 1000 graines aléatoires différentes, puis j'ai exécuté une prédiction avec les 1000 matrices, en utilisant à la fois les versions 1.2.0 et master. Le changement de prédiction change légèrement entre les graines, mais la différence n'est jamais supérieure à 9,2e-7, il est donc très probable que le changement de prédiction ait été causé par l'arithmétique à virgule flottante et non par une erreur logique .
Script pour l'expérimentation
**test.py** : générez 1000 matrices avec différentes graines aléatoires et exécutez une prédiction pour elles.
import numpy as np
import xgboost as xgb
import argparse
def main(args):
m2 = xgb.Booster({'nthread': '4'}) # init model
m2.load_model('xgb.model.bin') # load data
out = {}
for seed in range(1000):
rng = np.random.default_rng(seed=seed)
rx = rng.standard_normal(size=(100, 127 + 7 + 1))
rx = rx.astype(np.float32, order='C')
dtest = xgb.DMatrix(rx, missing=0.0)
out[str(seed)] = m2.predict(dtest)
np.savez(args.out_pred, **out)
if __name__ == '__main__':
parser = argparse.ArgumentParser()
parser.add_argument('--out-pred', type=str, required=True)
args = parser.parse_args()
main(args)
import numpy as np
import matplotlib.pyplot as plt
xgb120 = np.load('xgb120.npz')
xgblatest = np.load('xgblatest.npz')
percentile_pts = [50, 90, 99]
colors = ['tab:cyan', 'tab:olive', 'tab:green', 'tab:pink']
percentile = {}
for x in percentile_pts:
percentile[x] = []
percentile['max'] = []
for seed in range(1000):
diff = np.abs(xgb120[str(seed)] - xgblatest[str(seed)])
t = np.percentile(diff, percentile_pts)
for x, y in zip(percentile_pts, t):
percentile[x].append(y)
percentile['max'].append(np.max(diff))
bins = np.linspace(0, np.max(percentile['max']), 100)
idx = 0
for x in percentile_pts:
plt.hist(percentile[x], label=f'Percentile {x}%', bins=bins, alpha=0.8, color=colors[idx])
idx += 1
plt.hist(percentile['max'], label='Max', bins=bins, alpha=0.8, color=colors[idx])
plt.legend(loc='best')
plt.title('Distribution in prediction difference between XGBoost 1.2.0\nand master branch, tried over seed=[0..1000]')
plt.xlabel('Absolute difference')
plt.ylabel('Frequency')
plt.savefig('foobar.png', dpi=100)
Comme ici le problème est que + avec float ne forme pas un groupe, nous pouvons tester avec sum supprimé : Prédire sur un seul arbre. Le résultat devrait être exactement le même.
@trivialfis En effet, lorsque j'ai ajouté l'argument ntree_limit=1 à m2.predict() , la différence s'évanouit à 0.
Super! La prochaine chose est de savoir comment le documenter ou si nous devons le documenter.
Laissez-moi dormir sur elle. Pour l'instant, il suffit de dire que ce problème n'est pas vraiment un bug.
Commentaire le plus utile
@ShvetsKS
En effet, nous ( @RAMitchell , @trivialfis , et moi) sommes d'accord avec vous ici. Obliger la reproductibilité exacte de la prédiction entravera gravement notre capacité à apporter des changements. L'arithmétique à virgule flottante est notoirement non associative, donc la somme d'une liste de nombres sera légèrement différente selon l'ordre d'addition.
J'ai mené une expérience pour quantifier à quel point la prédiction change entre XGBoost 1.2.0 et la dernière branche

master:J'ai généré des données avec 1000 graines aléatoires différentes, puis j'ai exécuté une prédiction avec les 1000 matrices, en utilisant à la fois les versions 1.2.0 et master. Le changement de prédiction change légèrement entre les graines, mais la différence n'est jamais supérieure à 9,2e-7, il est donc très probable que le changement de prédiction ait été causé par l'arithmétique à virgule flottante et non par une erreur logique .
Script pour l'expérimentation
**test.py** : générez 1000 matrices avec différentes graines aléatoires et exécutez une prédiction pour elles.
Commande : `python test.py --out-pred [out.npz]`. Assurez-vous que votre environnement Python dispose de la bonne version de XGBoost. Supposons que `xgb120.npz` stocke le résultat pour XGBoost 1.2.0 et que `xgblatest.npz` stocke le résultat pour le dernier maître. **compare.py** : créez un histogramme pour la différence de prédiction